-検索条件
-検索結果
検索 (著者・登録者: huang & pp)の結果276件中、1から50件目までを表示しています

EMDB-43139: 
SARS-CoV-2 Spike S2 bound to Fab 54043-5

EMDB-44635: 
Inactive mu opioid receptor bound to Nb6, naloxone and NAM

PDB-9bjk: 
Inactive mu opioid receptor bound to Nb6, naloxone and NAM

EMDB-42676: 
5-HT2AR bound to Lisuride in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)

EMDB-42999: 
5HT2AR-miniGq heterotrimer in complex with a novel agonist obtained from large scale docking

PDB-8uwl: 
5-HT2AR bound to Lisuride in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)

PDB-8v6u: 
5HT2AR-miniGq heterotrimer in complex with a novel agonist obtained from large scale docking
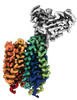
EMDB-43683: 
Cryo-EM structure of FLVCR2 in the inward-facing state with choline bound
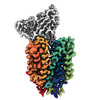
EMDB-43684: 
Cryo-EM structure of FLVCR2 in the outward-facing state with choline bound

PDB-8vzn: 
Cryo-EM structure of FLVCR2 in the inward-facing state with choline bound

PDB-8vzo: 
Cryo-EM structure of FLVCR2 in the outward-facing state with choline bound

EMDB-43991: 
Cryo-EM structure of apo state human Cav3.2

EMDB-43992: 
Cryo-EM structure of human Cav3.2 with TTA-A2

EMDB-43993: 
Cryo-EM structure of human Cav3.2 with TTA-P2

EMDB-43994: 
Cryo-EM structure of human Cav3.2 with ML218

EMDB-43995: 
Cryo-EM structure of human Cav3.2 with ACT-709478

PDB-9ayg: 
Cryo-EM structure of apo state human Cav3.2

PDB-9ayh: 
Cryo-EM structure of human Cav3.2 with TTA-A2

PDB-9ayj: 
Cryo-EM structure of human Cav3.2 with TTA-P2

PDB-9ayk: 
Cryo-EM structure of human Cav3.2 with ML218

PDB-9ayl: 
Cryo-EM structure of human Cav3.2 with ACT-709478

EMDB-17766: 
CryoEM structure of Nal1 protein, allele SPIKE, from Oryza sativa japonica group
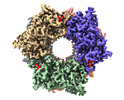
EMDB-17768: 
CryoEM structure of Nal1 protein, allele IR64, from Oryza sativa indica cultivar

PDB-8pn1: 
CryoEM structure of Nal1 protein, allele SPIKE, from Oryza sativa japonica group

PDB-8pn2: 
CryoEM structure of Nal1 protein, allele IR64, from Oryza sativa indica cultivar

EMDB-36138: 
CrtSPARTA Octamer bound with guide-target

EMDB-36059: 
Short ago complexed with TIR-APAZ

EMDB-36070: 
SPARTA monomer bound with guide-target, state 2

EMDB-36095: 
CrtSPARTA hetero-dimer bound with guide-target, state 1

EMDB-36114: 
SPARTA dimer bound with guide-target

EMDB-35452: 
Cryo-EM structure of ochratoxin A-detoxifying amidohydrolase ADH3

EMDB-35453: 
Cryo-EM structure of ochratoxin A-detoxifying amidohydrolase ADH3 in complex with Phe

EMDB-35454: 
Cryo-EM structure of ochratoxin A-detoxifying amidohydrolase ADH3 in complex with ochratoxin A

EMDB-36060: 
Cryo-EM structure of ochratoxin A-detoxifying amidohydrolase ADH3 mutant S88E in complex with ochratoxin A

EMDB-17756: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein

PDB-8pm2: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein
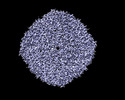
EMDB-28067: 
CryoEM reconstruction of tri-specific 298-52-80 Multabody (no symmetry applied)

EMDB-28068: 
CryoEM reconstruction of the tri-specific 298-52-80 Multabody (octahedral symmetry applied)

EMDB-14504: 
Three-dimensional structure of myosin binding protein C in rat cardiac muscle

EMDB-26767: 
The 2.19-angstrom CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - Complex minus stalk

EMDB-26801: 
The 1.67 Angstrom CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - catalytic dimer (Huc2S2L)

EMDB-26802: 
The CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - Full complex focused refinement of stalk

EMDB-27661: 
The 1.52 angstrom CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - catalytic dimer (Huc2S2L)

PDB-7utd: 
The 2.19-angstrom CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - Complex minus stalk

PDB-7uur: 
The 1.67 Angstrom CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - catalytic dimer (Huc2S2L)

PDB-7uus: 
The CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - Full complex focused refinement of stalk

PDB-8dqv: 
The 1.52 angstrom CryoEM structure of the [NiFe]-hydrogenase Huc from Mycobacterium smegmatis - catalytic dimer (Huc2S2L)

EMDB-27966: 
CryoEM structure of miniGq-coupled hM3Dq in complex with DCZ

EMDB-27967: 
CryoEM structure of miniGo-coupled hM4Di in complex with DCZ

EMDB-27968: 
CryoEM structure of miniGq-coupled hM3Dq in complex with CNO
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
