+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1634 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural analysis of substrate binding by the TatBC component of the twin-arginine protein transport system. | |||||||||
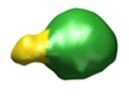
 マップデータ マップデータ | This is a 3D map of a TatBC complex with one sufI substrate bound. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | twin arginine / Tat /  protein transport / blue native PAGE / single particle electron microscopy protein transport / blue native PAGE / single particle electron microscopy | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 ネガティブ染色法 | |||||||||
 データ登録者 データ登録者 | Tarry MJ / Schaefer E / Chen S / Buchanan G / Greene NP / Lea SM / Palmer T / Saibil HR / Berks BC | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2009 ジャーナル: Proc Natl Acad Sci U S A / 年: 2009タイトル: Structural analysis of substrate binding by the TatBC component of the twin-arginine protein transport system. 著者: Michael J Tarry / Eva Schäfer / Shuyun Chen / Grant Buchanan / Nicholas P Greene / Susan M Lea / Tracy Palmer / Helen R Saibil / Ben C Berks /  要旨: The Tat system transports folded proteins across the bacterial cytoplasmic membrane and the thylakoid membrane of plant chloroplasts. In Escherichia coli substrate proteins initially bind to the ...The Tat system transports folded proteins across the bacterial cytoplasmic membrane and the thylakoid membrane of plant chloroplasts. In Escherichia coli substrate proteins initially bind to the integral membrane TatBC complex which then recruits the protein TatA to effect translocation. Overproduction of TatBC and the substrate protein SufI in the absence of TatA led to the accumulation of TatBC-SufI complexes that could be purified using an affinity tag on the substrate. Three-dimensional structures of the TatBC-SufI complexes and unliganded TatBC were obtained by single-particle electron microscopy and random conical tilt reconstruction. Comparison of the structures shows that substrate molecules bind on the periphery of the TatBC complex and that substrate binding causes a significant reduction in diameter of the TatBC part of the complex. Although the TatBC complex contains multiple copies of the signal peptide-binding TatC protomer, purified TatBC-SufI complexes contain only 1 or 2 SufI molecules. Where 2 substrates are present in the TatBC-SufI complex, they are bound at adjacent sites. These observations imply that only certain TatC protomers within the complex interact with substrate or that there is a negative cooperativity of substrate binding. Similar TatBC-substrate complexes can be generated by an alternative in vitro reconstitution method and using a different substrate protein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1634.map.gz emd_1634.map.gz | 40.4 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1634-v30.xml emd-1634-v30.xml emd-1634.xml emd-1634.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1634.gif EMD-1634.gif | 26.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1634 http://ftp.pdbj.org/pub/emdb/structures/EMD-1634 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1634 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1634 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1634.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1634.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a 3D map of a TatBC complex with one sufI substrate bound. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : tatBC structure with one sufI substrate bound
| 全体 | 名称: tatBC structure with one sufI substrate bound |
|---|---|
| 要素 |
|
-超分子 #1000: tatBC structure with one sufI substrate bound
| 超分子 | 名称: tatBC structure with one sufI substrate bound / タイプ: sample / ID: 1000 / Number unique components: 3 |
|---|
-分子 #1: twin arginine transporter
| 分子 | 名称: twin arginine transporter / タイプ: protein_or_peptide / ID: 1 / Name.synonym: tat / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 0.014 |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: negatively stained with 2% (wt/vol) uranyl acetate on glow discharged, continuous carbon-coated 300 mesh copper grids (Agar Scientific). |
| グリッド | 詳細: 300 |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 42000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダー: eucentric / 試料ホルダーモデル: OTHER / Tilt angle max: 50 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm 詳細: Micrographs were digitized on a Zeiss SCAI scanner at a pixel size of 7 micron, corresponding to 1.667 angstroms on the specimen. Subsequently, adjacent pixels were 3 x 3 averaged to yield a ...詳細: Micrographs were digitized on a Zeiss SCAI scanner at a pixel size of 7 micron, corresponding to 1.667 angstroms on the specimen. Subsequently, adjacent pixels were 3 x 3 averaged to yield a pixel size of 5 angstroms. |
| Tilt angle min | 0 |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / ソフトウェア - 名称: SPIDER 詳細: The tilted images were corrected for the effects of the contrast transfer function (CTF) by phase flipping, taking into account the defocus gradient across the micrographs and the position of ...詳細: The tilted images were corrected for the effects of the contrast transfer function (CTF) by phase flipping, taking into account the defocus gradient across the micrographs and the position of each particle. Images were processed using SPIDER version 11.12 and 15.06. Three-dimensional reconstruction was performed by the random conical tilt method in SPIDER. The particles were windowed into 64 x 64 pixel boxes. 使用した粒子像数: 1556 |
|---|
 ムービー
ムービー コントローラー
コントローラー