354件のPDBエントリーが新たに公開されました。(2024-04-24付)
354件のPDBエントリーが新たに公開されました。 公開済みのPDBエントリーは、218853件になりました。(2024-04-24付)
その他の更新情報は、PDB / EMDB 最新情報ページをご覧ください。
PDBj Newsletter Vol.24公開のお知らせ
[wwPDB] Paper Published on NMR Restraint Validation
We are pleased to announce the publication of this manuscript, addressing the challenge of validation of experimental biomolecular NMR structures against restraint data.
The NMR exchange (NEF) and NMR-STAR formats provide a standardized approach for representing commonly used NMR restraints. Using these restraint formats, a standardized validation system for assessing structural models of biopolymers against restraints has been developed and implemented in the wwPDB OneDep data harvesting system.
The resulting wwPDB Restraint Violation Report provides a model vs data assessment of biomolecule structures determined using distance and dihedral restraints, with extensions to other restraint types currently being implemented. These tools are useful for assessing NMR models, as well as for assessing biomolecular structure predictions based on distance restraints.
We present the rationale for model-vs-data restraint validation by the wwPDB, together with summary of validation tools and reports for NMR distance and dihedral restraints that have been developed, as implemented in the wwPDB validation pipeline and recommended by the wwPDB NMR-VTF committee.
Restraint Validation of Biomolecular Structures Determined by NMR in the Protein Data Bank
Kumaran Baskaran, Eliza Ploskon, Roberto Tejero, Masashi Yokochi, Deborah Harrus, Yuhe Liang, Ezra Peisach, Irina Persikova, Theresa A Ramelot, Monica Sekharan, James Tolchard, John D Westbrook, Benjamin Bardiaux, Charles Schwieters, Ardan Patwardhan, Sameer Velankar, Stephen K Burley, Genji Kurisu, Jeffrey C Hoch, Gaetano T Montelione, Geerten W Vuister, Jasmine Y Young
(2024) Structure 32, 1–14: doi: 10.1016/j.str.2024.02.011
The wwPDB plans to further enhance validation report by providing model-vs-data quality assessment for other kinds of restraints based on community recommendation and improve data representation on structures with multiple conformation states.

[ wwPDB News ]
[wwPDB] Paper Published on CryoEM Archiving and Validation Recommendations
A workshop was held at EMBL-EBI (Hinxton, UK) in January 2020 to discuss data requirements for deposition and validation of cryoEM structures, with a focus on single-particle analysis and setting community recommendations. The outcomes of this meeting have now been published in this manuscript which highlights the recent achievements made by the wwPDB in the space of 3DEM validation and the community recommendations going forward. Some of these recommendations have already been implemented, such as a three-tiered strategy powered by the Validation Analysis (VA) pipeline for the dissemination of validation information and ensuring the that VA can be run by external applications.
Community recommendations on cryoEM data archiving and validation
Gerard J. Kleywegt, Paul D. Adams, Sarah J. Butcher, Cathy Lawson, Alexis Rohou, Peter B. Rosenthal, Sriram Subramaniam, Maya Topf, Sanja Abbott, Philip R. Baldwin, John M. Berrisford, Gérard Bricogne, Preeti Choudhary, Tristan I. Croll, Radostin Danev, Sai J. Ganesan, Timothy Grant, Aleksandras Gutmanas, Richard Henderson, J. Bernard Heymann, Juha T. Huiskonen, Andrei Istrate, Takayuki Kato, Gabriel C. Lander, Shee-Mei Lok, Steven J. Ludtke, Garib N. Murshudov, Ryan Pye, Grigore D. Pintilie, Jane S. Richardson, Carsten Sachse, Osman Salih, Sjors H.W. Scheres, Gunnar F. Schroeder, Carlos Oscar S. Sorzano, Scott M. Stagg, Zhe Wang, Rangana Warshamanage, John D. Westbrook, Martyn D. Winn, Jasmine Y. Young, Stephen K. Burley, Jeffrey C. Hoch, Genji Kurisu, Kyle Morris, Ardan Patwardhan, Sameer Velankar
(2024) IUCrJ 11: 140–151 https://doi.org/10.1107/S2052252524001246
[ wwPDB News ]
[wwPDB] Poster Prize Awarded at The Biophysical Society Meeting
The wwPDB Foundation made an award for outstanding student presentations at the 2024 Biophysical Society Meeting (February 10-14, Philadelphia, PA).
Mechanisms of Light Signalling and Allosteric Regulation in Dual Sensor Photoreceptor PPHK
Irin Pottanani Tom (1), Heewhan Shin (1), Chang Liu (2), Indika Kumaeapperuma (1), Zhong Ren (1), Minglei Zhao (1),
Xiaojing Yang (1)
1) University of Illinois at Chicago, 2) University of Chicago
Many thanks to The Biophysical Society organizers and poster prize judges for making this award possible.
The wwPDB Foundation was established in 2010 to raise funds in support of the outreach activities of the wwPDB. The Foundation raised funds to help support PDB50 events, workshops, and educational publications. The Foundation is chartered as a 501(c)(3) entity exclusively for scientific, literary, charitable, and educational purposes.
The wwPDB Foundation is grateful for our industrial sponsors: Discngine, OpenEye Scientific, Roivant Sciences, Rigaku, and ThermoFisher Scientific. Individual sponsorships are also available.
Consider supporting the next 50 years of PDB's spirit of openness, cooperation, and education with a donation to the wwPDB Foundation.
[ wwPDB News ]
[wwPDB] 初期モデルの収集を強化しました
この度、新しい PDBx/mmCIF カテゴリ、_pdbx_initial_refinement_model を導入し、 X 線、電子顕微鏡、NMR 法の初期モデルに関する情報収集を改善しました。
これにより、実験的に得られたモデルと計算で得られたモデルが区別されます。 初期モデルが得られたリソースの出どころ(例えば、PDB、AlphaFoldDB、RoseTTAFoldなど)と そのアクセッションコード又は識別子が公開されている場合は、その情報を取得できます。
詳細な定義については、 pdbx_initial_refinement_modelをご覧ください。 以下はその例です。
_pdbx_initial_refinement_model.id 1 _pdbx_initial_refinement_model.entity_id_list 1 _pdbx_initial_refinement_model.type 'experimental model' _pdbx_initial_refinement_model.source_name PDB _pdbx_initial_refinement_model.accession_code 3LTQ
wwPDBは、すべてのPDBユーザーとソフトウェア開発者がコードを見直し、 将来のアプリケーションに対してはこの定義を採用することをお勧めします。
[ wwPDB News ]
サービス提供停止のお知らせ(ASH, DASH, SeSAW, MAFFTash, Spanner, SFAS)
以下のサービスについて、IT技術者の不足により継続が困難となったため、提供を停止します。
- ASH
- DASH
- SeSAW
- MAFFTash
- Spanner
- SFAS
[wwPDB] 10,000件以上のPDB登録を処理したバイオキュレータ
wwPDBの張 羽澄さんが処理したPDBエントリーが10,000件に到達しました。おめでとうございます。 張さんは、このマイルストーンに到達したPDBjで2番目、wwPDB全体で5番目のバイオキュレーターとなります。 2021年4月に見学有美子さんがwwPDBで初めてこのマイルストーンを達成しました。
張さんは大阪大学で環境工学の博士号を取得、国立循環器病研究センター勤務を経てPDBに加わりました。 2007年にPDBに加わり、現在は大阪の蛋白質研究奨励会内にあるPDBj支部に勤務しています。 科学データとさまざまな実験技術を深く理解し、卓越した品質のデータ編集に献身する高度な専門家としての地位を確立しました。 彼女の深いデータ編集の専門知識と卓越性への取り組みは、科学研究に重要な高品質のデータアーカイブに貢献してきました。 私たちは、張さんのこの素晴らしい快挙を祝福し、彼女の今後の活躍を期待しています。
 蛋白質研究奨励会の長谷俊治理事長と張 羽澄博士
蛋白質研究奨励会の長谷俊治理事長と張 羽澄博士
 Milestone tumbler.
Milestone tumbler.
[ wwPDB News ]
[wwPDB] Preprint Published on NMR Restraint Validation
 Graphical Abstract
Graphical Abstract
This manuscript addresses this challenge of validation of experimental biomolecular NMR structures against restraint data. The NMR exchange (NEF) and NMR-STAR formats provide a standardized approach for representing commonly used NMR restraints. Using these restraint formats, a standardized validation system for assessing structural models of biopolymers against restraints has been developed and implemented in the wwPDB OneDep data harvesting system. The resulting wwPDB Restraint Violation Report provides a model vs data assessment of biomolecule structures determined using distance and dihedral restraints, with extensions to other restraint types currently being implemented. These tools are useful for assessing NMR models, as well as for assessing biomolecular structure predictions based on distance restraints. We presented the rationale for model-vs-data restraint validation by the wwPDB, together with summary of validation tools and reports for NMR distance and dihedral restraints that have been developed, as implemented in the wwPDB validation pipeline and recommended by the wwPDB NMR-VTF committee.
Restraint Validation of Biomolecular Structures Determined by NMR in the Protein Data Bank
Kumaran Baskaran, Eliza Ploskon, Roberto Tejero, Masashi Yokochi, Deborah Harrus, Yuhe Liang, Ezra Peisach, Irina Persikova, Theresa A Ramelot, Monica Sekharan, James Tolchard, John D Westbrook, Benjamin Bardiaux, Charles Schwieters, Ardan Patwardhan, Sameer Velankar, Stephen K Burley, Genji Kurisu, Jeffrey C Hoch, Gaetano T Montelione, Geerten W Vuister, Jasmine Y Young
(2024) bioRxiv 2024.01.15.575520; doi: 10.1101/2024.01.15.575520
wwPDB plans to further enhance validation report by providing model-vs-data quality assessment for other kinds of restraints based on community recommendation and improve data representation on structures with multiple conformation states.
[ wwPDB News ]
[wwPDB] Preprint Published on CryoEM Archiving and Validation Recommendations

The number of released EMDB entries per year in a number of resolution bins, from 2010 until December 2023
A workshop was held at EMBL-EBI (Hinxton, UK) in January 2020 to discuss data requirements for deposition and validation of cryoEM structures, with a focus on single-particle analysis and set community recommendations.
Community recommendations on cryoEM data archiving and validation
Gerard J. Kleywegt, Paul D. Adams, Sarah J. Butcher, Cathy Lawson, Alexis Rohou, Peter B. Rosenthal, Sriram Subramaniam, Maya Topf, Sanja Abbott, Philip R. Baldwin, John M. Berrisford, Gérard Bricogne, Preeti Choudhary, Tristan I. Croll, Radostin Danev, Sai J. Ganesan, Timothy Grant, Aleksandras Gutmanas, Richard Henderson, J. Bernard Heymann, Juha T. Huiskonen, Andrei Istrate, Takayuki Kato, Gabriel C. Lander, Shee-Mei Lok, Steven J. Ludtke, Garib N. Murshudov, Ryan Pye, Grigore D. Pintilie, Jane S. Richardson, Carsten Sachse, Osman Salih, Sjors H.W. Scheres, Gunnar F. Schroeder, Carlos Oscar S. Sorzano, Scott M. Stagg, Zhe Wang, Rangana Warshamanage, John D. Westbrook, Martyn D. Winn, Jasmine Y. Young, Stephen K. Burley, Jeffrey C. Hoch, Genji Kurisu, Kyle Morris, Ardan Patwardhan, Sameer Velankar
(2023) arXiv doi: 10.48550/arXiv.2311.17640
Several community recommendations from this workshop have been incorporated into wwPDB validation reports including map analysis, FSC validation, and map-model fitness using Q-score. wwPDB plans to provide overall quality percentile on map-model fitness compared to other PDB entries in the wwPDB validation report as the next step.
[ wwPDB News ]
[wwPDB] Preprint Published on NextGen Archive
A new paper describes how the recently-announced NextGen Archive provides centralized access to integrated annotations and enriched structural information for PDB data:
NextGen Archive: Centralising Access to Integrated Annotations and Enriched Structural Information by the Worldwide Protein Data Bank
Preeti Choudhary, Zukang Feng, John Berrisford, Henry Chao, Yasuyo Ikegawa, Ezra Peisach, Dennis W. Piehl, James Smith, Ahsan Tanweer, Mihaly Varadi, John D. Westbrook, Jasmine Y. Young, Ardan Patwardhan, Kyle L. Morris, Jeffrey C. Hoch, Genji Kurisu, Sameer Velankar, Stephen K. Burley
(2023) bioRxiv doi: 10.1101/2023.10.24.563739
The PDB NextGen archive provides sequence annotation from external resources such as UniProt, SCOP2 and Pfam in addition to the content provided in the structure model files in the PDB main archive. The inclusion of UniProtKB numbering facilitates effortless structural comparisons between experimental and predicted protein models. These PDBx/mmCIF files are directly compatible with various data visualization tools, simplifying the display of annotations on 3D structure views.
[ wwPDB News ]
[wwPDB] Prizes Awarded at The Biophysical Society Japan Meeting
The wwPDB Foundation made awards to outstanding student presentations at the 2023 The Biophysical Society Japan Meeting (November 14-16, Nagoya, Japan).
Thermodynamic analysis of Fv-supercharged antibody–antigen interactions and control of interaction parameters
Keisuke Kasahara (1), Daisuke Kuroda (2), Jose Caaveiro (3), Satoru Nagatoishi (4), Kouhei Tsumoto (1,4)
1) Dept. Bioeng., Grad. Sch. Eng., Univ. Tokyo; 2) Res. Ctr. Drug Vaccine Dev., NIID;
3) Grad. Sch. Pharm. Sci., Kyusyu Univ., 4) Med. Dev. Dev. Reg. Res. Ctr., Grad. Sch. Eng., Univ. Tokyo
Harnessing the power of machine learning and high-throughput molecular dynamics simulations to predict protein-lipid interactions Kyle Ian Peter Le Huray (1,2), Frank Sobott (1), He Wang (3), Antreas Kalli (2)
1) School of Molecular and Cellular Biology, Faculty of Biological Sciences, University of Leeds, Leeds, UK; 2) Leeds Institute of Cardiovascular and Metabolic Medicine, School of Medicine, University of Leeds, Leeds, UK; 3) School of Computing, University of Leeds, Leeds, UK
 Katsuhiko Minami
Katsuhiko Minami
Replication-dependent histone (Repli-Histo) labeling revealed that chromatin motion can determine DNA replication timing
Katsuhiko Minami (1,2), Satoru Ide (1,2), Sachiko Tamura (1), Masato T. Kanemaki (1,2), Kazuhiro Maeshima (1,2)
1) National Institute of Genetics; 2) Graduate Institute for Advanced Studies, SOKENDAI
Many thanks to the meeting organizers and prize judges for making these awards possible.
The wwPDB Foundation was established in 2010 to raise funds in support of the outreach activities of the wwPDB. The Foundation raised funds to help support PDB50 events, workshops, and educational publications. The Foundation is chartered as a 501(c)(3) entity exclusively for scientific, literary, charitable, and educational purposes.
Consider supporting the next 50 years of PDB's spirit of openness, cooperation, and education with a donation to the wwPDB Foundation.
[ wwPDB News ]
[wwPDB] 拡張PDB IDフォーマット(pdb_00001abc)に関するリソースをご利用ください
wwPDBは、現行の4文字のPDBアクセッションコード(PDB ID)が2029年までにすべて消費されると予測しています。
PDBアーカイブの継続的な増加に伴い、wwPDBはPDBアクセッションコードの文字数を拡張し、"PDB "を先頭に付けることで フォーマットを改訂しました(例えば、"1abc "は "pdb_00001abc "になります)。 これにより、公開文献中のPDBエントリーをテキストマイニングで検出することが可能になり、 改訂データファイルをより有益で透明性の高い形で提供できるようになります。
拡張PDB ID(12文字)を持つエントリーは、4文字のPDB IDが消費されると、従来のPDBファイルフォーマットと互換性がなくなります。 wwPDBは、科学ジャーナル、PDBコミュニティ、ユーザーに対し、PDBx/mmCIFフォーマットと拡張PDB IDフォーマットの 使用にできるだけ早く移行することを奨励しています。
wwPDBリソースポータルページ(Extended PDB ID With 12 Characters)を通して、 PDBユーザーのこの移行を支援するリソースをご利用頂けます。 このページには、 PDB IDの拡張に関するFAQ、PDBx/mmCIFフォーマットに関する資料、 その他のPDBx/mmCIFリソースやソフトウェアツールへのリンクなど、 この変更に対応するための有用なリソースがリンクされています。 移行段階が進むにつれて、このページにはより多くのトレーニングリソースが追加される予定です。
さらに、2026年の移行期にはPDBの「ベータ」アーカイブが提供されます。 この "ベータ "アーカイブのディレクトリ構造は、PDBバージョンアーカイブのデータ構成を反映し、 https://files-beta.org/pub/pdb/data/entries/two-letter-hash/pdb_accession_code/entry_data_File_names という構成になります。 2文字のハッシュはn-2文字とn-3文字(後ろから3番目と2番目の文字)に基づいています。 例えば、PDBエントリー PDB_12345678 はディレクトリ/67/の下になります。 これにより、例えばPDBエントリー1abcが/abの下にある現在のPDBアーカイブとの一貫性が保たれます。
4文字のPDBアクセッションコードがすべて消費されると、このPDB「ベータ」アーカイブはPDBメインアーカイブとなり、現在のPDBアーカイブは削除されます。
ソフトウェア採用のための拡張PDB IDを含むサンプルファイルを GitHubから ダウンロードしてください。
wwPDB recently announced that PDB three-character Chemical Component IDs have been consumed. Five-character alphanumeric accession codes for CCD IDs are now issued by the OneDep system. wwPDBは最近、PDBの3文字の化合物IDが消費されたことを発表しました。 OneDepシステムでは、CCD IDに対して、5文字英数字アクセッションコードが発行されるようになりました。
ご質問やコメントは、info@wwpdb.orgまで、英語でご連絡ください。
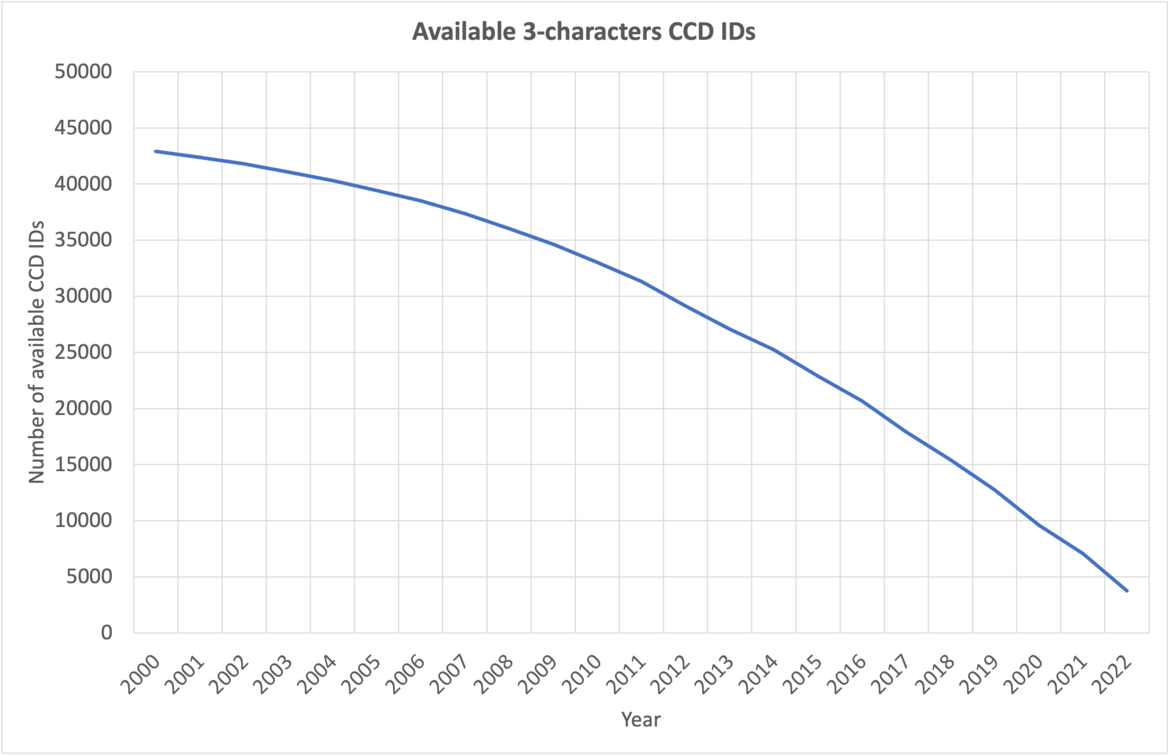 利用可能な3文字のCCD ID数の年推移
利用可能な3文字のCCD ID数の年推移
[ wwPDB News ]
[wwPDB] PDBアーカイブとEMDBアーカイブのスナップショットを公開しました

新しいアーカイブスナップショットをご利用頂けます
2024年1月2日現在のPDBアーカイブ (ftp://ftp.wwpdb.org、 https://s3.rcsb.org/又は PDBj:ftp://ftp.pdbj.org)のスナップショットを、 スナップショットサイト (wwPDB:ftp://snapshots.wwpdb.org、https://s3snapshots.rcsb.org/(AWS)又は PDBj:ftp://snapshots.pdbj.org)に追加しました。 2005年以来、毎年、スナップショットをアーカイブし、PDBアーカイブに関する研究に役立つデータセットを提供し続けています。
ディレクトリ 20240101は、 2024年1月2日時点での214,121件の実験的に決定された 座標ファイルと、関連する実験データを含んでいます。 座標及び関連データのファイルは、PDBx/mmCIFフォーマット、PDBフォーマット、XMLフォーマットファイルで、それぞれご利用頂けます。 各ファイルの日付とタイムスタンプは、そのファイルが最後に更新された日付を表しています。 上記ディレクトリ内のPDBアーカイブのスナップショットのサイズは、1,242 GBとなっています。
2024年1月1日現在のEMDBアーカイブ(ftp://ftp.ebi.ac.uk/pub/databases/emdb)のスナップショットは、 ftp://ftp.ebi.ac.uk/pub/databases/emdb_vault/20240101/と、 ftp://snapshots.pdbj.org/20240101/からご利用頂けます。 公開済みエントリー(32,033件)と廃止されたエントリー(282件)に対する、マップファイルとメタデータのXMLファイルを含んでおり、 スナップショットのサイズは、14 TBとなっています。
[ wwPDB News ]











