+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8594 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Automated tomogram annotation of PC12 cell | |||||||||
 マップデータ マップデータ | Automated tomogram annotation of PC12 cell | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Rattus (クマネズミ属) Rattus (クマネズミ属) | |||||||||
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Chen M / Dai W / Schmid MF / Chiu W / Ludtke SJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2017 ジャーナル: Nat Methods / 年: 2017タイトル: Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. 著者: Muyuan Chen / Wei Dai / Stella Y Sun / Darius Jonasch / Cynthia Y He / Michael F Schmid / Wah Chiu / Steven J Ludtke /   要旨: Cellular electron cryotomography offers researchers the ability to observe macromolecules frozen in action in situ, but a primary challenge with this technique is identifying molecular components ...Cellular electron cryotomography offers researchers the ability to observe macromolecules frozen in action in situ, but a primary challenge with this technique is identifying molecular components within the crowded cellular environment. We introduce a method that uses neural networks to dramatically reduce the time and human effort required for subcellular annotation and feature extraction. Subsequent subtomogram classification and averaging yield in situ structures of molecular components of interest. The method is available in the EMAN2.2 software package. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8594.map.gz emd_8594.map.gz | 206.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8594-v30.xml emd-8594-v30.xml emd-8594.xml emd-8594.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8594.png emd_8594.png | 346.1 KB | ||
| その他 |  emd_8594_additional_1.map.gz emd_8594_additional_1.map.gz emd_8594_additional_2.map.gz emd_8594_additional_2.map.gz emd_8594_additional_3.map.gz emd_8594_additional_3.map.gz emd_8594_additional_4.map.gz emd_8594_additional_4.map.gz | 124.8 MB 137.4 MB 98.5 MB 113.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8594 http://ftp.pdbj.org/pub/emdb/structures/EMD-8594 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8594 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8594 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8594.map.gz / 形式: CCP4 / 大きさ: 223.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8594.map.gz / 形式: CCP4 / 大きさ: 223.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Automated tomogram annotation of PC12 cell | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 28.6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Automatic annotation of double layer membrane in PC12 cell
| ファイル | emd_8594_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Automatic annotation of double layer membrane in PC12 cell | ||||||||||||
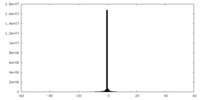
| 投影像・断面図 |
| ||||||||||||
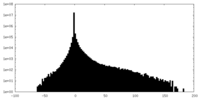
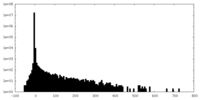
| 密度ヒストグラム |
-追加マップ: Automatic annotation of microtubules in PC12 cell
| ファイル | emd_8594_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Automatic annotation of microtubules in PC12 cell | ||||||||||||
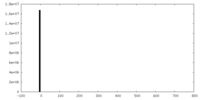
| 投影像・断面図 |
| ||||||||||||
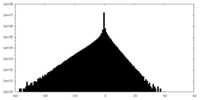
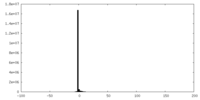
| 密度ヒストグラム |
-追加マップ: Automatic annotation of ribosomes in PC12 cell
| ファイル | emd_8594_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Automatic annotation of ribosomes in PC12 cell | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
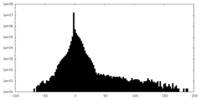
| 密度ヒストグラム |
-追加マップ: Automatic annotation of single layer membrane in PC12 cell
| ファイル | emd_8594_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Automatic annotation of single layer membrane in PC12 cell | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PC12 cell
| 全体 | 名称: PC12 cell |
|---|---|
| 要素 |
|
-超分子 #1: PC12 cell
| 超分子 | 名称: PC12 cell / タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Rattus (クマネズミ属) Rattus (クマネズミ属) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 切片作成 | その他: NO SECTIONING |
| 位置合わせマーカー | Manufacturer: EMS / 直径: 14 nm |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 2.0 e/Å2 |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / 使用した粒子像数: 30 |
|---|
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X