+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5858 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structures of Cas9 endonucleases reveal RNA-mediated conformational activation | |||||||||
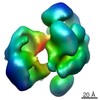
 マップデータ マップデータ | Reconstruction of apo-Cas9 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  CRISPR-Cas (CRISPR) / crRNA / CRISPR-Cas (CRISPR) / crRNA /  tracrRNA / tracrRNA /  Cas9 (Cas9) / Cas9 (Cas9) /  genome engineering (ゲノム編集) / bacterial immunity genome engineering (ゲノム編集) / bacterial immunity | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / 3'-5' exonuclease activity / DNA endonuclease activity / defense response to virus /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  DNA binding / DNA binding /  RNA binding / RNA binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 19.0 Å ネガティブ染色法 / 解像度: 19.0 Å | |||||||||
 データ登録者 データ登録者 | Jinek M / Jiang F / Taylor DW / Sternberg SH / Kaya E / Ma E / Anders C / Hauer M / Zhou K / Lin S ...Jinek M / Jiang F / Taylor DW / Sternberg SH / Kaya E / Ma E / Anders C / Hauer M / Zhou K / Lin S / Kaplan M / Iavarone AT / Charpentier E / Nogales E / Doudna JA | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2014 ジャーナル: Science / 年: 2014タイトル: Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. 著者: Martin Jinek / Fuguo Jiang / David W Taylor / Samuel H Sternberg / Emine Kaya / Enbo Ma / Carolin Anders / Michael Hauer / Kaihong Zhou / Steven Lin / Matias Kaplan / Anthony T Iavarone / ...著者: Martin Jinek / Fuguo Jiang / David W Taylor / Samuel H Sternberg / Emine Kaya / Enbo Ma / Carolin Anders / Michael Hauer / Kaihong Zhou / Steven Lin / Matias Kaplan / Anthony T Iavarone / Emmanuelle Charpentier / Eva Nogales / Jennifer A Doudna /  要旨: Type II CRISPR (clustered regularly interspaced short palindromic repeats)-Cas (CRISPR-associated) systems use an RNA-guided DNA endonuclease, Cas9, to generate double-strand breaks in invasive DNA ...Type II CRISPR (clustered regularly interspaced short palindromic repeats)-Cas (CRISPR-associated) systems use an RNA-guided DNA endonuclease, Cas9, to generate double-strand breaks in invasive DNA during an adaptive bacterial immune response. Cas9 has been harnessed as a powerful tool for genome editing and gene regulation in many eukaryotic organisms. We report 2.6 and 2.2 angstrom resolution crystal structures of two major Cas9 enzyme subtypes, revealing the structural core shared by all Cas9 family members. The architectures of Cas9 enzymes define nucleic acid binding clefts, and single-particle electron microscopy reconstructions show that the two structural lobes harboring these clefts undergo guide RNA-induced reorientation to form a central channel where DNA substrates are bound. The observation that extensive structural rearrangements occur before target DNA duplex binding implicates guide RNA loading as a key step in Cas9 activation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5858.map.gz emd_5858.map.gz | 10.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5858-v30.xml emd-5858-v30.xml emd-5858.xml emd-5858.xml | 11.6 KB 11.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5858.png emd_5858.png | 165.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5858 http://ftp.pdbj.org/pub/emdb/structures/EMD-5858 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5858 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5858 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5858.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5858.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of apo-Cas9 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.18 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : apo-Cas9
| 全体 | 名称: apo-Cas9 |
|---|---|
| 要素 |
|
-超分子 #1000: apo-Cas9
| 超分子 | 名称: apo-Cas9 / タイプ: sample / ID: 1000 / 集合状態: monomer of apo-Cas9 / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 160 KDa |
-分子 #1: CRISPR-associated endonuclease Cas9
| 分子 | 名称: CRISPR-associated endonuclease Cas9 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Cas9 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 159 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: Rosetta 2(DE3) Escherichia coli (大腸菌) / 組換株: Rosetta 2(DE3) |
| 配列 | UniProtKB: CRISPR-associated endonuclease Cas9/Csn1 GO: defense response to virus, maintenance of CRISPR repeat elements, 3'-5' exonuclease activity, DNA endonuclease activity,  RNA binding, RNA binding,  DNA binding DNA bindingInterPro:  INTERPRO: IPR010145, INTERPRO: IPR010145,  INTERPRO: IPR025978, HNH nuclease INTERPRO: IPR025978, HNH nuclease |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-Cl pH 7.5, 100 mM KCl, 5 mM MgCl2, 1 mM DTT, 5% glycerol |
| 染色 | タイプ: NEGATIVE 詳細: After adsorption for 1 min, we stained the samples consecutively with six droplets of 2% (w/v) uranyl acetate solution, gently blotted off the residual stain, and air-dried the sample in a fume hood. |
| グリッド | 詳細: glow-discharged 400 mesh continuous carbon grids |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): -1.5 µm / 最小 デフォーカス(公称値): -0.5 µm / 倍率(公称値): 100000 Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): -1.5 µm / 最小 デフォーカス(公称値): -0.5 µm / 倍率(公称値): 100000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 温度 | 平均: 78 K |
| アライメント法 | Legacy - 非点収差: Objective astigmatism was corrected at 210,000 times magnification Legacy - Electron beam tilt params: 0 |
| 詳細 | Data acquired using Leginon. |
| 日付 | 2013年3月19日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 281 / 平均電子線量: 20 e/Å2 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
- 画像解析
画像解析
| CTF補正 | 詳細: whole micrograph |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 19.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, SPARX / 詳細: Image pre-processing performed in Appion. / 使用した粒子像数: 18000 |
| 詳細 | Particles were selected using template based picking in Appion. |
 ムービー
ムービー コントローラー
コントローラー