+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5344 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Mediator-RNA polymerase II-TFIIF assembly in the absence of activator | |||||||||
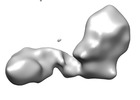
 マップデータ マップデータ | activator-free Mediator bound to pol II & TFIIF | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  transcription (転写 (生物学)) / Mediator / transcription (転写 (生物学)) / Mediator /  RNA polymerase II (RNAポリメラーゼII) / RNA polymerase II (RNAポリメラーゼII) /  TFIIF / activator TFIIF / activator | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / クライオ電子顕微鏡法 /  ネガティブ染色法 / 解像度: 32.0 Å ネガティブ染色法 / 解像度: 32.0 Å | |||||||||
 データ登録者 データ登録者 | Bernecky C / Taatjes DJ | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2012 ジャーナル: J Mol Biol / 年: 2012タイトル: Activator-mediator binding stabilizes RNA polymerase II orientation within the human mediator-RNA polymerase II-TFIIF assembly. 著者: Carrie Bernecky / Dylan J Taatjes /  要旨: The human Mediator complex controls RNA polymerase II (pol II) function in ways that remain incompletely understood. Activator-Mediator binding alters Mediator structure, and these activator-induced ...The human Mediator complex controls RNA polymerase II (pol II) function in ways that remain incompletely understood. Activator-Mediator binding alters Mediator structure, and these activator-induced structural shifts appear to play key roles in regulating transcription. A recent cryo-electron microscopy (EM) analysis revealed that pol II adopted a stable orientation within a Mediator-pol II-TFIIF assembly in which Mediator was bound to the activation domain of viral protein 16 (VP16). Whereas TFIIF was shown to be important for orienting pol II within this assembly, the potential role of the activator was not assessed. To determine how activator binding might affect pol II orientation, we isolated human Mediator-pol II-TFIIF complexes in which Mediator was not bound to an activator. Cryo-EM analysis of this assembly, coupled with pol II crystal structure docking, revealed that pol II binds Mediator at the same general location; however, in contrast to VP16-bound Mediator, pol II does not appear to stably orient in the absence of an activator. Variability in pol II orientation might be important mechanistically, perhaps to enable sense and antisense transcription at human promoters. Because Mediator interacts extensively with pol II, these results suggest that Mediator structural shifts induced by activator binding help stably orient pol II prior to transcription initiation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5344.map.gz emd_5344.map.gz | 14.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5344-v30.xml emd-5344-v30.xml emd-5344.xml emd-5344.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5344_1.png emd_5344_1.png | 45.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5344 http://ftp.pdbj.org/pub/emdb/structures/EMD-5344 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5344 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5344 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5344.map.gz / 形式: CCP4 / 大きさ: 15.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5344.map.gz / 形式: CCP4 / 大きさ: 15.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | activator-free Mediator bound to pol II & TFIIF | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.22 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Assembly of human Mediator, RNA polymerase II, and TFIIF
| 全体 | 名称: Assembly of human Mediator, RNA polymerase II, and TFIIF |
|---|---|
| 要素 |
|
-超分子 #1000: Assembly of human Mediator, RNA polymerase II, and TFIIF
| 超分子 | 名称: Assembly of human Mediator, RNA polymerase II, and TFIIF タイプ: sample / ID: 1000 / 集合状態: one Mediator complex binds one pol II-TFIIF / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 1.9 MDa |
-分子 #1: core Mediator
| 分子 | 名称: core Mediator / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Mediator 詳細: affinity purified using an antibody to the MED26 subunit 集合状態: 26 subunit complex / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human / 細胞: HeLa / Organelle: Nucleus Homo sapiens (ヒト) / 別称: human / 細胞: HeLa / Organelle: Nucleus |
| 分子量 | 理論値: 1.2 MDa |
-分子 #2: RNA polymerase II
| 分子 | 名称: RNA polymerase II / タイプ: protein_or_peptide / ID: 2 / Name.synonym: pol II / 詳細: unphosphorylated / 集合状態: 12-subunit complex / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞: HeLa / Organelle: Nucleus Homo sapiens (ヒト) / 別称: Human / 細胞: HeLa / Organelle: Nucleus |
| 分子量 | 理論値: 520 KDa |
-分子 #3: TFIIF
| 分子 | 名称: TFIIF / タイプ: protein_or_peptide / ID: 3 / Name.synonym: TFIIF / 詳細: RAP74 and RAP30 expressed separately then combined / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 10 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法, ネガティブ染色法,  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 詳細: 20 mM HEPES, 0.10 mM EDTA, 150 mM KCl, 0.02% NP-40, 35% glycerol |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: grids with adsorbed protein washed 3x with buffer containing 5% trehalose, 20 mM HEPES, 100 mM KCl, and 0.10 mM EDTA, then subject to cryo-negative staining in a saturated solution (1.2M) of ...詳細: grids with adsorbed protein washed 3x with buffer containing 5% trehalose, 20 mM HEPES, 100 mM KCl, and 0.10 mM EDTA, then subject to cryo-negative staining in a saturated solution (1.2M) of ammonium molybdate (pH 7.5) |
| グリッド | 詳細: thin carbon-coated holey carbon 400 mesh copper grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 90 K / 装置: OTHER 手法: blot for 2 seconds, dry for 3 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 29000 Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダー: side entry / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 12.7 µm / 実像数: 139 / 平均電子線量: 15 e/Å2 / Od range: 1 / ビット/ピクセル: 16 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: each micrograph |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 32.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider / 使用した粒子像数: 5489 |
| 詳細 | The particles were selected interactively at the computer terminal. |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Situs |
| 詳細 | Protocol: rigid body. the TFIIS chain S was removed before fitting |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: contour-based Laplacian correlation |
 ムービー
ムービー コントローラー
コントローラー