+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8tux | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
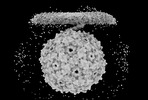
| タイトル | Capsid of mature PP7 virion with 3'end region of PP7 genomic RNA | ||||||||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / Maturation protein / PP7 / pepevirus VIRAL PROTEIN (ウイルスタンパク質) / Maturation protein / PP7 / pepevirus | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virion attachment to host cell pilus / T=3 icosahedral viral capsid /  virion component / virion component /  regulation of translation / symbiont entry into host cell / regulation of translation / symbiont entry into host cell /  RNA binding / identical protein binding RNA binding / identical protein binding類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Pseudomonas phage PP7 (ファージ) Pseudomonas phage PP7 (ファージ) | ||||||||||||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Thongchol, J. / Zhang, J. / Zeng, L. | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Removal of type IV pili by a small RNA virus. 著者: Jirapat Thongchol / Zihao Yu / Laith Harb / Yiruo Lin / Matthias Koch / Matthew Theodore / Utkarsh Narsaria / Joshua Shaevitz / Zemer Gitai / Yinghao Wu / Junjie Zhang / Lanying Zeng /  要旨: The retractile type IV pilus (T4P) is important for virulence of the opportunistic human pathogen . The single-stranded RNA (ssRNA) phage PP7 binds to T4P and is brought to the cell surface through ...The retractile type IV pilus (T4P) is important for virulence of the opportunistic human pathogen . The single-stranded RNA (ssRNA) phage PP7 binds to T4P and is brought to the cell surface through pilus retraction. Using fluorescence microscopy, we discovered that PP7 detaches T4P, which impairs cell motility and restricts the pathogen's virulence. Using cryo-electron microscopy, mutagenesis, optical trapping, and Langevin dynamics simulation, we resolved the structure of PP7, T4P, and the PP7/T4P complex and showed that T4P detachment is driven by the affinity between the phage maturation protein and its bound pilin, plus the pilus retraction force and speed, and pilus bending. Pilus detachment may be widespread among other ssRNA phages and their retractile pilus systems and offers new prospects for antibacterial prophylaxis and therapeutics. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8tux.cif.gz 8tux.cif.gz | 4.3 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8tux.ent.gz pdb8tux.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  8tux.json.gz 8tux.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tu/8tux https://data.pdbj.org/pub/pdb/validation_reports/tu/8tux ftp://data.pdbj.org/pub/pdb/validation_reports/tu/8tux ftp://data.pdbj.org/pub/pdb/validation_reports/tu/8tux | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  41632MC  8tumC  8tuwC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 30456.055 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Pseudomonas phage PP7 (ファージ) / 参照: GenBank: NC_001628 Pseudomonas phage PP7 (ファージ) / 参照: GenBank: NC_001628 | ||
|---|---|---|---|
| #2: タンパク質 | 分子量: 50763.766 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Pseudomonas phage PP7 (ファージ) / 参照: UniProt: Q38061 Pseudomonas phage PP7 (ファージ) / 参照: UniProt: Q38061#3: タンパク質 |  カプシド / CP / Coat protein カプシド / CP / Coat protein分子量: 13906.811 Da / 分子数: 178 / 由来タイプ: 天然 / 由来: (天然)  Pseudomonas phage PP7 (ファージ) / 参照: UniProt: P03630 Pseudomonas phage PP7 (ファージ) / 参照: UniProt: P03630 |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Pseudomonas phage PP7 / タイプ: COMPLEX 詳細: Maturation proteins of PP7, 3'end region of PP7 genomic RNA, coat protein shell of PP7 Entity ID: all / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  Pseudomonas phage PP7 (ファージ) Pseudomonas phage PP7 (ファージ) |
| ウイルスについての詳細 | 中空か: NO / エンベロープを持つか: NO / 単離: SPECIES / タイプ: VIRION |
| 天然宿主 | 生物種: Pseudomonas aeruginosa |
| ウイルス殻 | 直径: 280 nm |
| 緩衝液 | pH: 7.4 詳細: 137 mM NaCl, 2.7 mM KCl, 8 mM Na2HPO4, and 2 mM KH2PO4, pH 7.4 |
| 緩衝液成分 | 濃度: 1 x / 名称: PBS |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2200 nm / 最小 デフォーカス(公称値): 1200 nm Bright-field microscopy / 最大 デフォーカス(公称値): 2200 nm / 最小 デフォーカス(公称値): 1200 nm |
| 撮影 | 電子線照射量: 51.5 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | |||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | |||||||||
3次元再構成 | 解像度: 3.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 644601 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj






























