+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8tie | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
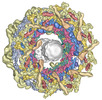
| タイトル | Double nuclear outer ring of Nup84-complexes from the yeast NPC | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード |  TRANSPORT PROTEIN (運搬体タンパク質) / TRANSPORT PROTEIN (運搬体タンパク質) /  nuclear pore complex (核膜孔) / nucleocytoplasmic transport / nuclear pore complex (核膜孔) / nucleocytoplasmic transport /  nucleoporin (ヌクレオポリン) / nucleoporin (ヌクレオポリン) /  membrane protein (膜タンパク質) / membrane protein (膜タンパク質) /  translocase (輸送酵素) translocase (輸送酵素) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA export from nucleus in response to heat stress / Seh1-associated complex / positive regulation of ER to Golgi vesicle-mediated transport / protein exit from endoplasmic reticulum / COPII-coated vesicle budding / COPII-mediated vesicle transport / nuclear pore localization / nuclear pore central transport channel / telomere tethering at nuclear periphery / regulation of TORC1 signaling ...mRNA export from nucleus in response to heat stress / Seh1-associated complex / positive regulation of ER to Golgi vesicle-mediated transport / protein exit from endoplasmic reticulum / COPII-coated vesicle budding / COPII-mediated vesicle transport / nuclear pore localization / nuclear pore central transport channel / telomere tethering at nuclear periphery / regulation of TORC1 signaling / nuclear pore outer ring / COPII vesicle coat / post-transcriptional tethering of RNA polymerase II gene DNA at nuclear periphery / positive regulation of protein exit from endoplasmic reticulum / structural constituent of nuclear pore / vacuolar membrane / silent mating-type cassette heterochromatin formation / nucleocytoplasmic transport / subtelomeric heterochromatin formation /  ribosomal large subunit export from nucleus / positive regulation of TOR signaling / mRNA transport / mRNA export from nucleus / ribosomal large subunit export from nucleus / positive regulation of TOR signaling / mRNA transport / mRNA export from nucleus /  核膜孔 / 核膜孔 /  : / positive regulation of TORC1 signaling / protein export from nucleus / cell periphery / protein import into nucleus / double-strand break repair / : / positive regulation of TORC1 signaling / protein export from nucleus / cell periphery / protein import into nucleus / double-strand break repair /  核膜 / 核膜 /  核膜 / 核膜 /  chromosome, telomeric region / endoplasmic reticulum membrane / structural molecule activity / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / chromosome, telomeric region / endoplasmic reticulum membrane / structural molecule activity / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II /  小胞体 / positive regulation of transcription by RNA polymerase II / identical protein binding 小胞体 / positive regulation of transcription by RNA polymerase II / identical protein binding類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.1 Å クライオ電子顕微鏡法 / 解像度: 8.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Akey, C.W. / Echeverria, I. / Ouch, C. / Fernandez-Martinez, J. / Rout, M.P. | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Implications of a multiscale structure of the yeast nuclear pore complex. 著者: Christopher W Akey / Ignacia Echeverria / Christna Ouch / Ilona Nudelman / Yi Shi / Junjie Wang / Brian T Chait / Andrej Sali / Javier Fernandez-Martinez / Michael P Rout /   要旨: Nuclear pore complexes (NPCs) direct the nucleocytoplasmic transport of macromolecules. Here, we provide a composite multiscale structure of the yeast NPC, based on improved 3D density maps from ...Nuclear pore complexes (NPCs) direct the nucleocytoplasmic transport of macromolecules. Here, we provide a composite multiscale structure of the yeast NPC, based on improved 3D density maps from cryogenic electron microscopy and AlphaFold2 models. Key features of the inner and outer rings were integrated into a comprehensive model. We resolved flexible connectors that tie together the core scaffold, along with equatorial transmembrane complexes and a lumenal ring that anchor this channel within the pore membrane. The organization of the nuclear double outer ring reveals an architecture that may be shared with ancestral NPCs. Additional connections between the core scaffold and the central transporter suggest that under certain conditions, a degree of local organization is present at the periphery of the transport machinery. These connectors may couple conformational changes in the scaffold to the central transporter to modulate transport. Collectively, this analysis provides insights into assembly, transport, and NPC evolution. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8tie.cif.gz 8tie.cif.gz | 1.6 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8tie.ent.gz pdb8tie.ent.gz | 1.3 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8tie.json.gz 8tie.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ti/8tie https://data.pdbj.org/pub/pdb/validation_reports/ti/8tie ftp://data.pdbj.org/pub/pdb/validation_reports/ti/8tie ftp://data.pdbj.org/pub/pdb/validation_reports/ti/8tie | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  41285MC  8t9lC  8tj5C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 7種, 14分子 albmcndoepfqgr
| #1: タンパク質 | 分子量: 120560.328 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: long arm of Nup84 Y-complex / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / Plasmid details: nucleus / 株: MATa ade2-1 ura3-1 his3-11 / 参照: UniProt: P35729 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / Plasmid details: nucleus / 株: MATa ade2-1 ura3-1 his3-11 / 参照: UniProt: P35729#2: タンパク質 | 分子量: 84972.438 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: short arm of Nup84 Y-complex / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: P46673 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: P46673#3: タンパク質 | 分子量: 145810.578 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: stem junction of Nup84 Y-complex / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: A0A8H4C085 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: A0A8H4C085#4: タンパク質 |  Protein targeting Protein targeting分子量: 33082.965 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: cofactor of Nup145C on stem of Nup84 Y-complex / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: Q04491 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: Q04491#5: タンパク質 | 分子量: 34652.078 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: cofactor Nup85 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303#6: タンパク質 | 分子量: 83718.867 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: tail of Nup84 Y-complex / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: P52891 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: P52891#7: タンパク質 | 分子量: 133452.672 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: tail Nup84 Y-complex / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: A0A6V8RYD2 Saccharomyces cerevisiae (パン酵母) / 細胞株: W303 / 参照: UniProt: A0A6V8RYD2 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 |
| |||||||||||||||||||||
| 由来(天然) |
| |||||||||||||||||||||
| 緩衝液 | pH: 7.5 詳細: 20mM HEPES,50mM Potassium acetate,20mM NaCl,2mM MgCl2,1mM DTT | |||||||||||||||||||||
| 試料 | 濃度: 0.1 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES / 詳細: One step affinity purified : YES / 詳細: One step affinity purified | |||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK III / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 283 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS 詳細: Preliminary grid screening done manually with individual images of low magnification montages of candidate meshes. |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(補正後): 37651 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 1500 nm / Cs Bright-field microscopy / 倍率(補正後): 37651 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 1500 nm / Cs : 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 40 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 4015 / 詳細: 3218 images retained after triage |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
| 画像スキャン | 横: 3838 / 縦: 3710 / 動画フレーム数/画像: 40 / 利用したフレーム数/画像: 2-40 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | 詳細: CTF correction applied in RELION during the alignment and reconstruction タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 26049 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 8.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 29655 / アルゴリズム: FOURIER SPACE 詳細: multibody in RELION followed by refinements in cryosparc 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL / Target criteria: cross correlation 詳細: Molecular dynamics flexible fitting after docking Alphafold2 CSMs for Nups into the 3D map of double Y complex ring with Chimera | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 詳細: none / Source name: Other / タイプ: experimental model |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj
