登録情報 データベース : PDB / ID : 2a5xタイトル Crystal Structure of a Cross-linked Actin Dimer Actin, alpha skeletal muscle キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Oryctolagus cuniculus (ウサギ)手法 / / / 解像度 : 2.49 Å データ登録者 Kudryashov, D.S. / Sawaya, M.R. / Adisetiyo, H. / Norcross, T. / Hegyi, G. / Reisler, E. / Yeates, T.O. ジャーナル : Proc.Natl.Acad.Sci.Usa / 年 : 2005タイトル : The crystal structure of a cross-linked actin dimer suggests a detailed molecular interface in F-actin著者 : Kudryashov, D.S. / Sawaya, M.R. / Adisetiyo, H. / Norcross, T. / Hegyi, G. / Reisler, E. / Yeates, T.O. 履歴 登録 2005年7月1日 登録サイト / 処理サイト 改定 1.0 2005年8月23日 Provider / タイプ 改定 1.1 2008年4月30日 Group 改定 1.2 2011年7月13日 Group / Version format compliance改定 1.3 2017年10月11日 Group / カテゴリ / Item / _software.name改定 1.4 2023年8月23日 Group Advisory / Data collection ... Advisory / Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_remark / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_remark.text / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす Remark 300 BIOMOLECULE The following transformation matrices produce the lateral and longitudinal protomers in ... BIOMOLECULE The following transformation matrices produce the lateral and longitudinal protomers in the theoretical model of the F-actin filament described in the primary reference. LATERAL -0.9541 -0.2106 -0.2130 48.6183 -0.1870 0.9743 -0.1256 33.0936 0.2340 -0.0800 -0.9689 18.1290 LONGITUDINAL 0.8998 0.0128 0.4361 -8.5982 -0.0332 0.9987 0.0392 53.9677 -0.4350 -0.0497 0.8991 9.2909
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード CONTRACTILE PROTEIN /
CONTRACTILE PROTEIN /  Muscle protein (骨格筋) / Nucleotide-binding /
Muscle protein (骨格筋) / Nucleotide-binding /  Structural protein (タンパク質)
Structural protein (タンパク質) 機能・相同性情報
機能・相同性情報 tropomyosin binding /
tropomyosin binding /  myosin heavy chain binding / mesenchyme migration /
myosin heavy chain binding / mesenchyme migration /  troponin I binding / actin filament bundle / filamentous actin / actin filament bundle assembly / skeletal muscle thin filament assembly / striated muscle thin filament ...cytoskeletal motor activator activity /
troponin I binding / actin filament bundle / filamentous actin / actin filament bundle assembly / skeletal muscle thin filament assembly / striated muscle thin filament ...cytoskeletal motor activator activity /  tropomyosin binding /
tropomyosin binding /  myosin heavy chain binding / mesenchyme migration /
myosin heavy chain binding / mesenchyme migration /  troponin I binding / actin filament bundle / filamentous actin / actin filament bundle assembly / skeletal muscle thin filament assembly / striated muscle thin filament / skeletal muscle myofibril / actin monomer binding / skeletal muscle fiber development /
troponin I binding / actin filament bundle / filamentous actin / actin filament bundle assembly / skeletal muscle thin filament assembly / striated muscle thin filament / skeletal muscle myofibril / actin monomer binding / skeletal muscle fiber development /  stress fiber /
stress fiber /  titin binding / actin filament polymerization /
titin binding / actin filament polymerization /  filopodium /
filopodium /  マイクロフィラメント /
マイクロフィラメント /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / calcium-dependent protein binding /
加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / calcium-dependent protein binding /  lamellipodium /
lamellipodium /  cell body /
cell body /  hydrolase activity / protein domain specific binding /
hydrolase activity / protein domain specific binding /  calcium ion binding / positive regulation of gene expression / magnesium ion binding /
calcium ion binding / positive regulation of gene expression / magnesium ion binding /  ATP binding / identical protein binding /
ATP binding / identical protein binding /  細胞質
細胞質
 Oryctolagus cuniculus (ウサギ)
Oryctolagus cuniculus (ウサギ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.49 Å
分子置換 / 解像度: 2.49 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2005
ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2005 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2a5x.cif.gz
2a5x.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2a5x.ent.gz
pdb2a5x.ent.gz PDB形式
PDB形式 2a5x.json.gz
2a5x.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/a5/2a5x
https://data.pdbj.org/pub/pdb/validation_reports/a5/2a5x ftp://data.pdbj.org/pub/pdb/validation_reports/a5/2a5x
ftp://data.pdbj.org/pub/pdb/validation_reports/a5/2a5x
 リンク
リンク 集合体
集合体

 要素
要素 / Alpha-actin 1
/ Alpha-actin 1
 Oryctolagus cuniculus (ウサギ) / 参照: UniProt: P68135
Oryctolagus cuniculus (ウサギ) / 参照: UniProt: P68135

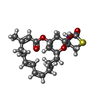
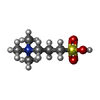







 Latrunculin
Latrunculin 2-Methyl-2,4-pentanediol
2-Methyl-2,4-pentanediol 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 8.2.2 / 波長: 1.1 / 波長: 1.1 Å
/ ビームライン: 8.2.2 / 波長: 1.1 / 波長: 1.1 Å : 1.1 Å / 相対比: 1
: 1.1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


















