登録情報 データベース : PDB / ID : 1qsvタイトル THE VEGF-BINDING DOMAIN OF FLT-1, 20 NMR STRUCTURES VASCULAR ENDOTHELIAL GROWTH FACTOR RECEPTOR 1 キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / データ登録者 Starovasnik, M.A. / Christinger, H.W. / Wiesmann, C. / Champe, M.A. / de Vos, A.M. / Skelton, N.J. ジャーナル : J.Mol.Biol. / 年 : 1999タイトル : Solution structure of the VEGF-binding domain of Flt-1: comparison of its free and bound states.著者 : Starovasnik, M.A. / Christinger, H.W. / Wiesmann, C. / Champe, M.A. / de Vos, A.M. / Skelton, N.J. 履歴 登録 1999年6月23日 登録サイト / 処理サイト 改定 1.0 1999年11月10日 Provider / タイプ 改定 1.1 2008年4月27日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2022年3月2日 Group / Database references / Derived calculationsカテゴリ database_2 / pdbx_nmr_software ... database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list Item / _database_2.pdbx_database_accession / _pdbx_nmr_software.name
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
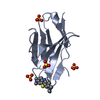
基本情報 要素
要素 血管内皮細胞増殖因子受容体
血管内皮細胞増殖因子受容体  キーワード
キーワード VEGF RECEPTOR (血管内皮細胞増殖因子受容体) / HORMONE-GROWTH FACTOR RECEPTOR COMPLEX
VEGF RECEPTOR (血管内皮細胞増殖因子受容体) / HORMONE-GROWTH FACTOR RECEPTOR COMPLEX 機能・相同性情報
機能・相同性情報 vascular endothelial growth factor receptor activity / embryonic morphogenesis / negative regulation of vascular endothelial cell proliferation / blood vessel morphogenesis /
vascular endothelial growth factor receptor activity / embryonic morphogenesis / negative regulation of vascular endothelial cell proliferation / blood vessel morphogenesis /  growth factor binding ...vascular endothelial growth factor receptor-1 signaling pathway / placental growth factor receptor activity / hyaloid vascular plexus regression / Neurophilin interactions with VEGF and VEGFR / VEGF binds to VEGFR leading to receptor dimerization /
growth factor binding ...vascular endothelial growth factor receptor-1 signaling pathway / placental growth factor receptor activity / hyaloid vascular plexus regression / Neurophilin interactions with VEGF and VEGFR / VEGF binds to VEGFR leading to receptor dimerization /  vascular endothelial growth factor receptor activity / embryonic morphogenesis / negative regulation of vascular endothelial cell proliferation / blood vessel morphogenesis /
vascular endothelial growth factor receptor activity / embryonic morphogenesis / negative regulation of vascular endothelial cell proliferation / blood vessel morphogenesis /  growth factor binding / monocyte chemotaxis / cellular response to vascular endothelial growth factor stimulus / vascular endothelial growth factor receptor signaling pathway / positive regulation of phospholipase C activity /
growth factor binding / monocyte chemotaxis / cellular response to vascular endothelial growth factor stimulus / vascular endothelial growth factor receptor signaling pathway / positive regulation of phospholipase C activity /  transmembrane receptor protein tyrosine kinase activity / positive regulation of MAP kinase activity /
transmembrane receptor protein tyrosine kinase activity / positive regulation of MAP kinase activity /  受容体型チロシンキナーゼ / peptidyl-tyrosine phosphorylation /
受容体型チロシンキナーゼ / peptidyl-tyrosine phosphorylation /  cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of angiogenesis /
cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of angiogenesis /  遊走 /
遊走 /  マイクロフィラメント /
マイクロフィラメント /  血管新生 / positive regulation of MAPK cascade / protein autophosphorylation /
血管新生 / positive regulation of MAPK cascade / protein autophosphorylation /  細胞分化 / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /
細胞分化 / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  receptor complex /
receptor complex /  エンドソーム / positive regulation of cell migration /
エンドソーム / positive regulation of cell migration /  focal adhesion / positive regulation of cell population proliferation /
focal adhesion / positive regulation of cell population proliferation /  extracellular space /
extracellular space /  ATP binding /
ATP binding /  細胞膜
細胞膜
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR / DISTANCE GEOMETRY, SIMULATED ANNEALING; MOLECULAR DYNAMICS
溶液NMR / DISTANCE GEOMETRY, SIMULATED ANNEALING; MOLECULAR DYNAMICS  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 1999
ジャーナル: J.Mol.Biol. / 年: 1999 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1qsv.cif.gz
1qsv.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1qsv.ent.gz
pdb1qsv.ent.gz PDB形式
PDB形式 1qsv.json.gz
1qsv.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/qs/1qsv
https://data.pdbj.org/pub/pdb/validation_reports/qs/1qsv ftp://data.pdbj.org/pub/pdb/validation_reports/qs/1qsv
ftp://data.pdbj.org/pub/pdb/validation_reports/qs/1qsv リンク
リンク 集合体
集合体
 要素
要素 血管内皮細胞増殖因子受容体 / FLT-1
血管内皮細胞増殖因子受容体 / FLT-1
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌) / 参照: UniProt: P17948
Escherichia coli (大腸菌) / 参照: UniProt: P17948 溶液NMR
溶液NMR 試料調製
試料調製
 解析
解析 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj






