+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8454 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy structure of a bovine CLC-K chloride channel, alternate (class 2) conformation | |||||||||
 マップデータ マップデータ | Structure of a bovine CLC-K chloride channel. Combined, unfiltered map of Class 2 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Stimuli-sensing channels / voltage-gated chloride channel activity / chloride transport /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Bos taurus (ウシ) / Bos taurus (ウシ) /   house mouse (ハツカネズミ) house mouse (ハツカネズミ) | |||||||||
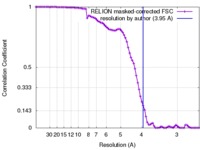
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.95 Å クライオ電子顕微鏡法 / 解像度: 3.95 Å | |||||||||
 データ登録者 データ登録者 | Park E / MacKinnon R | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Structure of a CLC chloride ion channel by cryo-electron microscopy. 著者: Eunyong Park / Ernest B Campbell / Roderick MacKinnon /  要旨: CLC proteins transport chloride (Cl) ions across cellular membranes to regulate muscle excitability, electrolyte movement across epithelia, and acidification of intracellular organelles. Some CLC ...CLC proteins transport chloride (Cl) ions across cellular membranes to regulate muscle excitability, electrolyte movement across epithelia, and acidification of intracellular organelles. Some CLC proteins are channels that conduct Cl ions passively, whereas others are secondary active transporters that exchange two Cl ions for one H. The structural basis underlying these distinctive transport mechanisms is puzzling because CLC channels and transporters are expected to share the same architecture on the basis of sequence homology. Here we determined the structure of a bovine CLC channel (CLC-K) using cryo-electron microscopy. A conserved loop in the Cl transport pathway shows a structure markedly different from that of CLC transporters. Consequently, the cytosolic constriction for Cl passage is widened in CLC-K such that the kinetic barrier previously postulated for Cl/H transporter function would be reduced. Thus, reduction of a kinetic barrier in CLC channels enables fast flow of Cl down its electrochemical gradient. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8454.map.gz emd_8454.map.gz | 39.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8454-v30.xml emd-8454-v30.xml emd-8454.xml emd-8454.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8454_fsc.xml emd_8454_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8454.png emd_8454.png | 59.6 KB | ||
| マスクデータ |  emd_8454_msk_1.map emd_8454_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_8454_additional_1.map.gz emd_8454_additional_1.map.gz emd_8454_additional_2.map.gz emd_8454_additional_2.map.gz | 49.3 MB 49.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8454 http://ftp.pdbj.org/pub/emdb/structures/EMD-8454 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8454 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8454 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8454.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8454.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of a bovine CLC-K chloride channel. Combined, unfiltered map of Class 2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_8454_msk_1.map emd_8454_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: B-factor-sharpend (-120 A^2) and lowpass-filtered (4.1 Ang)
| ファイル | emd_8454_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | B-factor-sharpend (-120 A^2) and lowpass-filtered (4.1 Ang) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: B-factor-sharpend (-120 A^2) and lowpass-filtered (3.9 Ang)
| ファイル | emd_8454_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | B-factor-sharpend (-120 A^2) and lowpass-filtered (3.9 Ang) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A bovine CLC-K channel complexed with a monoclonal antibody fragm...
| 全体 | 名称: A bovine CLC-K channel complexed with a monoclonal antibody fragment (Fab) |
|---|---|
| 要素 |
|
-超分子 #1: A bovine CLC-K channel complexed with a monoclonal antibody fragm...
| 超分子 | 名称: A bovine CLC-K channel complexed with a monoclonal antibody fragment (Fab) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Fab fragment generated by proteolytic (papain) cleavage of murine IgG antibody. |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換プラスミド: pFastBac (BacMam) Homo sapiens (ヒト) / 組換プラスミド: pFastBac (BacMam) |
-分子 #1: Chloride channel protein
| 分子 | 名称: Chloride channel protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
| 分子量 | 理論値: 73.521484 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRVRRGIRGG LDWLKRKLFC VGEDWYFLTV LGVLMALISF TMSFTVGRVV RAHKWLYREI GDSHLLRYLS WTVYPVALVS FSSGFSQSI TPFSGGSGIP ELKTILSGVV LEDYLDIKNF GAKAVGLTCT LASGSTIFLG KVGPFVHLSV MIAAYLGRVR A KATGESEN ...文字列: MRVRRGIRGG LDWLKRKLFC VGEDWYFLTV LGVLMALISF TMSFTVGRVV RAHKWLYREI GDSHLLRYLS WTVYPVALVS FSSGFSQSI TPFSGGSGIP ELKTILSGVV LEDYLDIKNF GAKAVGLTCT LASGSTIFLG KVGPFVHLSV MIAAYLGRVR A KATGESEN KSKRNEMLVA GAAVGVATVF AAPFSGVLFC IEVVSSHFSV WDYWRGFFAA TCGAFMFRLL AVFNSEQETI TS LYKTSFR VEVPFDLPEI FFFVALGAIC GVASCAYLFC QRKFLGFVKT NPVLSKLMAT SKPLYSALAA LVLASVTYPP GAG RFMASR LSMREYLDSL LDHNSWALLT RQASPPWPVE PDPQNLWFEW YHPQFTIFGT LAFFLVMKFW MLILATTIPM PAGY FMPIF IFGAAIGRLL GEALSVAFPE GIVAGGVTNP IMPGGYALAG AAAFSGAVTH SISTALLAFE LTGQIVHALP VLMAV LAAN AIAQSCQPSF YDGTIIVKKL PYLPWIRGRK ISSHRVTVEH FMNRAITTLA KDTPQEEVVK VVTSTDMAEY PLVAST ESQ TLVGTMRRAQ LVQALQAEPP SWAPGQQRCL QDILAEGCPV EPVTLKLSPE TSLHQAHNLF ELLNLQSLFV TSQGRAV GF VSWVELEKAI SKLTNPPAPK SNSLEVLFQ |
-分子 #2: Monoclonal antibody, Fab fragment, heavy chain
| 分子 | 名称: Monoclonal antibody, Fab fragment, heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   house mouse (ハツカネズミ) house mouse (ハツカネズミ) |
| 分子量 | 理論値: 12.633068 KDa |
| 配列 | 文字列: DVQLQESGPG LVKPSQSLSL TCTVTGDSVT SDYAWSWIRQ FPGKKLEWMG YITYSGNTIY NPSLKSRISI TRDTSKNQFF LQLKSVIIE DTATYYCSRG VDYWGQGTSV TVSS |
-分子 #3: Monoclonal antibody, Fab fragment, light chain
| 分子 | 名称: Monoclonal antibody, Fab fragment, light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   house mouse (ハツカネズミ) house mouse (ハツカネズミ) |
| 分子量 | 理論値: 11.824102 KDa |
| 配列 | 文字列: DIVMTQSPKF MSTSVGDRVS VTCKASQNVG TNVAWYQQKP GQSPKTLIYW ASYRYSGVPD RFTGSGSGTD FTLAISNVQS EDLAEYFCQ QYNSYPLTFG SGTKLELK |
-分子 #4: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 0.3 sec. / 平均電子線量: 1.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X