+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7136 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
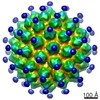
| タイトル | High-Resolution Structure Analysis of Antibody V5 and U4 Conformational Epitope on Human Papillomavirus 16 | |||||||||
 マップデータ マップデータ | Antibody V5 and U4 Conformational Epitope on Human Papillomavirus 16 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=7 icosahedral viral capsid / endocytosis involved in viral entry into host cell / host cell nucleus / virion attachment to host cell / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) / Mus musculus (ハツカネズミ) /   Mouse (ネズミ) / Mouse (ネズミ) /   Human papillomavirus type 16 (パピローマウイルス) Human papillomavirus type 16 (パピローマウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.7 Å クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Guan J / Bywaters SM / Brendle SA / Ashley RE / Makhov AM / Conway JF / Christenson ND / Hafenstein S | |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2017 ジャーナル: Viruses / 年: 2017タイトル: High-Resolution Structure Analysis of Antibody V5 and U4 Conformational Epitopes on Human Papillomavirus 16. 著者: Jian Guan / Stephanie M Bywaters / Sarah A Brendle / Robert E Ashley / Alexander M Makhov / James F Conway / Neil D Christensen / Susan Hafenstein /  要旨: Cancers attributable to human papillomavirus (HPV) place a huge burden on the health of both men and women. The current commercial vaccines are genotype specific and provide little therapeutic ...Cancers attributable to human papillomavirus (HPV) place a huge burden on the health of both men and women. The current commercial vaccines are genotype specific and provide little therapeutic benefit to patients with existing HPV infections. Identifying the conformational epitopes on the virus capsid supports the development of improved recombinant vaccines to maximize long-term protection against multiple types of HPV. Fragments of antibody (Fab) digested from the neutralizing monoclonal antibodies H16.V5 (V5) and H16.U4 (U4) were bound to HPV16 capsids and the structures of the two virus-Fab complexes were solved to near atomic resolution using cryo-electron microscopy. The structures reveal virus conformational changes, the Fab-binding mode to the capsid, the residues comprising the epitope and indicate a potential interaction of U4 with the minor structural protein, L2. Competition enzyme-linked immunosorbent assay (ELISA) showed V5 outcompetes U4 when added sequentially, demonstrating a steric interference even though the footprints do not overlap. Combined with our previously reported immunological and structural results, we propose that the virus may initiate host entry through an interaction between the icosahedral five-fold vertex of the capsid and receptors on the host cell. The highly detailed epitopes identified for the two antibodies provide a framework for continuing biochemical, genetic and biophysical studies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7136.map.gz emd_7136.map.gz | 1.1 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7136-v30.xml emd-7136-v30.xml emd-7136.xml emd-7136.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7136.png emd_7136.png | 333.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7136 http://ftp.pdbj.org/pub/emdb/structures/EMD-7136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7136 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7136.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7136.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Antibody V5 and U4 Conformational Epitope on Human Papillomavirus 16 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.147 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human papillomavirus type 16 / Antibody complex
| 全体 | 名称: Human papillomavirus type 16 / Antibody complex |
|---|---|
| 要素 |
|
-超分子 #1: Human papillomavirus type 16 / Antibody complex
| 超分子 | 名称: Human papillomavirus type 16 / Antibody complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Antibody U4
| 超分子 | 名称: Antibody U4 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-超分子 #3: Human papillomavirus type 16
| 超分子 | 名称: Human papillomavirus type 16 / タイプ: virus / ID: 3 / 親要素: 1 / 含まれる分子: #3 / NCBI-ID: 333760 / 生物種: Human papillomavirus type 16 / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROCOMPLEX / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: U4 Heavy chain
| 分子 | 名称: U4 Heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mouse (ネズミ) Mouse (ネズミ) |
| 分子量 | 理論値: 12.108347 KDa |
| 配列 | 文字列: SGGGLVKPGG SLKLSCEASG FTFSSYAMSW VRQTPEKRLE WVASISSGGN THYPDSVKGR FTISRDNARN ILYLQMSSLR SEDTAMYYC ARGLYYGYDE GSDFDYWGQG |
-分子 #2: U4 Light chain
| 分子 | 名称: U4 Light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mouse (ネズミ) Mouse (ネズミ) |
| 分子量 | 理論値: 12.09953 KDa |
| 配列 | 文字列: DIVMSQSPSS LAVSVGEKVT MSCKSSQSLL YSNTQKNYLA WYQQKPGQSP KLLIYWASTR ESGVPDRFTG SGSGTDFTLT ISSVKAEDL AVYYCQQYYS YPLTFGAGTK L |
-分子 #3: Major capsid protein L1
| 分子 | 名称: Major capsid protein L1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human papillomavirus type 16 (パピローマウイルス) Human papillomavirus type 16 (パピローマウイルス) |
| 分子量 | 理論値: 52.387277 KDa |
| 配列 | 文字列: YLPPVPVSKV VSTDEYVART NIYYHAGTSR LLAVGHPYFP IKKPNNNKIL VPKVSGLQYR VFRIHLPDPN KFGFPDTSFY NPDTQRLVW ACVGVEVGRG QPLGVGISGH PLLNKLDDTE NASAYAANAG VDNRECISMD YKQTQLCLIG CKPPIGEHWG K GSPCTNVA ...文字列: YLPPVPVSKV VSTDEYVART NIYYHAGTSR LLAVGHPYFP IKKPNNNKIL VPKVSGLQYR VFRIHLPDPN KFGFPDTSFY NPDTQRLVW ACVGVEVGRG QPLGVGISGH PLLNKLDDTE NASAYAANAG VDNRECISMD YKQTQLCLIG CKPPIGEHWG K GSPCTNVA VNPGDCPPLE LINTVIQDGD MVDTGFGAMD FTTLQANKSE VPLDICTSIC KYPDYIKMVS EPYGDSLFFY LR REQMFVR HLFNRAGAVG ENVPDDLYIK GSGSTANLAS SNYFPTPSGS MVTSDAQIFN KPYWLQRAQG HNNGICWGNQ LFV TVVDTT RSTNMSLCAA ISTSETTYKN TNFKEYLRHG EEYDLQFIFQ LCKITLTADV MTYIHSMNST ILEDWNFGLQ PPPG GTLED TYRFVTSQAI ACQKHTPPAP KEDPLKKYTF WEVNLKEKFS ADLDQFPLGR KFLLQAGLKA KPKF |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: FEI FALCON II (4k x 4k) #0 - 平均電子線量: 7.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: FEI FALCON II (4k x 4k) #1 - 平均電子線量: 7.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Auto3dem RMC |
|---|---|
| 初期 角度割当 | タイプ: COMMON LINE |
| 最終 角度割当 | タイプ: COMMON LINE |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 17612 |
| Image recording ID | 1 |
 ムービー
ムービー コントローラー
コントローラー