[日本語] English
 万見
万見- EMDB-7006: Human ribonucleotide reductase large subunit (alpha) with dATP and CDP -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7006 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human ribonucleotide reductase large subunit (alpha) with dATP and CDP リボヌクレオシド二リン酸レダクターゼ リボヌクレオシド二リン酸レダクターゼ | ||||||||||||
 マップデータ マップデータ | Human ribonucleotide reductase large subunit (alpha) with dATP and CDP リボヌクレオシド二リン酸レダクターゼ リボヌクレオシド二リン酸レダクターゼ | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Ribonucleotide Reductase Electron transfer Radical chemistry Thiyl radical /  OXIDOREDUCTASE (酸化還元酵素) OXIDOREDUCTASE (酸化還元酵素) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 ribonucleoside-diphosphate reductase activity / pyrimidine nucleobase metabolic process / ribonucleoside-diphosphate reductase activity / pyrimidine nucleobase metabolic process /  cell proliferation in forebrain / positive regulation of G0 to G1 transition / mitochondrial DNA replication / ribonucleoside diphosphate metabolic process / 2'-deoxyribonucleotide biosynthetic process / cell proliferation in forebrain / positive regulation of G0 to G1 transition / mitochondrial DNA replication / ribonucleoside diphosphate metabolic process / 2'-deoxyribonucleotide biosynthetic process /  ribonucleoside-diphosphate reductase complex / ribonucleoside-diphosphate reductase complex /  リボヌクレオシド二リン酸レダクターゼ / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor ... リボヌクレオシド二リン酸レダクターゼ / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor ... ribonucleoside-diphosphate reductase activity / pyrimidine nucleobase metabolic process / ribonucleoside-diphosphate reductase activity / pyrimidine nucleobase metabolic process /  cell proliferation in forebrain / positive regulation of G0 to G1 transition / mitochondrial DNA replication / ribonucleoside diphosphate metabolic process / 2'-deoxyribonucleotide biosynthetic process / cell proliferation in forebrain / positive regulation of G0 to G1 transition / mitochondrial DNA replication / ribonucleoside diphosphate metabolic process / 2'-deoxyribonucleotide biosynthetic process /  ribonucleoside-diphosphate reductase complex / ribonucleoside-diphosphate reductase complex /  リボヌクレオシド二リン酸レダクターゼ / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor / Interconversion of nucleotide di- and triphosphates / deoxyribonucleotide biosynthetic process / protein heterotetramerization / response to ionizing radiation / DNA synthesis involved in DNA repair / positive regulation of G1/S transition of mitotic cell cycle / positive regulation of G2/M transition of mitotic cell cycle / cell projection / male gonad development / disordered domain specific binding / retina development in camera-type eye / リボヌクレオシド二リン酸レダクターゼ / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor / Interconversion of nucleotide di- and triphosphates / deoxyribonucleotide biosynthetic process / protein heterotetramerization / response to ionizing radiation / DNA synthesis involved in DNA repair / positive regulation of G1/S transition of mitotic cell cycle / positive regulation of G2/M transition of mitotic cell cycle / cell projection / male gonad development / disordered domain specific binding / retina development in camera-type eye /  核膜 / 核膜 /  DNA修復 / neuronal cell body / DNA修復 / neuronal cell body /  ミトコンドリア / ミトコンドリア /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Brignole EJ / Drennan CL | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: 3.3-Å resolution cryo-EM structure of human ribonucleotide reductase with substrate and allosteric regulators bound. 著者: Edward J Brignole / Kuang-Lei Tsai / Johnathan Chittuluru / Haoran Li / Yimon Aye / Pawel A Penczek / JoAnne Stubbe / Catherine L Drennan / Francisco Asturias /  要旨: Ribonucleotide reductases (RNRs) convert ribonucleotides into deoxyribonucleotides, a reaction essential for DNA replication and repair. Human RNR requires two subunits for activity, the α subunit ...Ribonucleotide reductases (RNRs) convert ribonucleotides into deoxyribonucleotides, a reaction essential for DNA replication and repair. Human RNR requires two subunits for activity, the α subunit contains the active site, and the β subunit houses the radical cofactor. Here, we present a 3.3-Å resolution structure by cryo-electron microscopy (EM) of a dATP-inhibited state of human RNR. This structure, which was determined in the presence of substrate CDP and allosteric regulators ATP and dATP, has three α units arranged in an α ring. At near-atomic resolution, these data provide insight into the molecular basis for CDP recognition by allosteric specificity effectors dATP/ATP. Additionally, we present lower-resolution EM structures of human α in the presence of both the anticancer drug clofarabine triphosphate and β. Together, these structures support a model for RNR inhibition in which β is excluded from binding in a radical transfer competent position when α exists as a stable hexamer. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7006.map.gz emd_7006.map.gz | 6.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7006-v30.xml emd-7006-v30.xml emd-7006.xml emd-7006.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7006.png emd_7006.png | 189.3 KB | ||
| Filedesc metadata |  emd-7006.cif.gz emd-7006.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7006 http://ftp.pdbj.org/pub/emdb/structures/EMD-7006 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7006 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7006 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7006.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7006.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human ribonucleotide reductase large subunit (alpha) with dATP and CDP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human ribonucleotide reductase large subnunit (alpha)
| 全体 | 名称: Human ribonucleotide reductase large subnunit (alpha) |
|---|---|
| 要素 |
|
-超分子 #1: Human ribonucleotide reductase large subnunit (alpha)
| 超分子 | 名称: Human ribonucleotide reductase large subnunit (alpha) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Ribonucleoside-diphosphate reductase large subunit
| 分子 | 名称: Ribonucleoside-diphosphate reductase large subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号:  リボヌクレオシド二リン酸レダクターゼ リボヌクレオシド二リン酸レダクターゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 92.350391 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MHVIKRDGRQ ERVMFDKITS RIQKLCYGLN MDFVDPAQIT MKVIQGLYSG VTTVELDTLA AETAATLTT KHPDYAILAA RIAVSNLHKE TKKVFSDVME DLYNYINPHN GKHSPMVAKS TLDIVLANKD RLNSAIIYDR D FSYNYFGF ...文字列: MGSSHHHHHH SSGLVPRGSH MHVIKRDGRQ ERVMFDKITS RIQKLCYGLN MDFVDPAQIT MKVIQGLYSG VTTVELDTLA AETAATLTT KHPDYAILAA RIAVSNLHKE TKKVFSDVME DLYNYINPHN GKHSPMVAKS TLDIVLANKD RLNSAIIYDR D FSYNYFGF KTLERSYLLK INGKVAERPQ HMLMRVSVGI HKEDIDAAIE TYNLLSERWF THASPTLFNA GTNRPQLSSC FL LSMKDDS IEGIYDTLKQ CALISKSAGG IGVAVSCIRA TGSYIAGTNG NSNGLVPMLR VYNNTARYVD QGGNKRPGAF AIY LEPWHL DIFEFLDLKK NTGKEEQRAR DLFFALWIPD LFMKRVETNQ DWSLMCPNEC PGLDEVWGEE FEKLYASYEK QGRV RKVVK AQQLWYAIIE SQTETGTPYM LYKDSCNRKS NQQNLGTIKC SNLCTEIVEY TSKDEVAVCN LASLALNMYV TSEHT YDFK KLAEVTKVVV RNLNKIIDIN YYPVPEACLS NKRHRPIGIG VQGLADAFIL MRYPFESAEA QLLNKQIFET IYYGAL EAS CDLAKEQGPY ETYEGSPVSK GILQYDMWNV TPTDLWDWKV LKEKIAKYGI RNSLLIAPMP TASTAQILGN NESIEPY TS NIYTRRVLSG EFQIVNPHLL KDLTERGLWH EEMKNQIIAC NGSIQSIPEI PDDLKQLYKT VWEISQKTVL KMAAERGA F IDQSQSLNIH IAEPNYGKLT SMHFYGWKQG LKTGMYYLRT RPAANPIQFT LNKEKLKDKE KVSKEEEEKE RNTAAMVCS LENRDECLMC GS UniProtKB:  Ribonucleoside-diphosphate reductase large subunit Ribonucleoside-diphosphate reductase large subunit |
-分子 #2: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE
| 分子 | 名称: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 12 / 式: DTP |
|---|---|
| 分子量 | 理論値: 491.182 Da |
| Chemical component information |  ChemComp-DTP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 12 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: CYTIDINE-5'-DIPHOSPHATE
| 分子 | 名称: CYTIDINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 6 / 式: CDP |
|---|---|
| 分子量 | 理論値: 403.176 Da |
| Chemical component information | 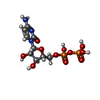 ChemComp-CDP: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 36 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| |||||||||||||||
| グリッド | モデル: Protochips C-Flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER 詳細: glow discharged at 20 mA in an EMITech K100X Grid was first cleaned in a Solarus 950 (Gatan) at 25 W for 10 s in 75/25 Ar/O2 gas mixture | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 75 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER 詳細: manual blot with a strip of Whatman paper until drop stops wicking determined visually. | |||||||||||||||
| 詳細 | 14 microM alpha and 0.05 mM dATP, 3 mM ATP, 1 mM CDP in 50 mM HEPES, pH 7.6, 15 mM MgCl2, 1 mM EDTA, 5 mM DTT, and 50 mM KCl |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 5-38 / 実像数: 2144 / 平均露光時間: 7.6 sec. / 平均電子線量: 44.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL / In silico モデル: Calculated ab initio |
|---|---|
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: SPARX |
| 最終 再構成 | 想定した対称性 - 点群: D3 (2回x3回 2面回転対称 ) )解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: SPARX / 使用した粒子像数: 43885 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6aui: |
 ムービー
ムービー コントローラー
コントローラー









