+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5798 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Ribosome with Elongation Factor G Trapped in the Pre-Translocation State | |||||||||
 マップデータ マップデータ | Reconstruction of a ribosome bound to EF-G and P/E site tRNA. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  protein structure (タンパク質構造) / protein structure (タンパク質構造) /  translation (翻訳 (生物学)) / translation (翻訳 (生物学)) /  EF-G (EF-G) / EF-G (EF-G) /  electron cryo-microscopy (低温電子顕微鏡法) / electron cryo-microscopy (低温電子顕微鏡法) /  single particle analysis (単粒子解析法) single particle analysis (単粒子解析法) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellular anatomical structure / ribosome disassembly /  guanosine tetraphosphate binding / translational elongation / guanosine tetraphosphate binding / translational elongation /  translation elongation factor activity / translation elongation factor activity /  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 /  GTPase activity / GTP binding / GTPase activity / GTP binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / synthetic construct (人工物) / unidentified (未定義) Escherichia coli (大腸菌) / synthetic construct (人工物) / unidentified (未定義) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.3 Å クライオ電子顕微鏡法 / 解像度: 7.3 Å | |||||||||
 データ登録者 データ登録者 | Brilot AF / Korostelev AA / Ermolenko DN / Grigorieff N | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2013 ジャーナル: Proc Natl Acad Sci U S A / 年: 2013タイトル: Structure of the ribosome with elongation factor G trapped in the pretranslocation state. 著者: Axel F Brilot / Andrei A Korostelev / Dmitri N Ermolenko / Nikolaus Grigorieff /  要旨: During protein synthesis, tRNAs and their associated mRNA codons move sequentially on the ribosome from the A (aminoacyl) site to the P (peptidyl) site to the E (exit) site in a process catalyzed by ...During protein synthesis, tRNAs and their associated mRNA codons move sequentially on the ribosome from the A (aminoacyl) site to the P (peptidyl) site to the E (exit) site in a process catalyzed by a universally conserved ribosome-dependent GTPase [elongation factor G (EF-G) in prokaryotes and elongation factor 2 (EF-2) in eukaryotes]. Although the high-resolution structure of EF-G bound to the posttranslocation ribosome has been determined, the pretranslocation conformation of the ribosome bound with EF-G and A-site tRNA has evaded visualization owing to the transient nature of this state. Here we use electron cryomicroscopy to determine the structure of the 70S ribosome with EF-G, which is trapped in the pretranslocation state using antibiotic viomycin. Comparison with the posttranslocation ribosome shows that the small subunit of the pretranslocation ribosome is rotated by ∼12° relative to the large subunit. Domain IV of EF-G is positioned in the cleft between the body and head of the small subunit outwardly of the A site and contacts the A-site tRNA. Our findings suggest a model in which domain IV of EF-G promotes the translocation of tRNA from the A to the P site as the small ribosome subunit spontaneously rotates back from the hybrid, rotated state into the nonrotated posttranslocation state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5798.map.gz emd_5798.map.gz | 105.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5798-v30.xml emd-5798-v30.xml emd-5798.xml emd-5798.xml | 14.6 KB 14.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5798_1.jpg emd_5798_1.jpg emd_5798_2.png emd_5798_2.png | 220.9 KB 224.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5798 http://ftp.pdbj.org/pub/emdb/structures/EMD-5798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5798 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5798.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5798.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of a ribosome bound to EF-G and P/E site tRNA. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ribosome bound to EF-G and P/E tRNA
| 全体 | 名称: Ribosome bound to EF-G and P/E tRNA |
|---|---|
| 要素 |
|
-超分子 #1000: Ribosome bound to EF-G and P/E tRNA
| 超分子 | 名称: Ribosome bound to EF-G and P/E tRNA / タイプ: sample / ID: 1000 / Number unique components: 6 |
|---|---|
| 分子量 | 理論値: 3 MDa |
-超分子 #1: 70S ribosome
| 超分子 | 名称: 70S ribosome / タイプ: complex / ID: 1 / 組換発現: No / データベース: NCBI / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: MRE600 Escherichia coli (大腸菌) / 株: MRE600 |
| 分子量 | 理論値: 3 MDa |
-分子 #1: Elongation Factor G
| 分子 | 名称: Elongation Factor G / タイプ: protein_or_peptide / ID: 1 / Name.synonym: EF-G / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K-12 Escherichia coli (大腸菌) / 株: K-12 |
| 分子量 | 理論値: 78 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: K-12 Escherichia coli (大腸菌) / 組換株: K-12 |
| 配列 | UniProtKB:  EF-G EF-GGO: translational elongation, GTP binding,  translation elongation factor activity, intracellular anatomical structure translation elongation factor activity, intracellular anatomical structureInterPro: Translation elongation factor EFG/EF2 |
-分子 #2: Transfer RNA
| 分子 | 名称: Transfer RNA / タイプ: rna / ID: 2 / Name.synonym: tRNA / 分類: TRANSFER / Structure: DOUBLE HELIX / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K-12 Escherichia coli (大腸菌) / 株: K-12 |
| 分子量 | 実験値: 25 KDa |
-分子 #3: Messenger RNA
| 分子 | 名称: Messenger RNA / タイプ: rna / ID: 3 / Name.synonym: mRNA / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12 KDa |
| 配列 | 文字列: GGCAAGGAGG UAAAAAUGUU UAAACGUAAA UCUACU |
-分子 #4: Fusidic Acid
| 分子 | 名称: Fusidic Acid / タイプ: ligand / ID: 4 / Name.synonym: Fus / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 1 KDa |
| Chemical component information | 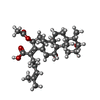 ChemComp-FUA: |
-分子 #5: Viomycin
| 分子 | 名称: Viomycin / タイプ: ligand / ID: 5 / Name.synonym: Vio / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 1 KDa |
| Chemical component information | 
ChemComp-PRD_000226: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 10 mM HEPES-KOH, 5 mM MgCl2, 90 mM NH4Cl, 2 mM spermidine, 0.1 mM spermine, 6 mM BME, 0.5 mM viomycin, 0.5 mM GTP, 0.5 mM fusidic acid |
| グリッド | 詳細: C-flat 1.2/1.3 holey carbon 400 mesh copper grid, glow discharged with a current of -20 mA for 45 seconds in an EMITECH K100X glow discharge unit |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK II 手法: Freshly glow-disharged grids were loaded into an FEI Mark II Vitrobot and equilibrated to 95% relative humidity at 22 degrees Celsius. 2 microliters of sample was applied through the side ...手法: Freshly glow-disharged grids were loaded into an FEI Mark II Vitrobot and equilibrated to 95% relative humidity at 22 degrees Celsius. 2 microliters of sample was applied through the side port, blotted for 7 seconds with a positional offset of 2, and plunged into liquid ethane. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 134615 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 6.95 µm / 最小 デフォーカス(公称値): 1.15 µm / 倍率(公称値): 133333 Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 6.95 µm / 最小 デフォーカス(公称値): 1.15 µm / 倍率(公称値): 133333 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| アライメント法 | Legacy - 非点収差: Automatically corrected using FEI software |
| 日付 | 2012年11月2日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON I (4k x 4k) デジタル化 - サンプリング間隔: 14.0 µm / 実像数: 13341 / 平均電子線量: 30 e/Å2 / ビット/ピクセル: 16 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: CTFFIND3, FREALIGN per micrograph |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.3 Å / 解像度の算出法: OTHER ソフトウェア - 名称: EMAN2, IMAGIC, FREALIGN, RSAMPLE, CTFFIND3 詳細: Refinement included data to 12 Angstrom resolution to limit FSC bias. See primary citation Supplementary Information for details. 使用した粒子像数: 1341961 |
| 詳細 | Refinement and 3D classification performed by Frealign. See primary citation Supplementary Information for details. |
 ムービー
ムービー コントローラー
コントローラー
























