+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5605 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
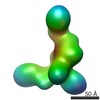
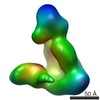
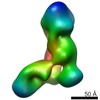
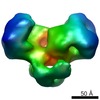
| タイトル | Substrate-specific structural rearrangements of human Dicer | |||||||||
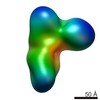
 マップデータ マップデータ | Negative stain EM reconstruction of Dicer-PACT in the canonical conformation | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA-mediated gene silencing / pre-miRNA processing / RNaseIII | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 peripheral nervous system myelin formation / regulation of regulatory ncRNA processing / tRNA-derived small RNA (tsRNA or tRNA-related fragment, tRF) biogenesis / global gene silencing by mRNA cleavage / tRNA decay / pre-miRNA binding / Small interfering RNA (siRNA) biogenesis / negative regulation of Schwann cell proliferation / positive regulation of myelination / peripheral nervous system myelin formation / regulation of regulatory ncRNA processing / tRNA-derived small RNA (tsRNA or tRNA-related fragment, tRF) biogenesis / global gene silencing by mRNA cleavage / tRNA decay / pre-miRNA binding / Small interfering RNA (siRNA) biogenesis / negative regulation of Schwann cell proliferation / positive regulation of myelination /  ribonuclease III ... ribonuclease III ... peripheral nervous system myelin formation / regulation of regulatory ncRNA processing / tRNA-derived small RNA (tsRNA or tRNA-related fragment, tRF) biogenesis / global gene silencing by mRNA cleavage / tRNA decay / pre-miRNA binding / Small interfering RNA (siRNA) biogenesis / negative regulation of Schwann cell proliferation / positive regulation of myelination / peripheral nervous system myelin formation / regulation of regulatory ncRNA processing / tRNA-derived small RNA (tsRNA or tRNA-related fragment, tRF) biogenesis / global gene silencing by mRNA cleavage / tRNA decay / pre-miRNA binding / Small interfering RNA (siRNA) biogenesis / negative regulation of Schwann cell proliferation / positive regulation of myelination /  ribonuclease III / ribonuclease III /  deoxyribonuclease I activity / deoxyribonuclease I activity /  apoptotic DNA fragmentation / miRNA metabolic process / nerve development / RISC-loading complex / positive regulation of Schwann cell differentiation / RISC complex assembly / miRNA processing / pre-miRNA processing / apoptotic DNA fragmentation / miRNA metabolic process / nerve development / RISC-loading complex / positive regulation of Schwann cell differentiation / RISC complex assembly / miRNA processing / pre-miRNA processing /  ribonuclease III activity / siRNA processing / siRNA binding / M-decay: degradation of maternal mRNAs by maternally stored factors / RISC complex / outer ear morphogenesis / middle ear morphogenesis / ribonuclease III activity / siRNA processing / siRNA binding / M-decay: degradation of maternal mRNAs by maternally stored factors / RISC complex / outer ear morphogenesis / middle ear morphogenesis /  skeletal system morphogenesis / MicroRNA (miRNA) biogenesis / negative regulation of tumor necrosis factor production / skeletal system morphogenesis / MicroRNA (miRNA) biogenesis / negative regulation of tumor necrosis factor production /  enzyme activator activity / negative regulation of tumor necrosis factor-mediated signaling pathway / positive regulation of intrinsic apoptotic signaling pathway / RNA endonuclease activity / neuron projection morphogenesis / enzyme activator activity / negative regulation of tumor necrosis factor-mediated signaling pathway / positive regulation of intrinsic apoptotic signaling pathway / RNA endonuclease activity / neuron projection morphogenesis /  helicase activity / response to virus / PKR-mediated signaling / helicase activity / response to virus / PKR-mediated signaling /  double-stranded RNA binding / cellular response to oxidative stress / protein stabilization / double-stranded RNA binding / cellular response to oxidative stress / protein stabilization /  免疫応答 / protein domain specific binding / negative regulation of cell population proliferation / negative regulation of gene expression / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / 免疫応答 / protein domain specific binding / negative regulation of cell population proliferation / negative regulation of gene expression / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II /  enzyme binding / protein homodimerization activity / enzyme binding / protein homodimerization activity /  DNA binding / DNA binding /  RNA binding / extracellular exosome / RNA binding / extracellular exosome /  核質 / 核質 /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 26.0 Å ネガティブ染色法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Taylor DW / Ma E / Shigematsu H / Cianfrocco MA / Noland CL / Nagayama K / Nogales E / Doudna JA / Wang HW | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2013 ジャーナル: Nat Struct Mol Biol / 年: 2013タイトル: Substrate-specific structural rearrangements of human Dicer. 著者: David W Taylor / Enbo Ma / Hideki Shigematsu / Michael A Cianfrocco / Cameron L Noland / Kuniaki Nagayama / Eva Nogales / Jennifer A Doudna / Hong-Wei Wang /  要旨: Dicer has a central role in RNA-interference pathways by cleaving double-stranded RNAs (dsRNAs) to produce small regulatory RNAs. Human Dicer can process long double-stranded and hairpin precursor ...Dicer has a central role in RNA-interference pathways by cleaving double-stranded RNAs (dsRNAs) to produce small regulatory RNAs. Human Dicer can process long double-stranded and hairpin precursor RNAs to yield short interfering RNAs (siRNAs) and microRNAs (miRNAs), respectively. Previous studies have shown that pre-miRNAs are cleaved more rapidly than pre-siRNAs in vitro and are the predominant natural Dicer substrates. We have used EM and single-particle analysis of Dicer-RNA complexes to gain insight into the structural basis for human Dicer's substrate preference. Our studies show that Dicer traps pre-siRNAs in a nonproductive conformation, whereas interactions of Dicer with pre-miRNAs and dsRNA-binding proteins induce structural changes in the enzyme that enable productive substrate recognition in the central catalytic channel. These findings implicate RNA structure and cofactors in determining substrate recognition and processing efficiency by human Dicer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5605.map.gz emd_5605.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5605-v30.xml emd-5605-v30.xml emd-5605.xml emd-5605.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5605.png emd_5605.png | 47.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5605 http://ftp.pdbj.org/pub/emdb/structures/EMD-5605 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5605 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5605 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5605.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5605.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain EM reconstruction of Dicer-PACT in the canonical conformation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human Dicer-PACT heterodimer in canonical conformation
| 全体 | 名称: Human Dicer-PACT heterodimer in canonical conformation |
|---|---|
| 要素 |
|
-超分子 #1000: Human Dicer-PACT heterodimer in canonical conformation
| 超分子 | 名称: Human Dicer-PACT heterodimer in canonical conformation タイプ: sample / ID: 1000 / 集合状態: monomer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 260 KDa |
-分子 #1: Endoribonuclease Dicer
| 分子 | 名称: Endoribonuclease Dicer / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Dicer, Helicase with RNase motif / コピー数: 1 / 集合状態: monomer / 組換発現: Yes / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 220 KDa |
| 配列 | UniProtKB:  Dicer / GO: pre-miRNA processing Dicer / GO: pre-miRNA processingInterPro: Ribonuclease III domain, PAZ domain, Helicase, C-terminal, Helicase superfamily 1/2, ATP-binding domain, Double-stranded RNA-binding domain, INTERPRO: IPR001159, DEAD/DEAH box helicase ...InterPro:  Ribonuclease III domain, PAZ domain, Ribonuclease III domain, PAZ domain,  Helicase, C-terminal, Helicase superfamily 1/2, ATP-binding domain, Double-stranded RNA-binding domain, INTERPRO: IPR001159, Helicase, C-terminal, Helicase superfamily 1/2, ATP-binding domain, Double-stranded RNA-binding domain, INTERPRO: IPR001159,  DEAD/DEAH box helicase domain, Dicer dimerisation domain DEAD/DEAH box helicase domain, Dicer dimerisation domain |
-分子 #2: Interferon-inducible double stranded RNA-dependent protein kinase...
| 分子 | 名称: Interferon-inducible double stranded RNA-dependent protein kinase activator A タイプ: protein_or_peptide / ID: 2 / Name.synonym: PACT / コピー数: 1 / 集合状態: monomer / 組換発現: Yes / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 34 KDa |
| 配列 | UniProtKB: Interferon-inducible double-stranded RNA-dependent protein kinase activator A GO:  double-stranded RNA binding double-stranded RNA bindingInterPro:  INTERPRO: IPR001159, Double-stranded RNA-binding domain INTERPRO: IPR001159, Double-stranded RNA-binding domain |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 20 mM HEPES, pH 7.5, 150 mM KCl, 3 mM EDTA, 1 mM DTT, and 2.5% glycerol |
| 染色 | タイプ: NEGATIVE 詳細: After adsorption for 1 min, we stained the samples consecutively with three droplets of 2% (w/v) uranyl formate solution, blotted off the residual stain and air-dried the sample in a hood. |
| グリッド | 詳細: glow-discharged holey carbon grids with a thin layer of carbon over the holes |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 5.2 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 5.2 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100,000 times magnification |
| 日付 | 2012年7月10日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC TVIPS (4k x 4k) 実像数: 400 / 平均電子線量: 20 e/Å2 |
- 画像解析
画像解析
| CTF補正 | 詳細: each micrograph |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 26.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN2/SPARX, multi-model / 使用した粒子像数: 10000 |
 ムービー
ムービー コントローラー
コントローラー