+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5174 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of 70S ribosome in the 100S ribosome in the hibernation stage | |||||||||
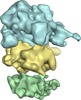
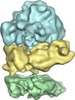
 マップデータ マップデータ | The density map of 70S ribosome with part of its partner in the 100S ribosome in hibernation stage. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cryoelectron microscopy (低温電子顕微鏡法) / cryoelectron microscopy (低温電子顕微鏡法) /  ribosomal protein ribosomal protein | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 18.0 Å クライオ電子顕微鏡法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Kato T / Yoshida H / Miyata T / Maki Y / Wada A / Namba K | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2010 ジャーナル: Structure / 年: 2010タイトル: Structure of the 100S ribosome in the hibernation stage revealed by electron cryomicroscopy. 著者: Takayuki Kato / Hideji Yoshida / Tomoko Miyata / Yasushi Maki / Akira Wada / Keiichi Namba /  要旨: In the stationary growth phase of bacteria, protein biosynthesis on ribosomes is suppressed, and the ribosomes are preserved in the cell by the formation of the 100S ribosome. The 100S ribosome is a ...In the stationary growth phase of bacteria, protein biosynthesis on ribosomes is suppressed, and the ribosomes are preserved in the cell by the formation of the 100S ribosome. The 100S ribosome is a dimer of the 70S ribosome and is formed by the binding of the ribosome modulation factor and the hibernation promoting factor. However, the binding mode between the two 70S ribosomes and the mechanism of complex formation are still poorly understood. Here, we report the structure of the 100S ribosome by electron cryomicroscopy and single-particle image analysis. The 100S ribosome purified from the cell in the stationary growth phase is composed of two transfer RNA-free 70S ribosomes, has two-fold symmetry, and is formed through interactions between their 30S subunits, where interactions between small subunit proteins, S2, S3 and S5, appear to be critical for the dimerization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5174.map.gz emd_5174.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5174-v30.xml emd-5174-v30.xml emd-5174.xml emd-5174.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5174.png emd_5174.png emd_5174_1.png emd_5174_1.png | 132 KB 139.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5174 http://ftp.pdbj.org/pub/emdb/structures/EMD-5174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5174 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5174.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5174.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The density map of 70S ribosome with part of its partner in the 100S ribosome in hibernation stage. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.69 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli 100S ribosome
| 全体 | 名称: E. coli 100S ribosome |
|---|---|
| 要素 |
|
-超分子 #1000: E. coli 100S ribosome
| 超分子 | 名称: E. coli 100S ribosome / タイプ: sample / ID: 1000 詳細: The 100S ribosome was fixed by GraFix with glutaraldehyde. 集合状態: dimer of 70S ribosome / Number unique components: 1 |
|---|
-超分子 #1: 100S ribosome
| 超分子 | 名称: 100S ribosome / タイプ: complex / ID: 1 / 組換発現: No / データベース: NCBI / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 / 詳細: 20 mM HEPES-KCL, 77 mM KCl, 15 mM (CH3COO)2Mg |
|---|---|
| グリッド | 詳細: Quantifoil 0.6/1 on 200 mesh molybdenum grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 100 K / 装置: OTHER / 詳細: Vitrification instrument: FEI Vitrobot / 手法: Blot for 7 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 88760 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 1.6 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.25 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 1.6 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.25 µm / 倍率(公称値): 50000 |
| 特殊光学系 | エネルギーフィルター - 名称: JEOL Omega filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 10.0 eV |
| 試料ステージ | 試料ホルダー: Top entry liquid helium-cooled cryo specimen holder 試料ホルダーモデル: JEOL |
| 温度 | 最低: 50 K / 最高: 70 K |
| 日付 | 2009年2月25日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F415 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 300 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 2次元分類 | クラス数: 552 |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 18.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 17461 |
| 詳細 | Each of the two 70S ribosome particle images with part of the dimer pair was extracted in a box with circler mask from 100S ribosome particle image. |
 ムービー
ムービー コントローラー
コントローラー