+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4231 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | C1-IgG1 complex on liposomes | |||||||||||||||||||||
 マップデータ マップデータ | C1-IgG1 complex observed on liposomes | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報complement component C1 complex / complement component C1q complex / negative regulation of macrophage differentiation / synapse pruning / negative regulation of granulocyte differentiation / vertebrate eye-specific patterning / complement-mediated synapse pruning / collagen trimer /  補体 / Classical antibody-mediated complement activation ...complement component C1 complex / complement component C1q complex / negative regulation of macrophage differentiation / synapse pruning / negative regulation of granulocyte differentiation / vertebrate eye-specific patterning / complement-mediated synapse pruning / collagen trimer / 補体 / Classical antibody-mediated complement activation ...complement component C1 complex / complement component C1q complex / negative regulation of macrophage differentiation / synapse pruning / negative regulation of granulocyte differentiation / vertebrate eye-specific patterning / complement-mediated synapse pruning / collagen trimer /  補体 / Classical antibody-mediated complement activation / neuron remodeling / Initial triggering of complement / 補体 / Classical antibody-mediated complement activation / neuron remodeling / Initial triggering of complement /  immunoglobulin complex / immunoglobulin complex /  complement activation, classical pathway / complement activation, classical pathway /  Regulation of Complement cascade / astrocyte activation / synapse organization / microglial cell activation / cell-cell signaling / Regulation of Complement cascade / astrocyte activation / synapse organization / microglial cell activation / cell-cell signaling /  amyloid-beta binding / postsynapse / collagen-containing extracellular matrix / blood microparticle / amyloid-beta binding / postsynapse / collagen-containing extracellular matrix / blood microparticle /  獲得免疫系 / 獲得免疫系 /  免疫応答 / 免疫応答 /  自然免疫系 / 自然免疫系 /  シナプス / シナプス /  extracellular space / extracellular region / extracellular space / extracellular region /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 25.0 Å クライオ電子顕微鏡法 / 解像度: 25.0 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Howes SC / Koning RI / de Jong RN / Beurskens FJ / Koster AJ / Sharp TH | |||||||||||||||||||||
| 資金援助 |  オランダ, 6件 オランダ, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2018 ジャーナル: Science / 年: 2018タイトル: Structures of C1-IgG1 provide insights into how danger pattern recognition activates complement. 著者: Deniz Ugurlar / Stuart C Howes / Bart-Jan de Kreuk / Roman I Koning / Rob N de Jong / Frank J Beurskens / Janine Schuurman / Abraham J Koster / Thomas H Sharp / Paul W H I Parren / Piet Gros /  要旨: Danger patterns on microbes or damaged host cells bind and activate C1, inducing innate immune responses and clearance through the complement cascade. How these patterns trigger complement initiation ...Danger patterns on microbes or damaged host cells bind and activate C1, inducing innate immune responses and clearance through the complement cascade. How these patterns trigger complement initiation remains elusive. Here, we present cryo-electron microscopy analyses of C1 bound to monoclonal antibodies in which we observed heterogeneous structures of single and clustered C1-immunoglobulin G1 (IgG1) hexamer complexes. Distinct C1q binding sites are observed on the two Fc-CH2 domains of each IgG molecule. These are consistent with known interactions and also reveal additional interactions, which are supported by functional IgG1-mutant analysis. Upon antibody binding, the C1q arms condense, inducing rearrangements of the C1rs proteases and tilting C1q's cone-shaped stalk. The data suggest that C1r may activate C1s within single, strained C1 complexes or between neighboring C1 complexes on surfaces. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4231.map.gz emd_4231.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4231-v30.xml emd-4231-v30.xml emd-4231.xml emd-4231.xml | 27.3 KB 27.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
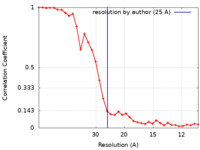
| FSC (解像度算出) |  emd_4231_fsc.xml emd_4231_fsc.xml | 3.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4231.png emd_4231.png | 82.6 KB | ||
| その他 |  emd_4231_additional_1.map.gz emd_4231_additional_1.map.gz emd_4231_additional_2.map.gz emd_4231_additional_2.map.gz emd_4231_additional_3.map.gz emd_4231_additional_3.map.gz emd_4231_additional_4.map.gz emd_4231_additional_4.map.gz emd_4231_half_map_1.map.gz emd_4231_half_map_1.map.gz emd_4231_half_map_2.map.gz emd_4231_half_map_2.map.gz | 10.8 MB 1.7 MB 10.7 MB 10.7 MB 2.2 MB 2.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4231 http://ftp.pdbj.org/pub/emdb/structures/EMD-4231 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4231 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4231 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4231.map.gz / 形式: CCP4 / 大きさ: 2.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4231.map.gz / 形式: CCP4 / 大きさ: 2.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C1-IgG1 complex observed on liposomes | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.38 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Class 1 of neighboring C1-IgG1 complexes
| ファイル | emd_4231_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
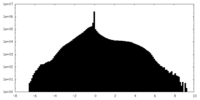
| 注釈 | Class 1 of neighboring C1-IgG1 complexes | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: C1-IgG1 complex observed on liposomes refined using mask...
| ファイル | emd_4231_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
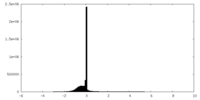
| 注釈 | C1-IgG1 complex observed on liposomes refined using mask that excluded the membrane and Fab regions | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Class 2 of neighboring C1-IgG1 complexes
| ファイル | emd_4231_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
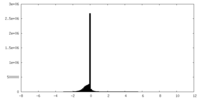
| 注釈 | Class 2 of neighboring C1-IgG1 complexes | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Class 3 of neighboring C1-IgG1 complexes
| ファイル | emd_4231_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
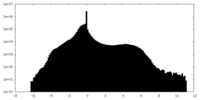
| 注釈 | Class 3 of neighboring C1-IgG1 complexes | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of C1-IgG1 complex observed on liposomes
| ファイル | emd_4231_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of C1-IgG1 complex observed on liposomes | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of C1-IgG1 complex observed on liposomes
| ファイル | emd_4231_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of C1-IgG1 complex observed on liposomes | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C1-IgG1 complex on liposomes
| 全体 | 名称: C1-IgG1 complex on liposomes |
|---|---|
| 要素 |
|
-超分子 #1: C1-IgG1 complex on liposomes
| 超分子 | 名称: C1-IgG1 complex on liposomes / タイプ: complex / ID: 1 / 親要素: 0 詳細: Liposomes containing di-nitrophenyl haptens were incubated with monoclonal antibodies and C1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 700 KDa |
-超分子 #2: IgG1 antibodies
| 超分子 | 名称: IgG1 antibodies / タイプ: complex / ID: 2 / 親要素: 1 詳細: Monoclonal IgG1 recombinantly expressed in HEK293 FreeStyle cells, purified and bound to liposomes. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-超分子 #3: C1 complex
| 超分子 | 名称: C1 complex / タイプ: complex / ID: 3 / 親要素: 1 詳細: C1 complex purified from human serum containing C1q, C1r, and C1s. Map from focused alignment and classification. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 96 % / チャンバー内温度: 294 K / 装置: LEICA EM GP 詳細: 3 microlitres applied and incubated for 30 seconds, blot for 1 second before plunging. | ||||||||||||
| 詳細 | Extruded liposomes were incubated with IgG1 and C1. Gold fiducials were added just before applying to grids. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 53000 Bright-field microscopy / Cs: 2.7 mm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 53000 |
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / エネルギーフィルター - 名称: GIF Quantum LS |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-6 / 平均電子線量: 1.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X