+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4201 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Localized reconstruction of the Rift Valley fever virus glycoprotein pentamer | |||||||||
 マップデータ マップデータ | Localized Reconstruction of Rift Valley fever virus pentamer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  enveloped virus (エンベロープ (ウイルス)) / enveloped virus (エンベロープ (ウイルス)) /  RVFV (リフトバレー熱) / RVFV (リフトバレー熱) /  glycoprotein (糖タンパク質) / glycoprotein (糖タンパク質) /  fusion protein (融合タンパク質) / fusion protein (融合タンパク質) /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell mitochondrial outer membrane / : / host cell Golgi membrane / : / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum membrane / symbiont entry into host cell / fusion of virus membrane with host endosome membrane / virion attachment to host cell / virion membrane /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Rift valley fever virus (リフトバレー熱ウイルス) / Rift valley fever virus (リフトバレー熱ウイルス) /    Rift Valley fever virus (リフトバレー熱ウイルス) Rift Valley fever virus (リフトバレー熱ウイルス) | |||||||||
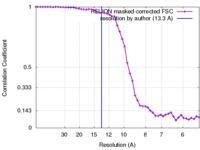
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 13.3 Å クライオ電子顕微鏡法 / 解像度: 13.3 Å | |||||||||
 データ登録者 データ登録者 | Halldorsson S / Huiskonen JT | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: Shielding and activation of a viral membrane fusion protein. 著者: Steinar Halldorsson / Sai Li / Mengqiu Li / Karl Harlos / Thomas A Bowden / Juha T Huiskonen /   要旨: Entry of enveloped viruses relies on insertion of hydrophobic residues of the viral fusion protein into the host cell membrane. However, the intermediate conformations during fusion remain unknown. ...Entry of enveloped viruses relies on insertion of hydrophobic residues of the viral fusion protein into the host cell membrane. However, the intermediate conformations during fusion remain unknown. Here, we address the fusion mechanism of Rift Valley fever virus. We determine the crystal structure of the Gn glycoprotein and fit it with the Gc fusion protein into cryo-electron microscopy reconstructions of the virion. Our analysis reveals how the Gn shields the hydrophobic fusion loops of the Gc, preventing premature fusion. Electron cryotomography of virions interacting with membranes under acidic conditions reveals how the fusogenic Gc is activated upon removal of the Gn shield. Repositioning of the Gn allows extension of Gc and insertion of fusion loops in the outer leaflet of the target membrane. These data show early structural transitions that enveloped viruses undergo during host cell entry and indicate that analogous shielding mechanisms are utilized across diverse virus families. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4201.map.gz emd_4201.map.gz | 7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4201-v30.xml emd-4201-v30.xml emd-4201.xml emd-4201.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4201_fsc.xml emd_4201_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4201.png emd_4201.png | 126.2 KB | ||
| Filedesc metadata |  emd-4201.cif.gz emd-4201.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4201 http://ftp.pdbj.org/pub/emdb/structures/EMD-4201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4201 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6f9fMC  4197C  4198C  4199C  4200C  4202C  4203C  4204C  4205C  4206C  4207C  4208C  4209C  4210C  4211C  6f8pC  6f9bC  6f9cC  6f9dC  6f9eC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4201.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4201.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Localized Reconstruction of Rift Valley fever virus pentamer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rift Valley fever virus
| 全体 | 名称:    Rift Valley fever virus (リフトバレー熱ウイルス) Rift Valley fever virus (リフトバレー熱ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Rift Valley fever virus
| 超分子 | 名称: Rift Valley fever virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Cultured in Vero cells / NCBI-ID: 11588 / 生物種: Rift Valley fever virus / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 名称: Glycoprotein shell / 直径: 1100.0 Å / T番号(三角分割数): 12 |
-分子 #1: Glycoprotein
| 分子 | 名称: Glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Rift valley fever virus (リフトバレー熱ウイルス) Rift valley fever virus (リフトバレー熱ウイルス) |
| 分子量 | 理論値: 34.968902 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EDPHLRNRPG KGHNYIDGMT QEDATCKPVT YAGACSSFDV LLEKGKFPLF QSYAHHRTLL EAVHDTIIAK ADPPSCDLQS AHGNPCMKE KLVMKTHCPN DYQSAHYLNN DGKMASVKCP PKYELTEDCN FCRQMTGASL KKGSYPLQDL FCQSSEDDGS K LKTKMKGV ...文字列: EDPHLRNRPG KGHNYIDGMT QEDATCKPVT YAGACSSFDV LLEKGKFPLF QSYAHHRTLL EAVHDTIIAK ADPPSCDLQS AHGNPCMKE KLVMKTHCPN DYQSAHYLNN DGKMASVKCP PKYELTEDCN FCRQMTGASL KKGSYPLQDL FCQSSEDDGS K LKTKMKGV CEVGVQALKK CDGQLSTAHE VVPFAVFKNS KKVYLDKLDL KTEENLLPDS FVCFEHKGQY KGKLDSGQTK RE LKSFDIS QCPKIGGHGS KKCTGDAAFC SAYECTAQYA NAYCSHANGS GIVQIQVSGV WKKPLCVGYE RVVVKRELS UniProtKB: Envelopment polyprotein |
-分子 #2: Glycoprotein
| 分子 | 名称: Glycoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Rift valley fever virus (リフトバレー熱ウイルス) Rift valley fever virus (リフトバレー熱ウイルス) |
| 分子量 | 理論値: 46.85557 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DPGCSELIQA SSRITTCSTE GVNTKCRLSG TALIRAGSVG AEACLMLKGV KEDQTKFLKI KTVSSELSCR EGQSYWTGSF SPKCLSSRR CHLVGECHVN RCLSWRDNET SAEFSFVGES TTMRENKCFE QCGGWGCGCF NVNPSCLFVH TYLQSVRKEA L RVFNCIDW ...文字列: DPGCSELIQA SSRITTCSTE GVNTKCRLSG TALIRAGSVG AEACLMLKGV KEDQTKFLKI KTVSSELSCR EGQSYWTGSF SPKCLSSRR CHLVGECHVN RCLSWRDNET SAEFSFVGES TTMRENKCFE QCGGWGCGCF NVNPSCLFVH TYLQSVRKEA L RVFNCIDW VHKLTLEITD FDGSVSTIDL GASSSRFTNW GSVSLSLDAE GISGSNSFSF IESPGKGYAI VDEPFSEIPR QG FLGEIRC NSESSVLSAH ESCLRAPNLI SYKPMIDQLE CTTNLIDPFV VFERGSLPQT RNDKTFAASK GNRGVQAFSK GSV QADLTL MFDNFEVDFV GAAVSCDAAF LNLTGCYSCN AGARVCLSIT STGTGSLSAH NKDGSLHIVL PSENGTKDQC QILH FTVPE VEEEFMYSCD GDERPLLVKG TLIAID UniProtKB: Envelopment polyprotein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: PBS |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
| 詳細 | Fixed with 0.2% v/v formaldehyde in PBS |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 22.0 e/Å2 |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6f9f: |
 ムービー
ムービー コントローラー
コントローラー