+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 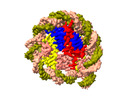 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of nucleosome core particle of Arabidopsis thaliana | |||||||||
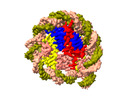
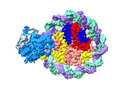
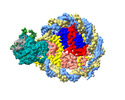
 マップデータ マップデータ | Arabidopsis nucleosome core particle | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  nucleosome (ヌクレオソーム) / nucleosome (ヌクレオソーム) /  chromatin (クロマチン) / chromatin (クロマチン) /  histone (ヒストン) / protein dna interaction / histone (ヒストン) / protein dna interaction /  nucleoprotein (核タンパク質) / nucleoprotein (核タンパク質) /  GENE REGULATION (遺伝子発現の調節) GENE REGULATION (遺伝子発現の調節) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA-mediated transformation / chloroplast thylakoid / chromocenter / response to water deprivation /  原形質連絡 / plant-type vacuole / 原形質連絡 / plant-type vacuole /  チラコイド / チラコイド /  色素体 / 色素体 /  葉緑体 / 葉緑体 /  葉緑体 ...DNA-mediated transformation / chloroplast thylakoid / chromocenter / response to water deprivation / 葉緑体 ...DNA-mediated transformation / chloroplast thylakoid / chromocenter / response to water deprivation /  原形質連絡 / plant-type vacuole / 原形質連絡 / plant-type vacuole /  チラコイド / チラコイド /  色素体 / 色素体 /  葉緑体 / 葉緑体 /  葉緑体 / response to bacterium / response to wounding / 葉緑体 / response to bacterium / response to wounding /  ペルオキシソーム / structural constituent of chromatin / ペルオキシソーム / structural constituent of chromatin /  ヌクレオソーム / protein heterodimerization activity / ヌクレオソーム / protein heterodimerization activity /  核小体 / 核小体 /  DNA binding / extracellular region / DNA binding / extracellular region /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Arabidopsis thaliana (シロイヌナズナ) / synthetic construct (人工物) Arabidopsis thaliana (シロイヌナズナ) / synthetic construct (人工物) | |||||||||
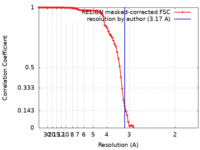
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.17 Å クライオ電子顕微鏡法 / 解像度: 3.17 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Zhang Z / Du J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2024 ジャーナル: Nat Plants / 年: 2024タイトル: Molecular basis of chromatin remodelling by DDM1 involved in plant DNA methylation. 著者: Yue Liu / Zhihui Zhang / Hongmiao Hu / Wei Chen / Fan Zhang / Qian Wang / Changshi Wang / Kaige Yan / Jiamu Du /   要旨: Eukaryotic gene regulation occurs at the chromatin level, which requires changing the chromatin structure by a group of ATP-dependent DNA translocases-namely, the chromatin remodellers. In plants, ...Eukaryotic gene regulation occurs at the chromatin level, which requires changing the chromatin structure by a group of ATP-dependent DNA translocases-namely, the chromatin remodellers. In plants, chromatin remodellers function in various biological processes and possess both conserved and plant-specific components. DECREASE IN DNA METHYLATION 1 (DDM1) is a plant chromatin remodeller that plays a key role in the maintenance DNA methylation. Here we determined the structures of Arabidopsis DDM1 in complex with nucleosome in ADP-BeF-bound, ADP-bound and nucleotide-free conformations. We show that DDM1 specifically recognizes the H4 tail and nucleosomal DNA. The conformational differences between ADP-BeF-bound, ADP-bound and nucleotide-free DDM1 suggest a chromatin remodelling cycle coupled to ATP binding, hydrolysis and ADP release. This, in turn, triggers conformational changes in the DDM1-bound nucleosomal DNA, which alters the nucleosome structure and promotes DNA sliding. Together, our data reveal the molecular basis of chromatin remodelling by DDM1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37538.map.gz emd_37538.map.gz | 5.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37538-v30.xml emd-37538-v30.xml emd-37538.xml emd-37538.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37538_fsc.xml emd_37538_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37538.png emd_37538.png | 99 KB | ||
| Filedesc metadata |  emd-37538.cif.gz emd-37538.cif.gz | 6.5 KB | ||
| その他 |  emd_37538_half_map_1.map.gz emd_37538_half_map_1.map.gz emd_37538_half_map_2.map.gz emd_37538_half_map_2.map.gz | 49.4 MB 49.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37538 http://ftp.pdbj.org/pub/emdb/structures/EMD-37538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37538 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37538.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37538.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Arabidopsis nucleosome core particle | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half1 map of Arabidopsis nucleosome core particle
| ファイル | emd_37538_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half1 map of Arabidopsis nucleosome core particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half2 map of Arabidopsis nucleosome core particle
| ファイル | emd_37538_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half2 map of Arabidopsis nucleosome core particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of the nucleosome core particle of Arabidopsis thaliana
| 全体 | 名称: Structure of the nucleosome core particle of Arabidopsis thaliana |
|---|---|
| 要素 |
|
-超分子 #1: Structure of the nucleosome core particle of Arabidopsis thaliana
| 超分子 | 名称: Structure of the nucleosome core particle of Arabidopsis thaliana タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Histone H3.1
| 分子 | 名称: Histone H3.1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 15.300968 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PATGGVKKPH RFRPGTVALR EIRKYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQSSA VAALQEAAEA YLVGLFEDTN LCAIHAKRVT IMPKDIQLAR RIRGERA UniProtKB:  ヒストンH3 ヒストンH3 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 11.436467 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK IFLENVIRDA VTYTEHARRK TVTAMDVVY ALKRQGRTLY GFGG UniProtKB:  ヒストンH4 ヒストンH4 |
-分子 #3: Histone H2A.6
| 分子 | 名称: Histone H2A.6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 13.680854 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MAGRGKTLGS GGAKKATSRS SKAGLQFPVG RIARFLKAGK YAERVGAGAP VYLAAVLEYL AAEVLELAGN AARDNKKTRI VPRHIQLAV RNDEELSKLL GDVTIANGGV MPNIHNLLLP KKAGASKPQE D UniProtKB: Histone H2A.6 |
-分子 #4: Histone H2B.6
| 分子 | 名称: Histone H2B.6 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 16.474459 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MAPRAEKKPA EKKPAAEKPV EEKSKAEKAP AEKKPKAGKK LPKEAGAGGD KKKKMKKKSV ETYKIYIFKV LKQVHPDIGI SSKAMGIMN SFINDIFEKL ASESSKLARY NKKPTITSRE IQTAVRLVLP GELAKHAVSE GTKAVTKFTS S UniProtKB: Histone H2B.6 |
-分子 #5: DNA (sense strand)
| 分子 | 名称: DNA (sense strand) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 45.123758 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC) ...文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT) (DA)(DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT) (DA)(DA) (DA)(DC)(DG)(DC)(DA)(DC)(DG) (DT)(DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT) (DC)(DC)(DC) (DC)(DC)(DG)(DC)(DG)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DC) (DA)(DA)(DG)(DG) (DG)(DG)(DA)(DT)(DT) (DA)(DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT) (DC)(DT)(DC)(DC)(DA) (DG)(DG)(DC)(DA) (DC)(DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT) (DA)(DT)(DA)(DT)(DA)(DC) (DA)(DT)(DC) (DC)(DG)(DA)(DT) |
-分子 #6: DNA (antisense strand)
| 分子 | 名称: DNA (antisense strand) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.626043 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DG)(DC)(DG)(DG)(DT) (DT)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC)(DT) (DC)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-32 / 実像数: 6776 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X