+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of HSV-1 gB with D48 Fab complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HSV-1 gB /  fab / fab /  neutralizing antibody (中和抗体) / neutralizing antibody (中和抗体) /  complex / complex /  Cryo-EM (低温電子顕微鏡法) / Cryo-EM (低温電子顕微鏡法) /  VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) / VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / host cell endosome membrane / symbiont entry into host cell /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane /  生体膜 / identical protein binding 生体膜 / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /    Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) | |||||||||
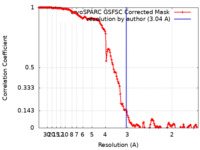
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.04 Å クライオ電子顕微鏡法 / 解像度: 3.04 Å | |||||||||
 データ登録者 データ登録者 | Yang J / Sun C / Fang X / Zeng M / Liu Z | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: hlife / 年: 2023 ジャーナル: hlife / 年: 2023タイトル: The structure of HSV-1 gB bound to a potent neutralizing antibody reveals a conservative antigenic domain across herpesviruses 著者: Sun C / Yang JW / Xie C / Fang XY / Bu GL / Zhao GX / Dai DL / Liu Z / Zeng MS | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37203.map.gz emd_37203.map.gz | 283.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37203-v30.xml emd-37203-v30.xml emd-37203.xml emd-37203.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37203_fsc.xml emd_37203_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37203.png emd_37203.png | 24.1 KB | ||
| Filedesc metadata |  emd-37203.cif.gz emd-37203.cif.gz | 5.8 KB | ||
| その他 |  emd_37203_half_map_1.map.gz emd_37203_half_map_1.map.gz emd_37203_half_map_2.map.gz emd_37203_half_map_2.map.gz | 301.9 MB 301.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37203 http://ftp.pdbj.org/pub/emdb/structures/EMD-37203 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37203 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37203 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8kfaMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37203.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37203.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.827 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_37203_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37203_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of HSV-1 gB with D48 Fab complex
| 全体 | 名称: Cryo-EM structure of HSV-1 gB with D48 Fab complex |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of HSV-1 gB with D48 Fab complex
| 超分子 | 名称: Cryo-EM structure of HSV-1 gB with D48 Fab complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: D48 heavy chain
| 分子 | 名称: D48 heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.742861 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVETGGG VVRPGRSLRL SCTTSGFSFS GSAMHWVRQA PGKGLEWVAV ISHDGNIIQY HDSVKGRFTI SRDNSKNVLL LQMNSLRVD DTAMYYCARD VWLLPATISY AFDFWGQGTM VTVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE P VTVSWNSG ...文字列: EVQLVETGGG VVRPGRSLRL SCTTSGFSFS GSAMHWVRQA PGKGLEWVAV ISHDGNIIQY HDSVKGRFTI SRDNSKNVLL LQMNSLRVD DTAMYYCARD VWLLPATISY AFDFWGQGTM VTVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE P VTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYICN VNHKPSNTKV DKRVGSHHHH HH |
-分子 #2: D48 light chain
| 分子 | 名称: D48 light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.476164 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VIWMTQSPPS LSASIGDTVT ITCRASQGIS NSIAWYQRRP GKAPELLVYA AYRLQSGVPS RLSGSGSGAE YTLTIKNMQP EDFATYYCQ QYYDNPLTFG GGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: VIWMTQSPPS LSASIGDTVT ITCRASQGIS NSIAWYQRRP GKAPELLVYA AYRLQSGVPS RLSGSGSGAE YTLTIKNMQP EDFATYYCQ QYYDNPLTFG GGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #3: Envelope glycoprotein B
| 分子 | 名称: Envelope glycoprotein B / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) |
| 分子量 | 理論値: 70.337844 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ANFYVCPPPT GATVVQFEQP RRCPTRPEGQ NYTEGIAVVF KENIAPYKFK ATMYYKDVTV SQVWFGHRYS QFMGIFEDRA PVPFEEVID KINAKGVCRS TAKYVRNNLE TTAFHRDDHE TDMELKPANA ATRTSRGWHT TDLKYNPSRV EAFHRYGTTV N CIVEEVDA ...文字列: ANFYVCPPPT GATVVQFEQP RRCPTRPEGQ NYTEGIAVVF KENIAPYKFK ATMYYKDVTV SQVWFGHRYS QFMGIFEDRA PVPFEEVID KINAKGVCRS TAKYVRNNLE TTAFHRDDHE TDMELKPANA ATRTSRGWHT TDLKYNPSRV EAFHRYGTTV N CIVEEVDA RSVYPYDEFV LATGDFVYMS PFYGYREGSH TEHTTYAADR FKQVDGFYAR DLTTKARATA PTTRNLLTTP KF TVAWDWV PKRPSVCTMT KWQEVDEMLR SEYGGSFRFS SDAISTTFTT NLTEYPLSRV DLGDCIGKDA RDAMDRIFAR RYN ATHIKV GQPQYYQANG GFLIAYQPLL SNTLAELYVR EHLREQSRKP PNPTPPPPGA SANASVERIK TTSSIEFARL QFTY NHIQR HVNDMLGRVA IAWCELQNHE LTLWNEARKL NPNAIASVTV GRRVSARMLG DVMAVSTCVP VAADNVIVQN SMRIS SRPG ACYSRPLVSF RYEDQGPLVE GQLGENNELR LTRDAIEPCT VGHRRYFTFG GGYVYFEEYA YSHQLSRADI TTVSTF IDL NITMLEDHEF VPLEVYTRHE IKDSGLLDYT EVQRRNQLHD LRFADIDTVI HA UniProtKB: Envelope glycoprotein B |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 1.56 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X