+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Cas12g-sgRNA binary complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  CRISPR-Cas (CRISPR) / CRISPR-Cas (CRISPR) /  RNA BINDING PROTEIN (RNA結合タンパク質) RNA BINDING PROTEIN (RNA結合タンパク質) | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Ouyang SY / Liu MX / Li ZK | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Genet / 年: 2023 ジャーナル: PLoS Genet / 年: 2023タイトル: Structural transitions upon guide RNA binding and their importance in Cas12g-mediated RNA cleavage. 著者: Mengxi Liu / Zekai Li / Jing Chen / Jinying Lin / Qiuhua Lu / Yangmiao Ye / Hongmin Zhang / Bo Zhang / Songying Ouyang /  要旨: Cas12g is an endonuclease belonging to the type V RNA-guided CRISPR-Cas family. It is known for its ability to cleave RNA substrates using a conserved endonuclease active site located in the RuvC ...Cas12g is an endonuclease belonging to the type V RNA-guided CRISPR-Cas family. It is known for its ability to cleave RNA substrates using a conserved endonuclease active site located in the RuvC domain. In this study, we determined the crystal structure of apo-Cas12g, the cryo-EM structure of the Cas12g-sgRNA binary complex and investigated conformational changes that occur during the transition from the apo state to the Cas12g-sgRNA binary complex. The conserved zinc finger motifs in Cas12g undergo an ordered-to-disordered transition from the apo to the sgRNA-bound state and their mutations negatively impact on target RNA cleavage. Moreover, we identified a lid motif in the RuvC domain that undergoes transformation from a helix to loop to regulate the access to the RuvC active site and subsequent cleavage of the RNA substrate. Overall, our study provides valuable insights into the mechanisms by which Cas12g recognizes sgRNA and the conformational changes it undergoes from sgRNA binding to the activation of the RNase active site, thereby laying a foundation for the potential repurposing of Cas12g as a tool for RNA-editing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35154.map.gz emd_35154.map.gz | 4.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35154-v30.xml emd-35154-v30.xml emd-35154.xml emd-35154.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35154.png emd_35154.png | 37.8 KB | ||
| マスクデータ |  emd_35154_msk_1.map emd_35154_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-35154.cif.gz emd-35154.cif.gz | 5.9 KB | ||
| その他 |  emd_35154_half_map_1.map.gz emd_35154_half_map_1.map.gz emd_35154_half_map_2.map.gz emd_35154_half_map_2.map.gz | 40.8 MB 40.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35154 http://ftp.pdbj.org/pub/emdb/structures/EMD-35154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35154 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35154.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35154.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_35154_msk_1.map emd_35154_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
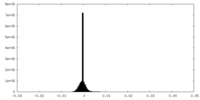
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35154_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
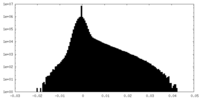
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35154_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas12g-sgRNA binary complex
| 全体 | 名称: Cas12g-sgRNA binary complex |
|---|---|
| 要素 |
|
-超分子 #1: Cas12g-sgRNA binary complex
| 超分子 | 名称: Cas12g-sgRNA binary complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #2: Cas12g
| 超分子 | 名称: Cas12g / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|
-超分子 #3: RNA
| 超分子 | 名称: RNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|
-分子 #1: RNA (139-MER)
| 分子 | 名称: RNA (139-MER) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 44.771703 KDa |
| 配列 | 文字列: GGGAUGCUUA CUUAGUCAUC UGGUUGGCAA ACCUCCGCGG ACCUUCGGGA CCAAUGGAGA GGAACCCAGC CGAGAAGCAU CGAGCCGGU AAAUGAAUUU ACCGGCUCUG ACACCAAUUC GAAAUUAACA CAAACAAGCU |
-分子 #2: Cas12g
| 分子 | 名称: Cas12g / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 89.847672 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MAQASSTPAV SPRPRPRYRE ERTLVRKLLP RPGQSKQEFR ENVKKLRKAF LQFNADVSGV CQWAIQFRPR YGKPAEPTET FWKFFLEPE TSLPPNDSRS PEFRRLQAFE AAAGINGAAA LDDPAFTNEL RDSILAVASR PKTKEAQRLF SRLKDYQPAH R MILAKVAA ...文字列: MAQASSTPAV SPRPRPRYRE ERTLVRKLLP RPGQSKQEFR ENVKKLRKAF LQFNADVSGV CQWAIQFRPR YGKPAEPTET FWKFFLEPE TSLPPNDSRS PEFRRLQAFE AAAGINGAAA LDDPAFTNEL RDSILAVASR PKTKEAQRLF SRLKDYQPAH R MILAKVAA EWIESRYRRA HQNWERNYEE WKKEKQEWEQ NHPELTPEIR EAFNQIFQQL EVKEKRVRIC PAARLLQNKD NC QYAGKNK HSVLCNQFNE FKKNHLQGKA IKFFYKDAEK YLRCGLQSLK PNVQGPFRED WNKYLRYMNL KEETLRGKNG GRL PHCKNL GQECEFNPHT ALCKQYQQQL SSRPDLVQHD ELYRKWRREY WREPRKPVFR YPSVKRHSIA KIFGENYFQA DFKN SVVGL RLDSMPAGQY LEFAFAPWPR NYRPQPGETE ISSVHLHFVG TRPRIGFRFR VPHKRSRFDC TQEELDELRS RTFPR KAQD QKFLEAARKR LLETFPGNAE QELRLLAVAL GTDSARAAFF IGKTFQQAFP LKIVKIEKLY EQWPNQKQAG DRRDAS SKQ PRPGLSRDHV GRHLQKMRAQ ASEIAQKRQE LTGTPAPETT TDQAAKKATL QPFDLRGLTV HTARMIRDWA RLNARQI IQ LAEENQVDLI VLESLRGFRP PGYENLDQEK KRRVAFFAHG RIRRKVTEKA VERGMRVVTV PYLASSKVCA ECRKKQKD N KQWEKNKKRG LFKCEGCGSQ AQVDENAARV LGRVFWGEIE LPTAIP |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 8.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 88696 |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X