+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The EREG-bound EGFR/HER2 ectodomain complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報luteinizing hormone signaling pathway / ovarian cumulus expansion /  排卵 / ERBB4-ERBB4 signaling pathway / negative regulation of smooth muscle cell differentiation / negative regulation of immature T cell proliferation in thymus / ERBB3:ERBB2 complex / ERBB2-ERBB4 signaling pathway / GRB7 events in ERBB2 signaling / immature T cell proliferation in thymus ...luteinizing hormone signaling pathway / ovarian cumulus expansion / 排卵 / ERBB4-ERBB4 signaling pathway / negative regulation of smooth muscle cell differentiation / negative regulation of immature T cell proliferation in thymus / ERBB3:ERBB2 complex / ERBB2-ERBB4 signaling pathway / GRB7 events in ERBB2 signaling / immature T cell proliferation in thymus ...luteinizing hormone signaling pathway / ovarian cumulus expansion /  排卵 / ERBB4-ERBB4 signaling pathway / negative regulation of smooth muscle cell differentiation / negative regulation of immature T cell proliferation in thymus / ERBB3:ERBB2 complex / ERBB2-ERBB4 signaling pathway / GRB7 events in ERBB2 signaling / immature T cell proliferation in thymus / RNA polymerase I core binding / primary follicle stage / female meiotic nuclear division / semaphorin receptor complex / PI3K events in ERBB4 signaling / 排卵 / ERBB4-ERBB4 signaling pathway / negative regulation of smooth muscle cell differentiation / negative regulation of immature T cell proliferation in thymus / ERBB3:ERBB2 complex / ERBB2-ERBB4 signaling pathway / GRB7 events in ERBB2 signaling / immature T cell proliferation in thymus / RNA polymerase I core binding / primary follicle stage / female meiotic nuclear division / semaphorin receptor complex / PI3K events in ERBB4 signaling /  oocyte maturation / regulation of microtubule-based process / ErbB-3 class receptor binding / positive regulation of innate immune response / Sema4D induced cell migration and growth-cone collapse / motor neuron axon guidance / response to hydroxyisoflavone / oocyte maturation / regulation of microtubule-based process / ErbB-3 class receptor binding / positive regulation of innate immune response / Sema4D induced cell migration and growth-cone collapse / motor neuron axon guidance / response to hydroxyisoflavone /  multivesicular body, internal vesicle lumen / positive regulation of prolactin secretion / negative regulation of cardiocyte differentiation / positive regulation of protein kinase C activity / diterpenoid metabolic process / Shc-EGFR complex / neurotransmitter receptor localization to postsynaptic specialization membrane / multivesicular body, internal vesicle lumen / positive regulation of prolactin secretion / negative regulation of cardiocyte differentiation / positive regulation of protein kinase C activity / diterpenoid metabolic process / Shc-EGFR complex / neurotransmitter receptor localization to postsynaptic specialization membrane /  転写 (生物学) / 転写 (生物学) /  月経周期 / Inhibition of Signaling by Overexpressed EGFR / 月経周期 / Inhibition of Signaling by Overexpressed EGFR /  epidermal growth factor receptor activity / EGFR interacts with phospholipase C-gamma / positive regulation of mucus secretion / response to UV-A / epidermal growth factor receptor activity / EGFR interacts with phospholipase C-gamma / positive regulation of mucus secretion / response to UV-A /  epidermal growth factor binding / PLCG1 events in ERBB2 signaling / tongue development / midgut development / ERBB2-EGFR signaling pathway / positive regulation of Rho protein signal transduction / hydrogen peroxide metabolic process / PTK6 promotes HIF1A stabilization / digestive tract morphogenesis / epidermal growth factor binding / PLCG1 events in ERBB2 signaling / tongue development / midgut development / ERBB2-EGFR signaling pathway / positive regulation of Rho protein signal transduction / hydrogen peroxide metabolic process / PTK6 promotes HIF1A stabilization / digestive tract morphogenesis /  regulation of nitric-oxide synthase activity / regulation of nitric-oxide synthase activity /  epidermal growth factor receptor binding / ERBB2 Activates PTK6 Signaling / morphogenesis of an epithelial fold / neuromuscular junction development / intracellular vesicle / Signaling by EGFR / Drug-mediated inhibition of ERBB2 signaling / Resistance of ERBB2 KD mutants to trastuzumab / Resistance of ERBB2 KD mutants to sapitinib / Resistance of ERBB2 KD mutants to tesevatinib / Resistance of ERBB2 KD mutants to neratinib / Resistance of ERBB2 KD mutants to osimertinib / Resistance of ERBB2 KD mutants to afatinib / Resistance of ERBB2 KD mutants to AEE788 / Resistance of ERBB2 KD mutants to lapatinib / epidermal growth factor receptor binding / ERBB2 Activates PTK6 Signaling / morphogenesis of an epithelial fold / neuromuscular junction development / intracellular vesicle / Signaling by EGFR / Drug-mediated inhibition of ERBB2 signaling / Resistance of ERBB2 KD mutants to trastuzumab / Resistance of ERBB2 KD mutants to sapitinib / Resistance of ERBB2 KD mutants to tesevatinib / Resistance of ERBB2 KD mutants to neratinib / Resistance of ERBB2 KD mutants to osimertinib / Resistance of ERBB2 KD mutants to afatinib / Resistance of ERBB2 KD mutants to AEE788 / Resistance of ERBB2 KD mutants to lapatinib /  Drug resistance in ERBB2 TMD/JMD mutants / Drug resistance in ERBB2 TMD/JMD mutants /  enzyme-linked receptor protein signaling pathway / response to cobalamin / negative regulation of epidermal growth factor receptor signaling pathway / enzyme-linked receptor protein signaling pathway / response to cobalamin / negative regulation of epidermal growth factor receptor signaling pathway /  transmembrane receptor protein tyrosine kinase activator activity / ERBB2-ERBB3 signaling pathway / protein tyrosine kinase activator activity / positive regulation of transcription by RNA polymerase I / regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / Signaling by ERBB4 / eyelid development in camera-type eye / oligodendrocyte differentiation / protein insertion into membrane / cerebral cortex cell migration / ERBB2 Regulates Cell Motility / regulation of JNK cascade / semaphorin-plexin signaling pathway / activation of phospholipase C activity / positive regulation of cell division / positive regulation of cyclin-dependent protein serine/threonine kinase activity / PI3K events in ERBB2 signaling / positive regulation of protein targeting to membrane / keratinocyte proliferation / negative regulation of mitotic cell cycle / hair follicle development / positive regulation of cell adhesion / transmembrane receptor protein tyrosine kinase activator activity / ERBB2-ERBB3 signaling pathway / protein tyrosine kinase activator activity / positive regulation of transcription by RNA polymerase I / regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / Signaling by ERBB4 / eyelid development in camera-type eye / oligodendrocyte differentiation / protein insertion into membrane / cerebral cortex cell migration / ERBB2 Regulates Cell Motility / regulation of JNK cascade / semaphorin-plexin signaling pathway / activation of phospholipase C activity / positive regulation of cell division / positive regulation of cyclin-dependent protein serine/threonine kinase activity / PI3K events in ERBB2 signaling / positive regulation of protein targeting to membrane / keratinocyte proliferation / negative regulation of mitotic cell cycle / hair follicle development / positive regulation of cell adhesion /  MAP kinase kinase kinase activity / Estrogen-dependent nuclear events downstream of ESR-membrane signaling / SHC1 events in ERBB4 signaling / positive regulation of G1/S transition of mitotic cell cycle / embryonic placenta development / anatomical structure morphogenesis / positive regulation of bone resorption / positive regulation of protein kinase activity / GAB1 signalosome / MAP kinase kinase kinase activity / Estrogen-dependent nuclear events downstream of ESR-membrane signaling / SHC1 events in ERBB4 signaling / positive regulation of G1/S transition of mitotic cell cycle / embryonic placenta development / anatomical structure morphogenesis / positive regulation of bone resorption / positive regulation of protein kinase activity / GAB1 signalosome /  regulation of angiogenesis / positive regulation of nitric oxide mediated signal transduction / regulation of angiogenesis / positive regulation of nitric oxide mediated signal transduction /  coreceptor activity / Schwann cell development coreceptor activity / Schwann cell development類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.53 Å クライオ電子顕微鏡法 / 解像度: 4.53 Å | |||||||||
 データ登録者 データ登録者 | Zhang Z / Bai X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Discov / 年: 2023 ジャーナル: Cell Discov / 年: 2023タイトル: Structure and dynamics of the EGFR/HER2 heterodimer. 著者: Xue Bai / Pengyu Sun / Xinghao Wang / Changkun Long / Shuyun Liao / Song Dang / Shangshang Zhuang / Yongtao Du / Xinyi Zhang / Nan Li / Kangmin He / Zhe Zhang /  要旨: HER2 belongs to the human epidermal growth factor receptor tyrosine kinase family. Its overexpression or hyperactivation is a leading cause for multiple types of cancers. HER2 functions mainly ...HER2 belongs to the human epidermal growth factor receptor tyrosine kinase family. Its overexpression or hyperactivation is a leading cause for multiple types of cancers. HER2 functions mainly through dimerization with other family members, such as EGFR. However, the molecular details for heterodimer assembly have not been completely understood. Here, we report cryo-EM structures of the EGF- and epiregulin-bound EGFR/HER2 ectodomain complexes at resolutions of 3.3 Å and 4.5 Å, respectively. Together with the functional analyses, we demonstrate that only the dimerization arm of HER2, but not that of EGFR, is essential for their heterodimer formation and signal transduction. Moreover, we analyze the differential membrane dynamics and transient interactions of endogenous EGFR and HER2 molecules in genome-edited cells using single-molecule live-cell imaging. Furthermore, we show that the interaction with HER2 could allow EGFR to resist endocytosis. Together, this work deepens our understanding of the unique structural properties and dynamics of the EGFR/HER2 complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34745.map.gz emd_34745.map.gz | 191.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34745-v30.xml emd-34745-v30.xml emd-34745.xml emd-34745.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34745.png emd_34745.png | 179 KB | ||
| その他 |  emd_34745_half_map_1.map.gz emd_34745_half_map_1.map.gz emd_34745_half_map_2.map.gz emd_34745_half_map_2.map.gz | 200.2 MB 200.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34745 http://ftp.pdbj.org/pub/emdb/structures/EMD-34745 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34745 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34745 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34745.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34745.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
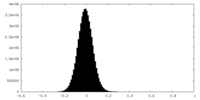
-ハーフマップ: #2
| ファイル | emd_34745_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
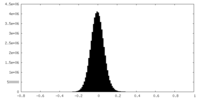
-ハーフマップ: #1
| ファイル | emd_34745_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EREG-bound EGFR/HER2 ectodomain complex
| 全体 | 名称: EREG-bound EGFR/HER2 ectodomain complex |
|---|---|
| 要素 |
|
-超分子 #1: EREG-bound EGFR/HER2 ectodomain complex
| 超分子 | 名称: EREG-bound EGFR/HER2 ectodomain complex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Receptor tyrosine-protein kinase erbB-2
| 分子 | 名称: Receptor tyrosine-protein kinase erbB-2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  受容体型チロシンキナーゼ 受容体型チロシンキナーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 82.149758 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MELAALCRWG LLLALLPPGA ASTQVCTGTD MKLRLPASPE THLDMLRHLY QGCQVVQGNL ELTYLPTNAS LSFLQDIQEV QGYVLIAHN QVRQVPLQRL RIVRGTQLFE DNYALAVLDN GDPLNNTTPV TGASPGGLRE LQLRSLTEIL KGGVLIQRNP Q LCYQDTIL ...文字列: MELAALCRWG LLLALLPPGA ASTQVCTGTD MKLRLPASPE THLDMLRHLY QGCQVVQGNL ELTYLPTNAS LSFLQDIQEV QGYVLIAHN QVRQVPLQRL RIVRGTQLFE DNYALAVLDN GDPLNNTTPV TGASPGGLRE LQLRSLTEIL KGGVLIQRNP Q LCYQDTIL WKDIFHKNNQ LALTLIDTNR SRACHPCSPM CKGSRCWGES SEDCQSLTRT VCAGGCARCK GPLPTDCCHE QC AAGCTGP KHSDCLACLH FNHSGICELH CPALVTYNTD TFESMPNPEG RYTFGASCVT ACPYNYLSTD VGSCTLVCPL HNQ EVTAED GTQRCEKCSK PCARVCYGLG MEHLREVRAV TSANIQEFAG CKKIFGSLAF LPESFDGDPA SNTAPLQPEQ LQVF ETLEE ITGYLYISAW PDSLPDLSVF QNLQVIRGRI LHNGAYSLTL QGLGISWLGL RSLRELGSGL ALIHHNTHLC FVHTV PWDQ LFRNPHQALL HTANRPEDEC VGEGLACHQL CARGHCWGPG PTQCVNCSQF LRGQECVEEC RVLQGLPREY VNARHC LPC HPECQPQNGS VTCFGPEADQ CVACAHYKDP PFCVARCPSG VKPDLSYMPI WKFPDEEGAC QPCPINCTHS CVDLDDK GC PAEQRASPLT SIISAVVGIL LVVVLGVVFG ILIKRRQQKI RKYTMRRLLQ EGGSENLYFQ GGGSAQLEKE LQALEKEN A QLEWELQALE KELAQSNSLE VLFQ |
-分子 #2: Epidermal growth factor receptor
| 分子 | 名称: Epidermal growth factor receptor / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号:  受容体型チロシンキナーゼ 受容体型チロシンキナーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 81.075297 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV VLGNLEITYV QRNYDLSFLK TIQEVAGYV LIALNTVERI PLENLQIIRG NMYYENSYAL AVLSNYDANK TGLKELPMRN LQEILHGAVR FSNNPALCNV E SIQWRDIV ...文字列: MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV VLGNLEITYV QRNYDLSFLK TIQEVAGYV LIALNTVERI PLENLQIIRG NMYYENSYAL AVLSNYDANK TGLKELPMRN LQEILHGAVR FSNNPALCNV E SIQWRDIV SSDFLSNMSM DFQNHLGSCQ KCDPSCPNGS CWGAGEENCQ KLTKIICAQQ CSGRCRGKSP SDCCHNQCAA GC TGPRESD CLVCRKFRDE ATCKDTCPPL MLYNPTTYQM DVNPEGKYSF GATCVKKCPR NYVVTDHGSC VRACGADSYE MEE DGVRKC KKCEGPCRKV CNGIGIGEFK DSLSINATNI KHFKNCTSIS GDLHILPVAF RGDSFTHTPP LDPQELDILK TVKE ITGFL LIQAWPENRT DLHAFENLEI IRGRTKQHGQ FSLAVVSLNI TSLGLRSLKE ISDGDVIISG NKNLCYANTI NWKKL FGTS GQKTKIISNR GENSCKATGQ VCHALCSPEG CWGPEPRDCV SCRNVSRGRE CVDKCNLLEG EPREFVENSE CIQCHP ECL PQAMNITCTG RGPDNCIQCA HYIDGPHCVK TCPAGVMGEN NTLVWKYADA GHVCHLCHPN CTYGCTGPGL EGCPTNG PK IPSIATGMVG ALLLLLVVAL GIGLFMRRRH IVRKRTLRRL LGGSENLYFQ GGGSAAQLKK KLQALKKKNA QLKWKLQA L KKKLAQSNSL EVLFQ |
-分子 #3: Proepiregulin
| 分子 | 名称: Proepiregulin / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5.57841 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: VAQVSITKCS SDMNGYCLHG QCIYLVDMSQ NYCRCEVGYT GVRCEHFFL |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: NOT APPLICABLE |
|---|---|
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.53 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 273388 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8hgp: |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X