+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
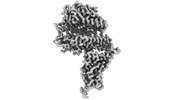
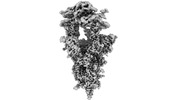
| タイトル | Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with rat ACE2 (local refinement) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  SARS-CoV-2 (SARSコロナウイルス2) / Omicron BA.2 / SARS-CoV-2 (SARSコロナウイルス2) / Omicron BA.2 /  spike protein (スパイクタンパク質) / spike protein (スパイクタンパク質) /  VIRAL PROTEIN (ウイルスタンパク質) / HYDROLASE-VIRAL PROTEIN complex VIRAL PROTEIN (ウイルスタンパク質) / HYDROLASE-VIRAL PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Metabolism of Angiotensinogen to Angiotensins / positive regulation of amino acid transport /  アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane / アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin / regulation of cardiac conduction ...Metabolism of Angiotensinogen to Angiotensins / positive regulation of amino acid transport / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin / regulation of cardiac conduction ...Metabolism of Angiotensinogen to Angiotensins / positive regulation of amino acid transport /  アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane / アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin / regulation of cardiac conduction / peptidyl-dipeptidase activity / maternal process involved in female pregnancy / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin / regulation of cardiac conduction / peptidyl-dipeptidase activity / maternal process involved in female pregnancy /  metallocarboxypeptidase activity / metallocarboxypeptidase activity /  carboxypeptidase activity / negative regulation of signaling receptor activity / positive regulation of cardiac muscle contraction / regulation of transmembrane transporter activity / brush border membrane / negative regulation of smooth muscle cell proliferation / carboxypeptidase activity / negative regulation of signaling receptor activity / positive regulation of cardiac muscle contraction / regulation of transmembrane transporter activity / brush border membrane / negative regulation of smooth muscle cell proliferation /  繊毛 / negative regulation of ERK1 and ERK2 cascade / 繊毛 / negative regulation of ERK1 and ERK2 cascade /  metallopeptidase activity / virus receptor activity / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / metallopeptidase activity / virus receptor activity / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space /  endopeptidase activity / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / endopeptidase activity / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell /  receptor ligand activity / host cell surface receptor binding / symbiont entry into host cell / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / receptor ligand activity / host cell surface receptor binding / symbiont entry into host cell / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane /  細胞膜 / 細胞膜 /  タンパク質分解 / タンパク質分解 /  extracellular space / extracellular region / extracellular space / extracellular region /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) / Rattus norvegicus (ドブネズミ) /   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.29 Å クライオ電子顕微鏡法 / 解像度: 3.29 Å | |||||||||
 データ登録者 データ登録者 | Zhao ZN / Xie YF / Chai Y / Qi JX / Gao GF | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural basis for receptor binding and broader interspecies receptor recognition of currently circulating Omicron sub-variants. 著者: Zhennan Zhao / Yufeng Xie / Bin Bai / Chunliang Luo / Jingya Zhou / Weiwei Li / Yumin Meng / Linjie Li / Dedong Li / Xiaomei Li / Xiaoxiong Li / Xiaoyun Wang / Junqing Sun / Zepeng Xu / ...著者: Zhennan Zhao / Yufeng Xie / Bin Bai / Chunliang Luo / Jingya Zhou / Weiwei Li / Yumin Meng / Linjie Li / Dedong Li / Xiaomei Li / Xiaoxiong Li / Xiaoyun Wang / Junqing Sun / Zepeng Xu / Yeping Sun / Wei Zhang / Zheng Fan / Xin Zhao / Linhuan Wu / Juncai Ma / Odel Y Li / Guijun Shang / Yan Chai / Kefang Liu / Peiyi Wang / George F Gao / Jianxun Qi /  要旨: Multiple SARS-CoV-2 Omicron sub-variants, such as BA.2, BA.2.12.1, BA.4, and BA.5, emerge one after another. BA.5 has become the dominant strain worldwide. Additionally, BA.2.75 is significantly ...Multiple SARS-CoV-2 Omicron sub-variants, such as BA.2, BA.2.12.1, BA.4, and BA.5, emerge one after another. BA.5 has become the dominant strain worldwide. Additionally, BA.2.75 is significantly increasing in some countries. Exploring their receptor binding and interspecies transmission risk is urgently needed. Herein, we examine the binding capacities of human and other 28 animal ACE2 orthologs covering nine orders towards S proteins of these sub-variants. The binding affinities between hACE2 and these sub-variants remain in the range as that of previous variants of concerns (VOCs) or interests (VOIs). Notably, R493Q reverse mutation enhances the bindings towards ACE2s from humans and many animals closely related to human life, suggesting an increased risk of cross-species transmission. Structures of S/hACE2 or RBD/hACE2 complexes for these sub-variants and BA.2 S binding to ACE2 of mouse, rat or golden hamster are determined to reveal the molecular basis for receptor binding and broader interspecies recognition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34217.map.gz emd_34217.map.gz | 454.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34217-v30.xml emd-34217-v30.xml emd-34217.xml emd-34217.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34217.png emd_34217.png | 34 KB | ||
| その他 |  emd_34217_half_map_1.map.gz emd_34217_half_map_1.map.gz emd_34217_half_map_2.map.gz emd_34217_half_map_2.map.gz | 475.6 MB 475.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34217 http://ftp.pdbj.org/pub/emdb/structures/EMD-34217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34217 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gryMC  7yhwC  7yj3C  7yv8C  7yvuC  8h06C  8h5cC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34217.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34217.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_34217_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34217_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with ...
| 全体 | 名称: Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with mouse ACE2 (local refinement) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with ...
| 超分子 | 名称: Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with mouse ACE2 (local refinement) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
-超分子 #2: Rat angiotensin-converting enzyme 2
| 超分子 | 名称: Rat angiotensin-converting enzyme 2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
-超分子 #3: Omicron BA.2 RBD
| 超分子 | 名称: Omicron BA.2 RBD / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Processed angiotensin-converting enzyme 2
| 分子 | 名称: Processed angiotensin-converting enzyme 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 分子量 | 理論値: 69.197625 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SLIEEKAESF LNKFNQEAED LSYQSSLASW NYNTNITEEN AQKMNEAAAK WSAFYEEQSK IAQNFSLQEI QNATIKRQLK ALQQSGSSA LSPDKNKQLN TILNTMSTIY STGKVCNSMN PQECFLLEPG LDEIMATSTD YNRRLWAWEG WRAEVGKQLR P LYEEYVVL ...文字列: SLIEEKAESF LNKFNQEAED LSYQSSLASW NYNTNITEEN AQKMNEAAAK WSAFYEEQSK IAQNFSLQEI QNATIKRQLK ALQQSGSSA LSPDKNKQLN TILNTMSTIY STGKVCNSMN PQECFLLEPG LDEIMATSTD YNRRLWAWEG WRAEVGKQLR P LYEEYVVL KNEMARANNY EDYGDYWRGD YEAEGVEGYN YNRNQLIEDV ENTFKEIKPL YEQLHAYVRT KLMEVYPSYI SP TGCLPAH LLGDMWGRFW TNLYPLTTPF LQKPNIDVTD AMVNQSWDAE RIFKEAEKFF VSVGLPQMTP GFWTNSMLTE PGD DRKVVC HPTAWDLGHG DFRIKMCTKV TMDNFLTAHH EMGHIQYDMA YAKQPFLLRN GANEGFHEAV GEIMSLSAAT PKHL KSIGL LPSNFQEDNE TEINFLLKQA LTIVGTLPFT YMLEKWRWMV FQDKIPREQW TKKWWEMKRE IVGVVEPLPH DETYC DPAS LFHVSNDYSF IRYYTRTIYQ FQFQEALCQA AKHDGPLHKC DISNSTEAGQ KLLNMLSLGN SGPWTLALEN VVGSRN MDV KPLLNYFQPL FVWLKEQNRN STVGWSTDWS PYAD UniProtKB:  アンジオテンシン変換酵素2 アンジオテンシン変換酵素2 |
-分子 #2: Spike protein S1
| 分子 | 名称: Spike protein S1 / タイプ: protein_or_peptide / ID: 2 / 詳細: Omicron BA.2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 分子量 | 理論値: 22.039873 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TNLCPFDEVF NATRFASVYA WNRKRISNCV ADYSVLYNFA PFFAFKCYGV SPTKLNDLCF TNVYADSFVI RGNEVSQIAP GQTGNIADY NYKLPDDFTG CVIAWNSNKL DSKVGGNYNY LYRLFRKSNL KPFERDISTE IYQAGNKPCN GVAGFNCYFP L RSYGFRPT ...文字列: TNLCPFDEVF NATRFASVYA WNRKRISNCV ADYSVLYNFA PFFAFKCYGV SPTKLNDLCF TNVYADSFVI RGNEVSQIAP GQTGNIADY NYKLPDDFTG CVIAWNSNKL DSKVGGNYNY LYRLFRKSNL KPFERDISTE IYQAGNKPCN GVAGFNCYFP L RSYGFRPT YGVGHQPYRV VVLSFELLHA PATVCGP UniProtKB:  スパイクタンパク質 スパイクタンパク質 |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: OTHER |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.29 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 50491 |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X