+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3320 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the ATP-treated CMG replicative helicase (compact state) | |||||||||
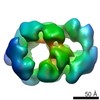
 マップデータ マップデータ | CMG treated with ATP in a tight Mcm5-2 AAA+ configuration | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cdc45 / GINS / MCM / CMG /  helicase (ヘリカーゼ) / helicase (ヘリカーゼ) /  DNA replication (DNA複製) DNA replication (DNA複製) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Unwinding of DNA / Switching of origins to a post-replicative state / DNA endoreduplication / Assembly of the pre-replicative complex / Activation of ATR in response to replication stress / Activation of the pre-replicative complex / eggshell chorion gene amplification / Orc1 removal from chromatin /  DNA amplification / DNA strand elongation involved in mitotic DNA replication ...Unwinding of DNA / Switching of origins to a post-replicative state / DNA endoreduplication / Assembly of the pre-replicative complex / Activation of ATR in response to replication stress / Activation of the pre-replicative complex / eggshell chorion gene amplification / Orc1 removal from chromatin / DNA amplification / DNA strand elongation involved in mitotic DNA replication ...Unwinding of DNA / Switching of origins to a post-replicative state / DNA endoreduplication / Assembly of the pre-replicative complex / Activation of ATR in response to replication stress / Activation of the pre-replicative complex / eggshell chorion gene amplification / Orc1 removal from chromatin /  DNA amplification / DNA strand elongation involved in mitotic DNA replication / GINS complex / mitotic DNA replication preinitiation complex assembly / resolution of meiotic recombination intermediates / premeiotic DNA replication / mitotic DNA replication / CMG complex / MCM complex / DNA replication preinitiation complex / double-strand break repair via break-induced replication / mitotic DNA replication initiation / chromosome condensation / DNA strand elongation involved in DNA replication / DNA duplex unwinding / DNA unwinding involved in DNA replication / DNA amplification / DNA strand elongation involved in mitotic DNA replication / GINS complex / mitotic DNA replication preinitiation complex assembly / resolution of meiotic recombination intermediates / premeiotic DNA replication / mitotic DNA replication / CMG complex / MCM complex / DNA replication preinitiation complex / double-strand break repair via break-induced replication / mitotic DNA replication initiation / chromosome condensation / DNA strand elongation involved in DNA replication / DNA duplex unwinding / DNA unwinding involved in DNA replication /  DNA replication origin binding / DNA replication initiation / DNA replication origin binding / DNA replication initiation /  DNA helicase activity / mitotic spindle organization / meiotic cell cycle / regulation of DNA-templated transcription elongation / DNA helicase activity / mitotic spindle organization / meiotic cell cycle / regulation of DNA-templated transcription elongation /  helicase activity / helicase activity /  single-stranded DNA binding / mitotic cell cycle / single-stranded DNA binding / mitotic cell cycle /  ヘリカーゼ / ヘリカーゼ /  DNA複製 / DNA複製 /  細胞分裂 / 細胞分裂 /  chromatin binding / chromatin binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) | |||||||||
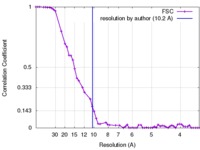
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 10.2 Å クライオ電子顕微鏡法 / 解像度: 10.2 Å | |||||||||
 データ登録者 データ登録者 | Renault L / Costa A | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2016 ジャーナル: Nat Commun / 年: 2016タイトル: Cryo-EM structures of the eukaryotic replicative helicase bound to a translocation substrate. 著者: Ferdos Abid Ali / Ludovic Renault / Julian Gannon / Hailey L Gahlon / Abhay Kotecha / Jin Chuan Zhou / David Rueda / Alessandro Costa /  要旨: The Cdc45-MCM-GINS (CMG) helicase unwinds DNA during the elongation step of eukaryotic genome duplication and this process depends on the MCM ATPase function. Whether CMG translocation occurs on ...The Cdc45-MCM-GINS (CMG) helicase unwinds DNA during the elongation step of eukaryotic genome duplication and this process depends on the MCM ATPase function. Whether CMG translocation occurs on single- or double-stranded DNA and how ATP hydrolysis drives DNA unwinding remain open questions. Here we use cryo-electron microscopy to describe two subnanometre resolution structures of the CMG helicase trapped on a DNA fork. In the predominant state, the ring-shaped C-terminal ATPase of MCM is compact and contacts single-stranded DNA, via a set of pre-sensor 1 hairpins that spiral around the translocation substrate. In the second state, the ATPase module is relaxed and apparently substrate free, while DNA intimately contacts the downstream amino-terminal tier of the MCM motor ring. These results, supported by single-molecule FRET measurements, lead us to suggest a replication fork unwinding mechanism whereby the N-terminal and AAA+ tiers of the MCM work in concert to translocate on single-stranded DNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3320.map.gz emd_3320.map.gz | 3.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3320-v30.xml emd-3320-v30.xml emd-3320.xml emd-3320.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3320_fsc.xml emd_3320_fsc.xml | 5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3320.png emd_3320.png | 1.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3320 http://ftp.pdbj.org/pub/emdb/structures/EMD-3320 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3320 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3320 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3320.map.gz / 形式: CCP4 / 大きさ: 17.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3320.map.gz / 形式: CCP4 / 大きさ: 17.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CMG treated with ATP in a tight Mcm5-2 AAA+ configuration | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.77 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : CMG treated with ATP
+超分子 #1000: CMG treated with ATP
+分子 #1: Mcm2
+分子 #2: Mcm3
+分子 #3: Mcm4
+分子 #4: Mcm5
+分子 #5: Mcm6
+分子 #6: Mcm7
+分子 #7: Cdc45
+分子 #8: Psf1
+分子 #9: Psf2
+分子 #10: Psf3
+分子 #11: Sld5
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 詳細: 25 mM Hepes, 50 mM sodium acetate, 10 mM magnesium acetate, 1 mM DTT, 1mM ATP |
|---|---|
| グリッド | 詳細: 400 mesh quantifoils 1.2/1.3 open holes |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: FEI VITROBOT MARK IV / 手法: Blot for 5 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 47000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 日付 | 2014年12月4日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) デジタル化 - サンプリング間隔: 14 µm / 実像数: 536 / 平均電子線量: 51 e/Å2 / 詳細: data was recorded as movies of 51 frames / ビット/ピクセル: 32 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 unidentified baculovirus (ウイルス)
unidentified baculovirus (ウイルス)