+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3120 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryoEM reconstruction of bnAb PGT128 in complex with BG505 SOSIP.664 Env trimer | |||||||||
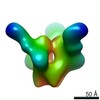
 マップデータ マップデータ | Reconstruction of PGT128 Fab in complex with BG505 SOSIP.664 Env trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  HIV-1 / Env / bnAb / HIV-1 / Env / bnAb /  antibody (抗体) / PGT128 antibody (抗体) / PGT128 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.47 Å クライオ電子顕微鏡法 / 解像度: 4.47 Å | |||||||||
 データ登録者 データ登録者 | Lee JH / de Val N / Lyumkis D / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2015 ジャーナル: Structure / 年: 2015タイトル: Model Building and Refinement of a Natively Glycosylated HIV-1 Env Protein by High-Resolution Cryoelectron Microscopy. 著者: Jeong Hyun Lee / Natalia de Val / Dmitry Lyumkis / Andrew B Ward /  要旨: Secretory and membrane proteins from mammalian cells undergo post-translational modifications, including N-linked glycosylation, which can result in a large number of possible glycoforms. This sample ...Secretory and membrane proteins from mammalian cells undergo post-translational modifications, including N-linked glycosylation, which can result in a large number of possible glycoforms. This sample heterogeneity can be problematic for structural studies, particularly X-ray crystallography. Thus, crystal structures of heavily glycosylated proteins such as the HIV-1 Env viral spike protein have been determined by removing the majority of glycans. This step is most frequently carried out using Endoglycosidase H (EndoH) and requires that all expressed glycans be in the high-mannose form, which is often not the native glycoform. With significantly improved technologies in single-particle cryoelectron microscopy, we demonstrate that it is now possible to refine and build natively glycosylated HIV-1 Env structures in solution to 4.36 Å resolution. At this resolution we can now analyze the complete epitope of a broadly neutralizing antibody (bnAb), PGT128, in the context of the trimer expressed with native glycans. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3120.map.gz emd_3120.map.gz | 59.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3120-v30.xml emd-3120-v30.xml emd-3120.xml emd-3120.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3120.png emd_3120.png | 117.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3120 http://ftp.pdbj.org/pub/emdb/structures/EMD-3120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3120 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3120.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3120.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of PGT128 Fab in complex with BG505 SOSIP.664 Env trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PGT128 Fab bound to BG505 SOSIP.664 HIV-1 Env trimer
| 全体 | 名称: PGT128 Fab bound to BG505 SOSIP.664 HIV-1 Env trimer |
|---|---|
| 要素 |
|
-超分子 #1000: PGT128 Fab bound to BG505 SOSIP.664 HIV-1 Env trimer
| 超分子 | 名称: PGT128 Fab bound to BG505 SOSIP.664 HIV-1 Env trimer タイプ: sample / ID: 1000 集合状態: Three monomers of PGT128 Fab bind one Env trimer Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 570 KDa |
-分子 #1: HIV-1 Envelope glycoprotein
| 分子 | 名称: HIV-1 Envelope glycoprotein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Env 詳細: The Env sequence is from the clade A virus BG505, truncated at residue 664 of gp41, and contains stabilizing SOSIP mutations. コピー数: 3 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: BG505 / 別称: HIV-1 |
| 分子量 | 理論値: 420 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-分子 #2: Immunoglobulin G PGT128
| 分子 | 名称: Immunoglobulin G PGT128 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: IgG PGT128 詳細: The fragment antigen binding (Fab) of PGT128 was used to form the complex. コピー数: 3 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 500 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM Tris, 150 mM NaCl, 0.675 mM DDM |
| グリッド | 詳細: 400 mesh C-Flat CF-2/2-4C, plasma treated for 5 seconds |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 160 K / 装置: HOMEMADE PLUNGER 手法: Grids were frozen using a manual plunger at 4 degrees. |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| Microscopy ID | 1 |
| アライメント法 | Legacy - 非点収差: Objective astigmatism was corrected at 22,500x magnification |
| 日付 | 2014年10月7日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / デジタル化 - サンプリング間隔: 5 µm / 実像数: 2111 / 平均電子線量: 33 e/Å2 詳細: Each full dose image is an aligned stack of frames recorded each using a dose of ~10 e-/Angstrom^2/sec. |
| Tilt angle min | 0 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| Microscopy ID | 2 |
| アライメント法 | Legacy - 非点収差: Objective astigmatism was corrected at 22,500x magnification |
| 日付 | 2014年11月7日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / デジタル化 - サンプリング間隔: 5 µm / 実像数: 2111 / 平均電子線量: 35 e/Å2 詳細: Each full dose image is an aligned stack of frames recorded each using a dose of ~10 e-/Angstrom^2/sec. |
| Tilt angle min | 0 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 4.47 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Imagic, Relion / 使用した粒子像数: 92095 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 4.47 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Imagic, Relion / 使用した粒子像数: 92095 |
 ムービー
ムービー コントローラー
コントローラー