+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of a meiotic triple helix from a pachytene S. cerevisiae cell. | ||||||||||||
 マップデータ マップデータ | NKY611 meiotic triple helix | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 33.0 Å クライオ電子顕微鏡法 / 解像度: 33.0 Å | ||||||||||||
 データ登録者 データ登録者 | Ma OX / Chong W / Lee JKE / Cai S / Siebert CA / Howe A / Zhang P / Shi J / Surana U / Gan L | ||||||||||||
| 資金援助 |  シンガポール, 3件 シンガポール, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2022 ジャーナル: PLoS One / 年: 2022タイトル: Cryo-ET detects bundled triple helices but not ladders in meiotic budding yeast. 著者: Olivia X Ma / Wen Guan Chong / Joy K E Lee / Shujun Cai / C Alistair Siebert / Andrew Howe / Peijun Zhang / Jian Shi / Uttam Surana / Lu Gan /   要旨: In meiosis, cells undergo two sequential rounds of cell division, termed meiosis I and meiosis II. Textbook models of the meiosis I substage called pachytene show that nuclei have conspicuous 100-nm- ...In meiosis, cells undergo two sequential rounds of cell division, termed meiosis I and meiosis II. Textbook models of the meiosis I substage called pachytene show that nuclei have conspicuous 100-nm-wide, ladder-like synaptonemal complexes and ordered chromatin loops. It remains unknown if these cells have any other large, meiosis-related intranuclear structures. Here we present cryo-ET analysis of frozen-hydrated budding yeast cells before, during, and after pachytene. We found no cryo-ET densities that resemble dense ladder-like structures or ordered chromatin loops. Instead, we found large numbers of 12-nm-wide triple-helices that pack into ordered bundles. These structures, herein called meiotic triple helices (MTHs), are present in meiotic cells, but not in interphase cells. MTHs are enriched in the nucleus but not enriched in the cytoplasm. Bundles of MTHs form at the same timeframe as synaptonemal complexes (SCs) in wild-type cells and in mutant cells that are unable to form SCs. These results suggest that in yeast, SCs coexist with previously unreported large, ordered assemblies. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31081.map.gz emd_31081.map.gz | 732.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31081-v30.xml emd-31081-v30.xml emd-31081.xml emd-31081.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31081.png emd_31081.png | 55.5 KB | ||
| その他 |  emd_31081_half_map_1.map.gz emd_31081_half_map_1.map.gz emd_31081_half_map_2.map.gz emd_31081_half_map_2.map.gz | 732.7 KB 733.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31081 http://ftp.pdbj.org/pub/emdb/structures/EMD-31081 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31081 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31081 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31081.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31081.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NKY611 meiotic triple helix | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.61 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map 2
| ファイル | emd_31081_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
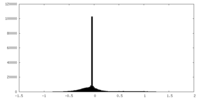
| 投影像・断面図 |
| ||||||||||||
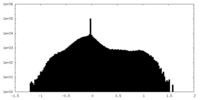
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_31081_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Subtomogram average of a triple helix from pachytene S. cerevisia...
| 全体 | 名称: Subtomogram average of a triple helix from pachytene S. cerevisiae cells. |
|---|---|
| 要素 |
|
-超分子 #1: Subtomogram average of a triple helix from pachytene S. cerevisia...
| 超分子 | 名称: Subtomogram average of a triple helix from pachytene S. cerevisiae cells. タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: NKY611 / Organelle: Nucleus Saccharomyces cerevisiae (パン酵母) / 株: NKY611 / Organelle: Nucleus |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: Sporulation medium (2% potassium acetate) |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: NITROGEN / 装置: FEI VITROBOT MARK IV 詳細: The Vitrobot's cup was used to condense the ethane cryogen. Crimped copper tubes containing cell paste were held ~ 3 cm above the surface of the liquid ethane, then dropped.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 30400 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 18000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 18000 |
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 平均電子線量: 1.6 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 抽出 | トモグラム数: 13 / 使用した粒子像数: 841 |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 33.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0.7) / 使用したサブトモグラム数: 841 |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X