+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of RdrA from Streptococcus suis RADAR defense system | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 | KAP family P-loop domain / KAP family P-loop domain / P-loop containing nucleoside triphosphate hydrolase / KAP NTPase domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |   Streptococcus suis (バクテリア) Streptococcus suis (バクテリア) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.5 Å クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Duncan-Lowey B / Johnson AG / Rawson S / Mayer ML / Kranzusch PJ | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Cryo-EM structure of the RADAR supramolecular anti-phage defense complex. 著者: Brianna Duncan-Lowey / Nitzan Tal / Alex G Johnson / Shaun Rawson / Megan L Mayer / Shany Doron / Adi Millman / Sarah Melamed / Taya Fedorenko / Assaf Kacen / Alexander Brandis / Tevie ...著者: Brianna Duncan-Lowey / Nitzan Tal / Alex G Johnson / Shaun Rawson / Megan L Mayer / Shany Doron / Adi Millman / Sarah Melamed / Taya Fedorenko / Assaf Kacen / Alexander Brandis / Tevie Mehlman / Gil Amitai / Rotem Sorek / Philip J Kranzusch /   要旨: RADAR is a two-protein bacterial defense system that was reported to defend against phage by "editing" messenger RNA. Here, we determine cryo-EM structures of the RADAR defense complex, revealing ...RADAR is a two-protein bacterial defense system that was reported to defend against phage by "editing" messenger RNA. Here, we determine cryo-EM structures of the RADAR defense complex, revealing RdrA as a heptameric, two-layered AAA+ ATPase and RdrB as a dodecameric, hollow complex with twelve surface-exposed deaminase active sites. RdrA and RdrB join to form a giant assembly up to 10 MDa, with RdrA docked as a funnel over the RdrB active site. Surprisingly, our structures reveal an RdrB active site that targets mononucleotides. We show that RdrB catalyzes ATP-to-ITP conversion in vitro and induces the massive accumulation of inosine mononucleotides during phage infection in vivo, limiting phage replication. Our results define ATP mononucleotide deamination as a determinant of RADAR immunity and reveal supramolecular assembly of a nucleotide-modifying machine as a mechanism of anti-phage defense. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29326.map.gz emd_29326.map.gz | 230.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29326-v30.xml emd-29326-v30.xml emd-29326.xml emd-29326.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29326.png emd_29326.png | 75.4 KB | ||
| その他 |  emd_29326_half_map_1.map.gz emd_29326_half_map_1.map.gz emd_29326_half_map_2.map.gz emd_29326_half_map_2.map.gz | 226.4 MB 226.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29326 http://ftp.pdbj.org/pub/emdb/structures/EMD-29326 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29326 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29326 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29326.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29326.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_29326_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
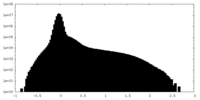
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29326_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
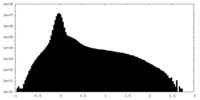
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RdrA heptameric complex
| 全体 | 名称: RdrA heptameric complex |
|---|---|
| 要素 |
|
-超分子 #1: RdrA heptameric complex
| 超分子 | 名称: RdrA heptameric complex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Streptococcus suis (バクテリア) Streptococcus suis (バクテリア) |
-分子 #1: KAP NTPase domain-containing protein
| 分子 | 名称: KAP NTPase domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Streptococcus suis (バクテリア) Streptococcus suis (バクテリア) |
| 分子量 | 理論値: 106.413305 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MTKINWEKYK KVIKKEFSEK EETEEVKNYI FSSQLDKVNL ILEHMDTVGG IHSRNIAITG DRGTGKTSFI ETLKLVLEKQ NYYVFDIVS PTVLSSHLNI LEIVISSIYR EIDQFIDSHD VHDRGRLIQH LKKVMNAIAV EKKQSDYFKQ SKPEIEMLTD L SHRTFLDE ...文字列: MTKINWEKYK KVIKKEFSEK EETEEVKNYI FSSQLDKVNL ILEHMDTVGG IHSRNIAITG DRGTGKTSFI ETLKLVLEKQ NYYVFDIVS PTVLSSHLNI LEIVISSIYR EIDQFIDSHD VHDRGRLIQH LKKVMNAIAV EKKQSDYFKQ SKPEIEMLTD L SHRTFLDE EIKELFCYFK KVLNNRQDSC KEVIKDLVLI IDDLDLVENN LVYDLLRDIQ HYLDSQLIVI FAYKEGQLEQ SM FEHLAKG NEALLNHGVI DSNAIFGQIE RFLTKLVPLS NRIPLFKQDE LLNKTIGEFL ASLDPSYGVG ENLEFITKDS EKN KNNLTI REWFYESIFY RTNLKLDPID IREEASRLMP KTLREMVQLC EELHSMQVIT RSMDKLAGVE GLRKNIGAFR RYIG YKNST YFNLATMEFF QKWELAESHQ ANYLAYHFLM SYYQESFEQN QKLGYPLNLS KSGYPLTLRT MEPYNITLGD IYALM EELK YTEGISADTY YIVYILKVYY SLRLSELLYN VVLHHKLFVH VKEEATTFYM ATSTEELQIE KNHEQEATKL TDKEYR EHI MTAIEKVPAL QAYLELVNAQ FMPQNFNYDR SGSRDDDFYL ISWLKDDDLP EYSRLFKSLF LNSEVAAKGQ IQRNIGK RE SVFRYRNLYS YLPLQLTSAT FYKIDFLAFA IKADLLMYNV VRFVEEEGDT IPYFMSNMFH IDVFVRHNYN ENNNKGKF A YIAKQIVFGL WQGSNQRAHD LKHWYKSFDT VFGTKIEALH LLVDIAEQIK ISDKQTTDVS ALSEEQKRDE QAKKVAEKL AAIYHHIGMS RILSRLHQLP FIAEIKSNKE LLQHFSEAIV KLEKYASDTI NVGNLSQFRE SLKKIGQTYP SIQVLVDKLH RKQKLYVEF IQDFIETVNK LGEADESN |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 42.02 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: OTHER |
|---|---|
| 最終 角度割当 | タイプ: OTHER |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 193305 |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X