+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2827 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
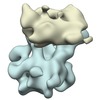
| タイトル | Structure of the mitoribosome with a hyper-rotated 37S subunit from yeast | |||||||||
 マップデータ マップデータ | Subtomogram average of the mitoribosome with a hyper-rotated 37S subunit from yeast | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ribosome (リボソーム) / ribosome (リボソーム) /  mitoribosome / mitoribosome /  mitochondria (ミトコンドリア) / Mba1 / tomography subtomogram analysis mitochondria (ミトコンドリア) / Mba1 / tomography subtomogram analysis | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / クライオ電子顕微鏡法 /  ネガティブ染色法 / 解像度: 40.0 Å ネガティブ染色法 / 解像度: 40.0 Å | |||||||||
 データ登録者 データ登録者 | Pfeffer S / Woellhaf MW / Herrmann JM / Foerster F | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2015 ジャーナル: Nat Commun / 年: 2015タイトル: Organization of the mitochondrial translation machinery studied in situ by cryoelectron tomography. 著者: Stefan Pfeffer / Michael W Woellhaf / Johannes M Herrmann / Friedrich Förster /  要旨: Whereas the structure and function of cytosolic ribosomes have been studied in great detail, we know surprisingly little about the structural basis of mitochondrial protein synthesis. Here we used ...Whereas the structure and function of cytosolic ribosomes have been studied in great detail, we know surprisingly little about the structural basis of mitochondrial protein synthesis. Here we used cryoelectron tomography and subtomogram analysis to visualize mitoribosomes in isolated yeast mitochondria, avoiding perturbations during ribosomal purification. Most mitoribosomes reside in immediate proximity to the inner mitochondrial membrane, in line with their specialization in the synthesis of hydrophobic membrane proteins. The subtomogram average of membrane-associated mitoribosomes reveals two distinct membrane contact sites, formed by the 21S rRNA expansion segment 96-ES1 and the inner membrane protein Mba1. On the basis of our data, we further hypothesize that Mba1 is not just a passive mitoribosome receptor on the inner membrane, but that it spatially aligns mitoribosomes with the membrane insertion machinery. This study reveals detailed insights into the supramolecular organization of the mitochondrial translation machinery and its association with the inner membrane in translation-competent mitochondria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2827.map.gz emd_2827.map.gz | 3.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2827-v30.xml emd-2827-v30.xml emd-2827.xml emd-2827.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2827.tif emd_2827.tif | 564.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2827 http://ftp.pdbj.org/pub/emdb/structures/EMD-2827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2827 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2827.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2827.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of the mitoribosome with a hyper-rotated 37S subunit from yeast | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mitoribosome with a hyper-rotated 37S subunit in isolated mitocho...
| 全体 | 名称: Mitoribosome with a hyper-rotated 37S subunit in isolated mitochondria from yeast |
|---|---|
| 要素 |
|
-超分子 #1000: Mitoribosome with a hyper-rotated 37S subunit in isolated mitocho...
| 超分子 | 名称: Mitoribosome with a hyper-rotated 37S subunit in isolated mitochondria from yeast タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: 73S mitoribosome
| 超分子 | 名称: 73S mitoribosome / タイプ: complex / ID: 1 / 組換発現: No / Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: D273-10B / 別称: Baker's yeast / Organelle: Mitochondrion Saccharomyces cerevisiae (パン酵母) / 株: D273-10B / 別称: Baker's yeast / Organelle: Mitochondrion |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法, ネガティブ染色法,  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 / 詳細: 20 mM Hepes pH 7.6, 50 mM KCl, 2 mM MgCl2 |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: No staining |
| グリッド | 詳細: Lacey carbon molybdenum grids |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / チャンバー内湿度: 60 % / 装置: FEI VITROBOT MARK IV / 手法: Blot time: 4s; blot force: 0 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 7.0 µm / 最小 デフォーカス(公称値): 5.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 7.0 µm / 最小 デフォーカス(公称値): 5.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° |
| 日付 | 2013年11月10日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 平均電子線量: 100 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 40.0 Å / 解像度の算出法: OTHER ソフトウェア - 名称: PyTom, tom_toolbox, av3_toolbox 使用したサブトモグラム数: 120 |
| 詳細 | Tomogram reconstruction and template matching against a single particle cryo-EM reconstruction of the 73S yeast mitoribosome were accomplished using PyTom. Tomogram areas corresponding to cross correlation peaks within mitochondria were visually inspected to identify true positive matches. For the retained coordinates, unbinned subtomograms were reconstructed individually from the weighted projections and iteratively aligned using PyTom. Aligned subtomograms were classified using constrained principal component analysis. |
 ムービー
ムービー コントローラー
コントローラー