+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
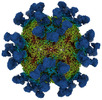
| タイトル | 9H2 Fab-poliovirus 1 complex | ||||||||||||
 マップデータ マップデータ | Cryosparc sharpened | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  Complex / Complex /  Fab / Fab /  poliovirus (ポリオウイルス) / poliovirus (ポリオウイルス) /  neutralizing (中和抗体) / VIRUS-IMMUNE SYSTEM complex neutralizing (中和抗体) / VIRUS-IMMUNE SYSTEM complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host translation initiation / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid ...symbiont-mediated suppression of host translation initiation / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / picornain 3C / T=pseudo3 icosahedral viral capsid ...symbiont-mediated suppression of host translation initiation / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell /  : / : /  カプシド / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / host cell cytoplasm / カプシド / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / host cell cytoplasm /  RNA helicase activity / induction by virus of host autophagy / RNA helicase activity / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |    Human poliovirus 1 Mahoney (ポリオウイルス) / Human poliovirus 1 Mahoney (ポリオウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.13 Å クライオ電子顕微鏡法 / 解像度: 3.13 Å | ||||||||||||
 データ登録者 データ登録者 | Charnesky AJ | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: A human monoclonal antibody binds within the poliovirus receptor-binding site to neutralize all three serotypes. 著者: Andrew J Charnesky / Julia E Faust / Hyunwook Lee / Rama Devudu Puligedda / Daniel J Goetschius / Nadia M DiNunno / Vaskar Thapa / Carol M Bator / Sung Hyun Joseph Cho / Rahnuma Wahid / Kutub ...著者: Andrew J Charnesky / Julia E Faust / Hyunwook Lee / Rama Devudu Puligedda / Daniel J Goetschius / Nadia M DiNunno / Vaskar Thapa / Carol M Bator / Sung Hyun Joseph Cho / Rahnuma Wahid / Kutub Mahmood / Scott Dessain / Konstantin M Chumakov / Amy Rosenfeld / Susan L Hafenstein /  要旨: Global eradication of poliovirus remains elusive, and it is critical to develop next generation vaccines and antivirals. In support of this goal, we map the epitope of human monoclonal antibody 9H2 ...Global eradication of poliovirus remains elusive, and it is critical to develop next generation vaccines and antivirals. In support of this goal, we map the epitope of human monoclonal antibody 9H2 which is able to neutralize the three serotypes of poliovirus. Using cryo-EM we solve the near-atomic structures of 9H2 fragments (Fab) bound to capsids of poliovirus serotypes 1, 2, and 3. The Fab-virus complexes show that Fab interacts with the same binding mode for each serotype and at the same angle of interaction relative to the capsid surface. For each of the Fab-virus complexes, we find that the binding site overlaps with the poliovirus receptor (PVR) binding site and maps across and into a depression in the capsid called the canyon. No conformational changes to the capsid are induced by Fab binding for any complex. Competition binding experiments between 9H2 and PVR reveal that 9H2 impedes receptor binding. Thus, 9H2 outcompetes the receptor to neutralize poliovirus. The ability to neutralize all three serotypes, coupled with the critical importance of the conserved receptor binding site make 9H2 an attractive antiviral candidate for future development. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
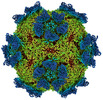
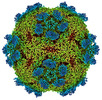
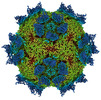
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27943.map.gz emd_27943.map.gz | 631.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27943-v30.xml emd-27943-v30.xml emd-27943.xml emd-27943.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27943.png emd_27943.png | 249.7 KB | ||
| Filedesc metadata |  emd-27943.cif.gz emd-27943.cif.gz | 6.5 KB | ||
| その他 |  emd_27943_additional_1.map.gz emd_27943_additional_1.map.gz emd_27943_half_map_1.map.gz emd_27943_half_map_1.map.gz emd_27943_half_map_2.map.gz emd_27943_half_map_2.map.gz | 603.8 MB 612.9 MB 612.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27943 http://ftp.pdbj.org/pub/emdb/structures/EMD-27943 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27943 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27943 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27943.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27943.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc sharpened | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
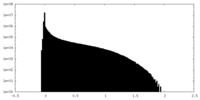
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: DeepEMhancer sharpened
| ファイル | emd_27943_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
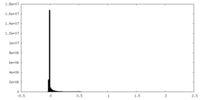
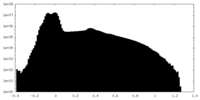
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_27943_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
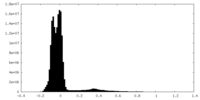
| 密度ヒストグラム |
-ハーフマップ: Half Map B
| ファイル | emd_27943_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
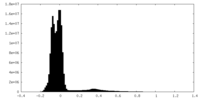
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human poliovirus 1 Mahoney
| 全体 | 名称:    Human poliovirus 1 Mahoney (ポリオウイルス) Human poliovirus 1 Mahoney (ポリオウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human poliovirus 1 Mahoney
| 超分子 | 名称: Human poliovirus 1 Mahoney / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 12081 / 生物種: Human poliovirus 1 Mahoney / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human poliovirus 1 Mahoney (ポリオウイルス) Human poliovirus 1 Mahoney (ポリオウイルス)株: Mahoney |
| 分子量 | 理論値: 31.38424 KDa |
| 配列 | 文字列: ATSRDALPNT EASGPTHSKE IPALTAVETG ATNPLVPSDT VQTRHVVQHR SRSESSIESF FARGACVTIM TVDNPASTTN KDKLFAVWK ITYKDTVQLR RKLEFFTYSR FDMELTFVVT ANFTETNNGH ALNQVYQIMY VPPGAPVPEK WDDYTWQTSS N PSIFYTYG ...文字列: ATSRDALPNT EASGPTHSKE IPALTAVETG ATNPLVPSDT VQTRHVVQHR SRSESSIESF FARGACVTIM TVDNPASTTN KDKLFAVWK ITYKDTVQLR RKLEFFTYSR FDMELTFVVT ANFTETNNGH ALNQVYQIMY VPPGAPVPEK WDDYTWQTSS N PSIFYTYG TAPARISVPY VGISNAYSHF YDGFSKVPLK DQSAALGDSL YGAASLNDFG ILAVRVVNDH NPTKVTSKIR VY LKPKHIR VWCPRPPRAV AYYGPGVDYK DGTLTPLSTK DLTTY UniProtKB: Genome polyprotein |
-分子 #2: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human poliovirus 1 Mahoney (ポリオウイルス) Human poliovirus 1 Mahoney (ポリオウイルス)株: Mahoney |
| 分子量 | 理論値: 29.360998 KDa |
| 配列 | 文字列: GYSDRVLQLT LGNSTITTQE AANSVVAYGR WPEYLRDSEA NPVDQPTEPD VAACRFYTLD TVSWTKESRG WWWKLPDALR DMGLFGQNM YYHYLGRSGY TVHVQCNASK FHQGALGVFA VPEMCLAGDS NTTTMHTSYQ NANPGEKGGT FTGTFTPDNN Q TSPARRFC ...文字列: GYSDRVLQLT LGNSTITTQE AANSVVAYGR WPEYLRDSEA NPVDQPTEPD VAACRFYTLD TVSWTKESRG WWWKLPDALR DMGLFGQNM YYHYLGRSGY TVHVQCNASK FHQGALGVFA VPEMCLAGDS NTTTMHTSYQ NANPGEKGGT FTGTFTPDNN Q TSPARRFC PVDYLLGNGT LLGNAFVFPH QIINLRTNNC ATLVLPYVNS LSIDSMVKHN NWGIAILPLA PLNFASESSP EI PITLTIA PMCCEFNGLR NITLPRLQ UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human poliovirus 1 Mahoney (ポリオウイルス) Human poliovirus 1 Mahoney (ポリオウイルス)株: Mahoney |
| 分子量 | 理論値: 26.235115 KDa |
| 配列 | 文字列: GLPVMNTPGS NQYLTADNFQ SPCALPEFDV TPPIDIPGEV KNMMELAEID TMIPFDLSAT KKNTMEMYRV RLSDKPHTDD PILCLSLSP ASDPRLSHTM LGEILNYYTH WAGSLKFTFL FCGSMMATGK LLVSYAPPGA DPPKKRKEAM LGTHVIWDIG L QSSCTMVV ...文字列: GLPVMNTPGS NQYLTADNFQ SPCALPEFDV TPPIDIPGEV KNMMELAEID TMIPFDLSAT KKNTMEMYRV RLSDKPHTDD PILCLSLSP ASDPRLSHTM LGEILNYYTH WAGSLKFTFL FCGSMMATGK LLVSYAPPGA DPPKKRKEAM LGTHVIWDIG L QSSCTMVV PWISNTTYRQ TIDDSFTEGG YISVFYQTRI VVPLSTPREM DILGFVSACN DFSVRLLRDT THIEQKA UniProtKB: Genome polyprotein |
-分子 #4: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human poliovirus 1 Mahoney (ポリオウイルス) Human poliovirus 1 Mahoney (ポリオウイルス)株: Mahoney |
| 分子量 | 理論値: 7.39305 KDa |
| 配列 | 文字列: GAQVSSQKVG AHENSNRAYG GSTINYTTIN YYRDSASNAA SKQDFSQDPS KFTEPIKDVL IKTAPMLN UniProtKB: Genome polyprotein |
-分子 #5: 9H2 Fab heavy chain
| 分子 | 名称: 9H2 Fab heavy chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.052716 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LVQSGAELKK PGASVKFSCQ ASGFTFTTYD IHWVRQAPGQ GLEWMGMISP SRDSTIYAQK FQGRVTMTSD TSTSTVYMEL TSLRSEDTA LYYCATASRP SAWVFRSLYT YYYMDVWGTG TTVTVSS |
-分子 #6: 9H2 Fab light chain
| 分子 | 名称: 9H2 Fab light chain / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.738903 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSALTQPASV SGSPGQSITI SCTGTITDIG YYNYVSWYQQ HPGKAPKLII FDVTNRPSGV SDRFSGSKSG NTASLTISGL QAEDEGDYY CFSHRSNNIR VFGGGTKLTV L |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.13 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 79471 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.13 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 79471 |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X