+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3H03 Fab in complex with influenza virus neuraminidase from A/Brevig Mission/1/1918 (H1N1) | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  influenza (インフルエンザ) / influenza (インフルエンザ) /  monoclonal antibody (モノクローナル抗体) / NA / monoclonal antibody (モノクローナル抗体) / NA /  H1N1 (H1N1亜型) / cross-reactive antibody / H1N1 (H1N1亜型) / cross-reactive antibody /  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exo-alpha-(2->3)-sialidase activity / exo-alpha-(2->6)-sialidase activity / exo-alpha-(2->8)-sialidase activity /  ノイラミニダーゼ / ノイラミニダーゼ /  viral budding from plasma membrane / carbohydrate metabolic process / host cell plasma membrane / virion membrane / viral budding from plasma membrane / carbohydrate metabolic process / host cell plasma membrane / virion membrane /  生体膜 / 生体膜 /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Influenza A virus (A/Brevig Mission/1/1918(H1N1)) (A型インフルエンザウイルス) / Influenza A virus (A/Brevig Mission/1/1918(H1N1)) (A型インフルエンザウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.7 Å クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Turner HL / Ozorowski G / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2023 ジャーナル: Immunity / 年: 2023タイトル: Human anti-N1 monoclonal antibodies elicited by pandemic H1N1 virus infection broadly inhibit HxN1 viruses in vitro and in vivo. 著者: Lena Hansen / Meagan McMahon / Hannah L Turner / Xueyong Zhu / Jackson S Turner / Gabriel Ozorowski / Daniel Stadlbauer / Juha Vahokoski / Aaron J Schmitz / Amena A Rizk / Wafaa B Alsoussi / ...著者: Lena Hansen / Meagan McMahon / Hannah L Turner / Xueyong Zhu / Jackson S Turner / Gabriel Ozorowski / Daniel Stadlbauer / Juha Vahokoski / Aaron J Schmitz / Amena A Rizk / Wafaa B Alsoussi / Shirin Strohmeier / Wenli Yu / José Alberto Choreño-Parra / Luis Jiménez-Alvarez / Alfredo Cruz-Lagunas / Joaquín Zúñiga / Philip A Mudd / Rebecca J Cox / Ian A Wilson / Andrew B Ward / Ali H Ellebedy / Florian Krammer /    要旨: Neuraminidase (NA) is one of the two influenza virus surface glycoproteins, and antibodies that target it are an independent correlate of protection. However, our current understanding of NA ...Neuraminidase (NA) is one of the two influenza virus surface glycoproteins, and antibodies that target it are an independent correlate of protection. However, our current understanding of NA antigenicity is incomplete. Here, we describe human monoclonal antibodies (mAbs) from a patient with a pandemic H1N1 virus infection in 2009. Two mAbs exhibited broad reactivity and inhibited NA enzyme activity of seasonal H1N1 viruses circulating before and after 2009, as well as viruses with avian or swine N1s. The mAbs provided robust protection from lethal challenge with human H1N1 and avian H5N1 viruses in mice, and both target an epitope on the lateral face of NA. In summary, we identified two broadly protective NA antibodies that share a novel epitope, inhibited NA activity, and provide protection against virus challenge in mice. Our work reaffirms that NA should be included as a target in future broadly protective or universal influenza virus vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27920.map.gz emd_27920.map.gz | 230.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27920-v30.xml emd-27920-v30.xml emd-27920.xml emd-27920.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27920_fsc.xml emd_27920_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27920.png emd_27920.png | 116.5 KB | ||
| マスクデータ |  emd_27920_msk_1.map emd_27920_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_27920_half_map_1.map.gz emd_27920_half_map_1.map.gz emd_27920_half_map_2.map.gz emd_27920_half_map_2.map.gz | 226 MB 226 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27920 http://ftp.pdbj.org/pub/emdb/structures/EMD-27920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27920 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
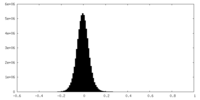
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27920.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27920.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.725 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
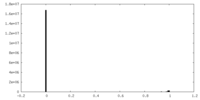
-マスク #1
| ファイル |  emd_27920_msk_1.map emd_27920_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
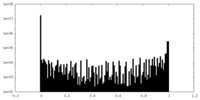
-ハーフマップ: half map A
| ファイル | emd_27920_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
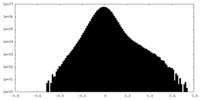
-ハーフマップ: half map B
| ファイル | emd_27920_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 3H03 Fab in complex with influenza virus neuraminidase from A/Bre...
| 全体 | 名称: 3H03 Fab in complex with influenza virus neuraminidase from A/Brevig Mission/1/1918 (H1N1) |
|---|---|
| 要素 |
|
-超分子 #1: 3H03 Fab in complex with influenza virus neuraminidase from A/Bre...
| 超分子 | 名称: 3H03 Fab in complex with influenza virus neuraminidase from A/Brevig Mission/1/1918 (H1N1) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A/Brevig Mission/1/1918(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Brevig Mission/1/1918(H1N1)) (A型インフルエンザウイルス) |
-分子 #1: Neuraminidase
| 分子 | 名称: Neuraminidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  ノイラミニダーゼ ノイラミニダーゼ |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A/Brevig Mission/1/1918(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Brevig Mission/1/1918(H1N1)) (A型インフルエンザウイルス)株: A/Brevig Mission/1/1918 H1N1 |
| 分子量 | 理論値: 49.726387 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: ADPHHHHHHS SSDYSDLQRV KQELLEEVKK ELQKVKEEII EAFVQELRKR GSLVPRGSPS RSVILTGNSS LCPISGWAIY SKDNGIRIG SKGDVFVIRE PFISCSHLEC RTFFLTQGAL LNDKHSNGTV KDRSPYRTLM SCPVGEAPSP YNSRFESVAW S ASACHDGM ...文字列: ADPHHHHHHS SSDYSDLQRV KQELLEEVKK ELQKVKEEII EAFVQELRKR GSLVPRGSPS RSVILTGNSS LCPISGWAIY SKDNGIRIG SKGDVFVIRE PFISCSHLEC RTFFLTQGAL LNDKHSNGTV KDRSPYRTLM SCPVGEAPSP YNSRFESVAW S ASACHDGM GWLTIGISGP DNGAVAVLKY NGIITDTIKS WRNNILRTQE SECACVNGSC FTIMTDGPSN GQASYKILKI EK GKVTKSI ELNAPNYHYE ECSCYPDTGK VMCVCRDNWH GSNRPWVSFD QNLDYQIGYI CSGVFGDNPR PNDGTGSCGP VSS NGANGI KGFSFRYDNG VWIGRTKSTS SRSGFEMIWD PNGWTETDSS FSVRQDIVAI TDWSGYSGSF VQHPELTGLD CMRP CFWVE LIRGQPKENT IWTSGSSISF CGVNSDTVGW SWPDGAELPF SIDK UniProtKB:  ノイラミニダーゼ ノイラミニダーゼ |
-分子 #2: 3H03 fragment antigen binding light chain
| 分子 | 名称: 3H03 fragment antigen binding light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.361855 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSTSVGDRVT ITCRASQTIS TYLNWYQQKP GKAPELLIYV ASSLQSGVPS RFSGTGSGTE FTLTISSLQP GDFATYYCQ QSYSSPFTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSTSVGDRVT ITCRASQTIS TYLNWYQQKP GKAPELLIYV ASSLQSGVPS RFSGTGSGTE FTLTISSLQP GDFATYYCQ QSYSSPFTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #3: 3H03 fragment antigen binding heavy chain
| 分子 | 名称: 3H03 fragment antigen binding heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.484332 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSL TCTVSGDSIS SSYYYWGWIR QSPVKGLEWI GSFFYSGNTN YNPSLKSRVT ISVDTSKNQF SLNLRSVTA ADTAVYYCAR HVTSISSWNR GVYLDSWGRG ALVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF P EPVTVSWN ...文字列: QVQLQESGPG LVKPSETLSL TCTVSGDSIS SSYYYWGWIR QSPVKGLEWI GSFFYSGNTN YNPSLKSRVT ISVDTSKNQF SLNLRSVTA ADTAVYYCAR HVTSISSWNR GVYLDSWGRG ALVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF P EPVTVSWN SGALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKRVEPKS C |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 190000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 3862 / 平均露光時間: 7.51 sec. / 平均電子線量: 41.1 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X