+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron tomograms of cryo-FIB milled WT dividing E. coli | ||||||||||||||||||
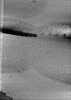
 マップデータ マップデータ | Cryo-electron tomogram of Cryo-FIB milled WT Dividing E. coli. Septation stage. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード |  Cell division (細胞分裂) / Cell division (細胞分裂) /  CELL CYCLE (細胞周期) CELL CYCLE (細胞周期) | ||||||||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||||||||||||||
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | ||||||||||||||||||
 データ登録者 データ登録者 | Navarro PP / Vettiger A / Ananda VY / Montero Llopis P / Allolio C / Bernhardt TG / Chao LH | ||||||||||||||||||
| 資金援助 |  スイス, European Union, スイス, European Union,  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2022 ジャーナル: Nat Microbiol / 年: 2022タイトル: Cell wall synthesis and remodelling dynamics determine division site architecture and cell shape in Escherichia coli. 著者: Paula P Navarro / Andrea Vettiger / Virly Y Ananda / Paula Montero Llopis / Christoph Allolio / Thomas G Bernhardt / Luke H Chao /   要旨: The bacterial division apparatus catalyses the synthesis and remodelling of septal peptidoglycan (sPG) to build the cell wall layer that fortifies the daughter cell poles. Understanding of this ...The bacterial division apparatus catalyses the synthesis and remodelling of septal peptidoglycan (sPG) to build the cell wall layer that fortifies the daughter cell poles. Understanding of this essential process has been limited by the lack of native three-dimensional views of developing septa. Here, we apply state-of-the-art cryogenic electron tomography (cryo-ET) and fluorescence microscopy to visualize the division site architecture and sPG biogenesis dynamics of the Gram-negative bacterium Escherichia coli. We identify a wedge-like sPG structure that fortifies the ingrowing septum. Experiments with strains defective in sPG biogenesis revealed that the septal architecture and mode of division can be modified to more closely resemble that of other Gram-negative (Caulobacter crescentus) or Gram-positive (Staphylococcus aureus) bacteria, suggesting that a conserved mechanism underlies the formation of different septal morphologies. Finally, analysis of mutants impaired in amidase activation (ΔenvC ΔnlpD) showed that cell wall remodelling affects the placement and stability of the cytokinetic ring. Taken together, our results support a model in which competition between the cell elongation and division machineries determines the shape of cell constrictions and the poles they form. They also highlight how the activity of the division system can be modulated to help generate the diverse array of shapes observed in the bacterial domain. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: Cell wall synthesis and remodeling dynamics determine bacterial division site architecture and cell shape 著者: Navarro PP / Vettiger A / Ananda VY / Llopis PM / Allolio C / Bernhardt TG / Chao LH | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27479.map.gz emd_27479.map.gz | 141.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27479-v30.xml emd-27479-v30.xml emd-27479.xml emd-27479.xml | 34.4 KB 34.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27479.png emd_27479.png | 199.4 KB | ||
| Filedesc metadata |  emd-27479.cif.gz emd-27479.cif.gz | 4.6 KB | ||
| その他 |  emd_27479_additional_1.map.gz emd_27479_additional_1.map.gz emd_27479_additional_10.map.gz emd_27479_additional_10.map.gz emd_27479_additional_11.map.gz emd_27479_additional_11.map.gz emd_27479_additional_2.map.gz emd_27479_additional_2.map.gz emd_27479_additional_3.map.gz emd_27479_additional_3.map.gz emd_27479_additional_4.map.gz emd_27479_additional_4.map.gz emd_27479_additional_5.map.gz emd_27479_additional_5.map.gz emd_27479_additional_6.map.gz emd_27479_additional_6.map.gz emd_27479_additional_7.map.gz emd_27479_additional_7.map.gz emd_27479_additional_8.map.gz emd_27479_additional_8.map.gz emd_27479_additional_9.map.gz emd_27479_additional_9.map.gz | 69.4 MB 224.5 MB 26.8 MB 266 MB 254 MB 59.1 MB 103.3 MB 20.7 MB 224.9 MB 24.5 MB 107.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27479 http://ftp.pdbj.org/pub/emdb/structures/EMD-27479 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27479 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27479 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27479.map.gz / 形式: CCP4 / 大きさ: 193 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_27479.map.gz / 形式: CCP4 / 大きさ: 193 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-electron tomogram of Cryo-FIB milled WT Dividing E. coli. Septation stage. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 20.52 Å | ||||||||||||||||||||
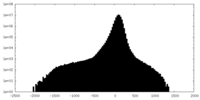
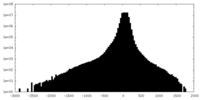
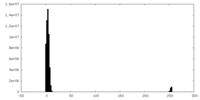
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E. coli. Pole.
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
+追加マップ: Cryo-electron tomogram of Cryo-FIB milled WT Dividing E....
- 試料の構成要素
試料の構成要素
-全体 : Dividing E. coli
| 全体 | 名称: Dividing E. coli |
|---|---|
| 要素 |
|
-超分子 #1: Dividing E. coli
| 超分子 | 名称: Dividing E. coli / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: MG1755 Escherichia coli (大腸菌) / 株: MG1755 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 9 |
|---|---|
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 294.15 K / 装置: FEI VITROBOT MARK IV |
| 切片作成 | 集束イオンビーム - 装置: OTHER / 集束イオンビーム - イオン: OTHER / 集束イオンビーム - 電圧: 5 / 集束イオンビーム - 電流: 0.5 / 集束イオンビーム - 時間: 600 / 集束イオンビーム - 温度: 88 K / 集束イオンビーム - Initial thickness: 1000 / 集束イオンビーム - 最終 厚さ: 150 集束イオンビーム - 詳細: The value given for _em_focused_ion_beam.instrument is Aquilos 2. This is not in a list of allowed values {'OTHER', 'DB235'} so OTHER is written into the XML file. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 3.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 3.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 1.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / ソフトウェア - 名称:  IMOD / 使用した粒子像数: 56 IMOD / 使用した粒子像数: 56 |
|---|
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X