+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate | |||||||||||||||
 マップデータ マップデータ | KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||||||||
 データ登録者 データ登録者 | Spangler CJ / Skrajna A / Foley CA / Budziszewski GR / Azzam DN / James LI / Frye SV / McGinty RK | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: Structural basis of paralog-specific KDM2A/B nucleosome recognition. 著者: Cathy J Spangler / Aleksandra Skrajna / Caroline A Foley / Anh Nguyen / Gabrielle R Budziszewski / Dalal N Azzam / Eyla C Arteaga / Holly C Simmons / Charlotte B Smith / Nathaniel A Wesley / ...著者: Cathy J Spangler / Aleksandra Skrajna / Caroline A Foley / Anh Nguyen / Gabrielle R Budziszewski / Dalal N Azzam / Eyla C Arteaga / Holly C Simmons / Charlotte B Smith / Nathaniel A Wesley / Emily M Wilkerson / Jeanne-Marie E McPherson / Dmitri Kireev / Lindsey I James / Stephen V Frye / Dennis Goldfarb / Robert K McGinty /  要旨: The nucleosome acidic patch is a major interaction hub for chromatin, providing a platform for enzymes to dock and orient for nucleosome-targeted activities. To define the molecular basis of acidic ...The nucleosome acidic patch is a major interaction hub for chromatin, providing a platform for enzymes to dock and orient for nucleosome-targeted activities. To define the molecular basis of acidic patch recognition proteome wide, we performed an amino acid resolution acidic patch interactome screen. We discovered that the histone H3 lysine 36 (H3K36) demethylase KDM2A, but not its closely related paralog, KDM2B, requires the acidic patch for nucleosome binding. Despite fundamental roles in transcriptional repression in health and disease, the molecular mechanisms governing nucleosome substrate specificity of KDM2A/B, or any related JumonjiC (JmjC) domain lysine demethylase, remain unclear. We used a covalent conjugate between H3K36 and a demethylase inhibitor to solve cryogenic electron microscopy structures of KDM2A and KDM2B trapped in action on a nucleosome substrate. Our structures show that KDM2-nucleosome binding is paralog specific and facilitated by dynamic nucleosomal DNA unwrapping and histone charge shielding that mobilize the H3K36 sequence for demethylation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26810.map.gz emd_26810.map.gz | 9.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26810-v30.xml emd-26810-v30.xml emd-26810.xml emd-26810.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26810_fsc.xml emd_26810_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26810.png emd_26810.png | 119.8 KB | ||
| その他 |  emd_26810_half_map_1.map.gz emd_26810_half_map_1.map.gz emd_26810_half_map_2.map.gz emd_26810_half_map_2.map.gz | 41.3 MB 41.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26810 http://ftp.pdbj.org/pub/emdb/structures/EMD-26810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26810 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26810.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26810.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate half...
| ファイル | emd_26810_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
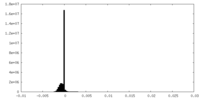
| 密度ヒストグラム |
-ハーフマップ: KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate half...
| ファイル | emd_26810_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
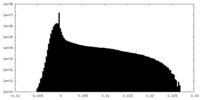
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent co...
| 全体 | 名称: KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate |
|---|---|
| 要素 |
|
-超分子 #1: KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent co...
| 超分子 | 名称: KDM2B-nucleosome complex stabilized by H3K36C-UNC8015 covalent conjugate タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 320 KDa |
-分子 #1: human histone H3
| 分子 | 名称: human histone H3 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVCKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAVM ALQEASEAYL VGLFEDTNLA AIHAKRVTIM PKDIQLARRI RGERA |
-分子 #2: human histone H4
| 分子 | 名称: human histone H4 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYAL KRQGRTLYGF GG |
-分子 #3: human histone H2A
| 分子 | 名称: human histone H2A / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: SGRGKQGGKA RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAIRND EELNKLLGKV TIAQGGVLPN IQAVLLPKKT ESHHKAKGK |
-分子 #4: human histone H2B
| 分子 | 名称: human histone H2B / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: PEPAKSAPAP KKGSKKAVTK AQKKDGKKRK RSRKESYSVY VYKVLKQVHP DTGISSKAMG IMNSFVNDIF ERIAGEASRL AHYNKRSTIT SREIQTAVRL LLPGELAKHA VSEGTKAVTK YTSSK |
-分子 #7: Lysine-specific demethylase 2B (KDM2B)
| 分子 | 名称: Lysine-specific demethylase 2B (KDM2B) / タイプ: protein_or_peptide / ID: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: GSEPEEERIR YSQRLRGTMR RRYEDDGISD DEIEGKRTFD LEEKLHTNKY NANFVTFMEG KDFNVEYIQR GGLRDPLIFK NSDGLGIKMP DPDFTVNDVK MCVGSRRMVD VMDVNTQKGI EMTMAQWTRY YETPEEEREK LYNVISLEFS HTRLENMVQR PSTVDFIDWV ...文字列: GSEPEEERIR YSQRLRGTMR RRYEDDGISD DEIEGKRTFD LEEKLHTNKY NANFVTFMEG KDFNVEYIQR GGLRDPLIFK NSDGLGIKMP DPDFTVNDVK MCVGSRRMVD VMDVNTQKGI EMTMAQWTRY YETPEEEREK LYNVISLEFS HTRLENMVQR PSTVDFIDWV DNMWPRHLKE SQTESTNAIL EMQYPKVQKY CLMSVRGCYT DFHVDFGGTS VWYHIHQGGK VFWLIPPTAH NLELYENWLL SGKQGDIFLG DRVSDCQRIE LKQGYTFVIP SGWIHAVYTP TDTLVFGGNF LHSFNIPMQL KIYNIEDRTR VPNKFRYPFY YEMCWYVLER YVYCITNRSH LTKEFQKESL SMDLELNGLE SGNGDEEAVD REPRRLSSRR SVLTSPVANG VNLDYDGLGK TCRSLPSLKK TLAGDSSSDC SRGSHNGQVW DPQCAPRKDR QVHLTHFELE GLRCLVDKLE SLPLHKKCVP TGIEDEDALI ADVKILLEEL ANSDPKLALT GVPIVQWPKR DKLKFPTRPK VRVPTIPITK PHTMKPAPRL TPVRPAAASP IVSGARRRRV RCRKCKACVQ GECGVCHYCR DMKKFGGPGR MKQSCVLRQC LAPRLPHSVT CSLCGEVDQN EETQDFEKKL MECCICNEIV HPGCLQMDGE GLLNEELPNC WECPKCYQED SSEKAQHHHH HH |
-分子 #5: DNA 185-MER
| 分子 | 名称: DNA 185-MER / タイプ: dna / ID: 5 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: ATCCCTATAC GCGGCCGCCC TGGAGAATCC CGGTGCCGAG GCCGCTCAAT TGGTCGTAGA CAGCTCTAGC ACCGCTTAAA CGCACGTACG CGCTGTCCCC CGCGTTTTAA CCGCCAAGGG GATTACTCCC TAGTCTCCAG GCACGTGTCA GATATATACA TCCTGTGCAT GTATTGAACA GCGAT |
-分子 #6: DNA 185-MER
| 分子 | 名称: DNA 185-MER / タイプ: dna / ID: 6 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: ATCGCTGTTC AATACATGCA CAGGATGTAT ATATCTGACA CGTGCCTGGA GACTAGGGAG TAATCCCCTT GGCGGTTAAA ACGCGGGGGA CAGCGCGTAC GTGCGTTTAA GCGGTGCTAG AGCTGTCTAC GACCAATTGA GCGGCCTCGG CACCGGGATT CTCCAGGGCG GCCGCGTATA GGGAT |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 45000 Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 15671 / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X