+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2612 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Incomplete pneumolysin oligomers form membrane pores | |||||||||
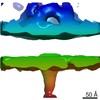
 マップデータ マップデータ | Sub-tomogram average of 197 pre-pores. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cholesterol-dependent cytolysin (コレステロール依存性細胞溶解素) / cholesterol-dependent cytolysin (コレステロール依存性細胞溶解素) /  pneumolysin / proteolipid toroidal pore pneumolysin / proteolipid toroidal pore | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 cholesterol binding / cholesterol binding /  toxin activity / killing of cells of another organism / host cell plasma membrane / extracellular region / toxin activity / killing of cells of another organism / host cell plasma membrane / extracellular region /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 | synthetic construct (人工物) /   Streptococcus pneumoniae (肺炎レンサ球菌) Streptococcus pneumoniae (肺炎レンサ球菌) | |||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 29.0 Å クライオ電子顕微鏡法 / 解像度: 29.0 Å | |||||||||
 データ登録者 データ登録者 | Sonnen AF-P / Plitzko JM / Gilbert RJC | |||||||||
 引用 引用 |  ジャーナル: Open Biol / 年: 2014 ジャーナル: Open Biol / 年: 2014タイトル: Incomplete pneumolysin oligomers form membrane pores. 著者: Andreas F-P Sonnen / Jürgen M Plitzko / Robert J C Gilbert /  要旨: Pneumolysin is a member of the cholesterol-dependent cytolysin (CDC) family of pore-forming proteins that are produced as water-soluble monomers or dimers, bind to target membranes and oligomerize ...Pneumolysin is a member of the cholesterol-dependent cytolysin (CDC) family of pore-forming proteins that are produced as water-soluble monomers or dimers, bind to target membranes and oligomerize into large ring-shaped assemblies comprising approximately 40 subunits and approximately 30 nm across. This pre-pore assembly then refolds to punch a large hole in the lipid bilayer. However, in addition to forming large pores, pneumolysin and other CDCs form smaller lesions characterized by low electrical conductance. Owing to the observation of arc-like (rather than full-ring) oligomers by electron microscopy, it has been hypothesized that smaller oligomers explain smaller functional pores. To investigate whether this is the case, we performed cryo-electron tomography of pneumolysin oligomers on model lipid membranes. We then used sub-tomogram classification and averaging to determine representative membrane-bound low-resolution structures and identified pre-pores versus pores by the presence of membrane within the oligomeric curve. We found pre-pore and pore forms of both complete (ring) and incomplete (arc) oligomers and conclude that arc-shaped oligomeric assemblies of pneumolysin can form pores. As the CDCs are evolutionarily related to the membrane attack complex/perforin family of proteins, which also form variably sized pores, our findings are of relevance to that class of proteins as well. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2612.map.gz emd_2612.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2612-v30.xml emd-2612-v30.xml emd-2612.xml emd-2612.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD2612.gif EMD2612.gif | 39.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2612 http://ftp.pdbj.org/pub/emdb/structures/EMD-2612 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2612 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2612 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2612.map.gz / 形式: CCP4 / 大きさ: 4.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2612.map.gz / 形式: CCP4 / 大きさ: 4.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-tomogram average of 197 pre-pores. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.72 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pneumolysin prepore formed on a cholesterol-containing liposome
| 全体 | 名称: Pneumolysin prepore formed on a cholesterol-containing liposome |
|---|---|
| 要素 |
|
-超分子 #1000: Pneumolysin prepore formed on a cholesterol-containing liposome
| 超分子 | 名称: Pneumolysin prepore formed on a cholesterol-containing liposome タイプ: sample / ID: 1000 / 集合状態: Full ring oligomer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 2 MDa |
-超分子 #1: synthetic membrane
| 超分子 | 名称: synthetic membrane / タイプ: organelle_or_cellular_component / ID: 1 詳細: Liposome formed of a 10:10:1 mixture of phosphatidylcholine:cholesterol:dicetyl phosphate 組換発現: No |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: pneumolysin
| 分子 | 名称: pneumolysin / タイプ: protein_or_peptide / ID: 1 詳細: pneumolysin added to the synthetic membranes spontaneously forms oligomers on their surfaces. 集合状態: 40mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Streptococcus pneumoniae (肺炎レンサ球菌) / 細胞中の位置: cytoplasm Streptococcus pneumoniae (肺炎レンサ球菌) / 細胞中の位置: cytoplasm |
| 分子量 | 実験値: 52 KDa / 理論値: 52 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: M15 / 組換プラスミド: pKK233-2 Escherichia coli (大腸菌) / 組換株: M15 / 組換プラスミド: pKK233-2 |
| 配列 | UniProtKB:  Pneumolysin Pneumolysin |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: PBS |
|---|---|
| グリッド | 詳細: C-flat or lacy carbon-coated grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 100 K / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 8.0 µm / 倍率(公称値): 64171 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 8.0 µm / 倍率(公称値): 64171 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF2002 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダー: Nitrogen cooled / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -65 ° / Tilt series - Axis1 - Max angle: 65 ° |
| 日付 | 2008年6月1日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN MULTISCAN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 29.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: TOM, Toolbox, Bsoft, XMIPP / 使用したサブトモグラム数: 197 |
|---|---|
| 詳細 | Subtomograms were selected manually in Bshow and subjected to automatic maximum-likelihood based classification using XMIPP. |
 ムービー
ムービー コントローラー
コントローラー