+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 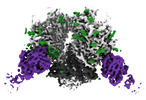 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The Envelope Glycoprotein SIVmac239.K180S SOSIP trimer in complex with 3 copies of the neutralizing antibody K11 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SIV / Env /  vaccine (ワクチン) / vaccine (ワクチン) /  neutralizing antibody (中和抗体) / neutralizing antibody (中和抗体) /  glycoprotein (糖タンパク質) / glycoprotein (糖タンパク質) /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane fusion involved in viral entry into host cell / host cell endosome membrane / membrane => GO:0016020 / symbiont entry into host cell /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Simian immunodeficiency virus (サル免疫不全ウイルス) / Simian immunodeficiency virus (サル免疫不全ウイルス) /   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Berndsen ZT / Ward AB | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Molecular insights into antibody-mediated protection against the prototypic simian immunodeficiency virus. 著者: Fangzhu Zhao / Zachary T Berndsen / Nuria Pedreño-Lopez / Alison Burns / Joel D Allen / Shawn Barman / Wen-Hsin Lee / Srirupa Chakraborty / Sandrasegaram Gnanakaran / Leigh M Sewall / ...著者: Fangzhu Zhao / Zachary T Berndsen / Nuria Pedreño-Lopez / Alison Burns / Joel D Allen / Shawn Barman / Wen-Hsin Lee / Srirupa Chakraborty / Sandrasegaram Gnanakaran / Leigh M Sewall / Gabriel Ozorowski / Oliver Limbo / Ge Song / Peter Yong / Sean Callaghan / Jessica Coppola / Kim L Weisgrau / Jeffrey D Lifson / Rebecca Nedellec / Thomas B Voigt / Fernanda Laurino / Johan Louw / Brandon C Rosen / Michael Ricciardi / Max Crispin / Ronald C Desrosiers / Eva G Rakasz / David I Watkins / Raiees Andrabi / Andrew B Ward / Dennis R Burton / Devin Sok /   要旨: SIVmac239 infection of macaques is a favored model of human HIV infection. However, the SIVmac239 envelope (Env) trimer structure, glycan occupancy, and the targets and ability of neutralizing ...SIVmac239 infection of macaques is a favored model of human HIV infection. However, the SIVmac239 envelope (Env) trimer structure, glycan occupancy, and the targets and ability of neutralizing antibodies (nAbs) to protect against SIVmac239 remain unknown. Here, we report the isolation of SIVmac239 nAbs that recognize a glycan hole and the V1/V4 loop. A high-resolution structure of a SIVmac239 Env trimer-nAb complex shows many similarities to HIV and SIVcpz Envs, but with distinct V4 features and an extended V1 loop. Moreover, SIVmac239 Env has a higher glycan shield density than HIV Env that may contribute to poor or delayed nAb responses in SIVmac239-infected macaques. Passive transfer of a nAb protects macaques from repeated intravenous SIVmac239 challenge at serum titers comparable to those described for protection of humans against HIV infection. Our results provide structural insights for vaccine design and shed light on antibody-mediated protection in the SIV model. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
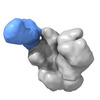
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25676.map.gz emd_25676.map.gz | 97.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25676-v30.xml emd-25676-v30.xml emd-25676.xml emd-25676.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25676_fsc.xml emd_25676_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25676.png emd_25676.png | 98.9 KB | ||
| マスクデータ |  emd_25676_msk_1.map emd_25676_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_25676_half_map_1.map.gz emd_25676_half_map_1.map.gz emd_25676_half_map_2.map.gz emd_25676_half_map_2.map.gz | 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25676 http://ftp.pdbj.org/pub/emdb/structures/EMD-25676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25676 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7t4gMC  7t2pC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25676.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25676.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25676_msk_1.map emd_25676_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
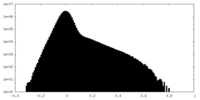
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_25676_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
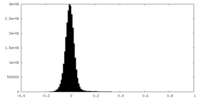
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_25676_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
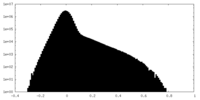
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SIVmac239.K180S SOSIP trimer in complex with 3 copies of K11 IgG
| 全体 | 名称: SIVmac239.K180S SOSIP trimer in complex with 3 copies of K11 IgG |
|---|---|
| 要素 |
|
-超分子 #1: SIVmac239.K180S SOSIP trimer in complex with 3 copies of K11 IgG
| 超分子 | 名称: SIVmac239.K180S SOSIP trimer in complex with 3 copies of K11 IgG タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
-分子 #1: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 分子量 | 理論値: 60.324441 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGCLGNQLLI AILLLSVYGI YCTLYVTVFY GVPAWRNATI PLFCATKNRD TWGTTQCLPD NGDYSEMALN VTESFDAWNN TVTEQAIED VWQLFETSIK PCVKLSPLCI TMRCNKSETD RWGLTKSITT TASTTSTTAS AKVDMVNETS SCIAQDNCTG L EQEQMISC ...文字列: MGCLGNQLLI AILLLSVYGI YCTLYVTVFY GVPAWRNATI PLFCATKNRD TWGTTQCLPD NGDYSEMALN VTESFDAWNN TVTEQAIED VWQLFETSIK PCVKLSPLCI TMRCNKSETD RWGLTKSITT TASTTSTTAS AKVDMVNETS SCIAQDNCTG L EQEQMISC KFNMTGLKRD KSKEYNETWY SADLVCEQGN NTGNESRCYM NHCNTSVIQE SCDKHYWDAI RFRYCAPPGY AL LRCNDTN YSGFMPKCSK VVVSSCTRMM ETQTSTWFGF NGTRAENRTY IYWHGRDNRT IISLNKYYNL TMKCRRPGNK TVL PVTIMS GLVFHSQPIN DRPKQAWCWF GGKWKDAIKE VKQTIVKHPR YTGTNNTDKI NLTAPGGGDP EVTFMWTNCR GEFL YCKMN WFLNWVEDRN TANQKPKEQH KRNYVPCHIR QIINTWHKVG KNVYLPPREG DLTCNSTVTS LIANIDWIDG NQTNI TMSA EVAELYRLEL GDYKLVEITP IGLAPTSCKR YTTGGTSRRR RRR |
-分子 #2: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 分子量 | 理論値: 18.1446 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GVFVLGFLGF LATAGSAMGA ASLTLTAQSR TLLAGIVQQQ QQLLDVPKRQ QELLRLTVWG TKNLQTRVTA IEKYLKDQAQ LNAWGCAFR QVCCTTVPWP NASLIPKWNN ETWQEWERKV DFLEENITAL LEEAQIQQEK NMYELQKLNG SGHHHHHHHH |
-分子 #3: K11 IgG heavy chain
| 分子 | 名称: K11 IgG heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
| 分子量 | 理論値: 53.29077 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DWTWRILFLV AAATGAHSQV QLQESGPGVV RPSQTLSLTC AVSGDTVSSC CFFWTWIRQP PGKGLEWIGN IYSDNDNTNY NPSLKTRIS ISKDMSKNQF SLKLNSLTAT DTAIYYCARE SPSRGNFCYA YLYGNCPLHF DLWGQGVLVT VSSASTKGPS V FPLAPSSR ...文字列: DWTWRILFLV AAATGAHSQV QLQESGPGVV RPSQTLSLTC AVSGDTVSSC CFFWTWIRQP PGKGLEWIGN IYSDNDNTNY NPSLKTRIS ISKDMSKNQF SLKLNSLTAT DTAIYYCARE SPSRGNFCYA YLYGNCPLHF DLWGQGVLVT VSSASTKGPS V FPLAPSSR STSESTAALG CLVKDYFPEP VTVSWNSGSL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYVCNVN HK PSNTKVD KRVEIKTCGG GSKPPTCPPC TSPELLGGPS VFLFPPKPKD TLMISRTPEV TCVVVDVSQE DPDVKFNWYV NGA EVHHAQ TKPRETQYNS TYRVVSVLTV THQDWLNGKE YTCKVSNKAL PAPIQKTISK DKGQPREPQV YTLPPSREEL TKNQ VSLTC LVKGFYPSDI VVEWESSGQP ENTYKTTPPV LDSDGSYFLY SKLTVDKSRW QQGNVFSCSV MHEALHNHYT QKSLS LSPG K |
-分子 #4: K11 IgG light chain
| 分子 | 名称: K11 IgG light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
| 分子量 | 理論値: 25.29315 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ETDTLLLWVL LLWVPGSTGD ILLTQSPSSL SGSVGDRVTI TCRASQGINS YLNWYQQKPG KAPKLLIYFA NRLQSGVPSR FSGSGSGTE FTLTISSLQS EDGATYYCQQ YDTFPTFGPG TKLDIKRTVA APSVFIFPPS EDQVKSGTVS VVCLLNNFYP R EASVKWKV ...文字列: ETDTLLLWVL LLWVPGSTGD ILLTQSPSSL SGSVGDRVTI TCRASQGINS YLNWYQQKPG KAPKLLIYFA NRLQSGVPSR FSGSGSGTE FTLTISSLQS EDGATYYCQQ YDTFPTFGPG TKLDIKRTVA APSVFIFPPS EDQVKSGTVS VVCLLNNFYP R EASVKWKV DGALKTGNSQ ESVTEQDSKD NTYSLSSTLT LSSTEYQSHK VYACEVTHQG LSSPVTKSFN RGEC |
-分子 #13: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 13 / コピー数: 36 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7t4g: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X