+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23587 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the elongation module of the bacillamide NRPS, BmdB, in complex with the oxidase, BmdC | |||||||||
 マップデータ マップデータ | EM map of the locally refined bacillamide NRPS, BmdB, complexed with the oxidase, BmdC | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Thermoactinomyces vulgaris (バクテリア) Thermoactinomyces vulgaris (バクテリア) | |||||||||
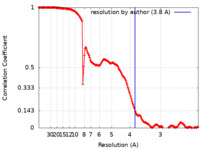
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Sharon I / Strauss M / Schmeing TM | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structures and function of a tailoring oxidase in complex with a nonribosomal peptide synthetase module. 著者: Camille Marie Fortinez / Kristjan Bloudoff / Connor Harrigan / Itai Sharon / Mike Strauss / T Martin Schmeing /  要旨: Nonribosomal peptide synthetases (NRPSs) are large modular enzymes that synthesize secondary metabolites and natural product therapeutics. Most NRPS biosynthetic pathways include an NRPS and ...Nonribosomal peptide synthetases (NRPSs) are large modular enzymes that synthesize secondary metabolites and natural product therapeutics. Most NRPS biosynthetic pathways include an NRPS and additional proteins that introduce chemical modifications before, during or after assembly-line synthesis. The bacillamide biosynthetic pathway is a common, three-protein system, with a decarboxylase that prepares an NRPS substrate, an NRPS, and an oxidase. Here, the pathway is reconstituted in vitro. The oxidase is shown to perform dehydrogenation of the thiazoline in the peptide intermediate while it is covalently attached to the NRPS, as the penultimate step in bacillamide D synthesis. Structural analysis of the oxidase reveals a dimeric, two-lobed architecture with a remnant RiPP recognition element and a dramatic wrapping loop. The oxidase forms a stable complex with the NRPS and dimerizes it. We visualized co-complexes of the oxidase bound to the elongation module of the NRPS using X-ray crystallography and cryo-EM. The three active sites (for adenylation, condensation/cyclization, and oxidation) form an elegant arc to facilitate substrate delivery. The structures enabled a proof-of-principle bioengineering experiment in which the BmdC oxidase domain is embedded into the NRPS. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23587.map.gz emd_23587.map.gz | 398.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23587-v30.xml emd-23587-v30.xml emd-23587.xml emd-23587.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23587_fsc.xml emd_23587_fsc.xml | 16.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23587.png emd_23587.png | 236.7 KB | ||
| マスクデータ |  emd_23587_msk_1.map emd_23587_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23587_half_map_1.map.gz emd_23587_half_map_1.map.gz emd_23587_half_map_2.map.gz emd_23587_half_map_2.map.gz | 391.9 MB 391.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23587 http://ftp.pdbj.org/pub/emdb/structures/EMD-23587 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23587 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23587 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23587.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23587.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map of the locally refined bacillamide NRPS, BmdB, complexed with the oxidase, BmdC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.14 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23587_msk_1.map emd_23587_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A of the locally refined bacillamide...
| ファイル | emd_23587_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of the locally refined bacillamide NRPS, BmdB, complexed with the oxidase, BmdC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B of the locally refined bacillamide...
| ファイル | emd_23587_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of the locally refined bacillamide NRPS, BmdB, complexed with the oxidase, BmdC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Elongation module of the bacillamide NRPS, BmdB, in complex with ...
| 全体 | 名称: Elongation module of the bacillamide NRPS, BmdB, in complex with the oxidase, BmdC |
|---|---|
| 要素 |
|
-超分子 #1: Elongation module of the bacillamide NRPS, BmdB, in complex with ...
| 超分子 | 名称: Elongation module of the bacillamide NRPS, BmdB, in complex with the oxidase, BmdC タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Thermoactinomyces vulgaris (バクテリア) Thermoactinomyces vulgaris (バクテリア) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: BmdC, Oxidase
| 分子 | 名称: BmdC, Oxidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermoactinomyces vulgaris (バクテリア) Thermoactinomyces vulgaris (バクテリア) |
| 分子量 | 理論値: 38.177254 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GAMGEQIFYW SPAKHWRMSD EGVVIGESTY TGMILEWFPE FYFFAQTGVT INRLLERFSS GSEKEANEIL ELLIQDRVLV EGILPPREV FSPQEGLFVN PYSEQIRYSK EALDYYVSEQ LNRTHAACRS TKIQLETSGA LPDIIQKRRS CRRFDMKTPV S FATFSNLL ...文字列: GAMGEQIFYW SPAKHWRMSD EGVVIGESTY TGMILEWFPE FYFFAQTGVT INRLLERFSS GSEKEANEIL ELLIQDRVLV EGILPPREV FSPQEGLFVN PYSEQIRYSK EALDYYVSEQ LNRTHAACRS TKIQLETSGA LPDIIQKRRS CRRFDMKTPV S FATFSNLL SSLKQRKEDK ILYNYASAGG LYPIDVFVYV KPRRVEGVKA GFYYFNPADH SLVLVNNIDQ VIKDDHELIN QD IFAQSAF SVYLVYNARA SMPKYGAAGY FYACIEAGII TATLNMVAED LNVGLCSIGH MNFEEIQTFL KLEDHQVILH AIE GGLKID GAAAENLYFQ |
-分子 #2: BmdB, bacillamide NRPS
| 分子 | 名称: BmdB, bacillamide NRPS / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermoactinomyces vulgaris (バクテリア) Thermoactinomyces vulgaris (バクテリア) |
| 分子量 | 理論値: 117.373578 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GAMEADLSEP FSLTEVQTAY MLGRNPQFEL SGISPQTYFE YETELDIARL SRSFQKVIQR HPMLRAVILP EGKQQILRDV PEYEIEVES LVSMPPEKQA ARLREERSRM IDHVFPLGQW PLFELKAFQL QEHTYLLCFR YDALLMDGAS MNLVGQDLMH Y YHQPDAQL ...文字列: GAMEADLSEP FSLTEVQTAY MLGRNPQFEL SGISPQTYFE YETELDIARL SRSFQKVIQR HPMLRAVILP EGKQQILRDV PEYEIEVES LVSMPPEKQA ARLREERSRM IDHVFPLGQW PLFELKAFQL QEHTYLLCFR YDALLMDGAS MNLVGQDLMH Y YHQPDAQL PPLSFTFQDY MHIYDDMKRG TEYETAKAYW TNKLPDFPPA PSLLLAKDPA EIGTPNFQSL TTIITKDKWL KL RRLAQDK QVTPSALLCT VYGEVLAFWS NQRRLAINLT VFNRYPVHDE VEQIVGDFTS LILLDMDMDQ KQPFFTKVEQ TQS TLLDGL EHRHYDGVEF IRDYTRYHQM RPKAVMPIVF TSMLAGAGAF AWEEIGSLRH IHARTPQVYL DNVVIEKNGE LLVS WNYVE ELFDAEVMES MFTQFVELLD QLVEQGDINP LRISQKDYAL IDQYNATAEP IPAATLHQLF IDQAQRTPDQ VAVVF EQEW LTYSELDQRS NQVARFLQSR GIGRGDRVGV LAKRQVETII NLMAVLKAGA AYVPIDPDHP YERQTYILEN SSCKIL LDS DLYETMEISS YADGDLTPVA EPEDTAYVIY TSGSTGRPKG VIITHQAASN TIQDINRKFE VNEEDRIIGI SSMCFDL SV YDIFGTLSAG ATLVMIRDPR DMRELVRTVE RRGITIWNTV PAIMDLALDH VGSHFENISL RLVLLSGDWI PLPLPAKI N RHFPVADVIS LGGATEASIW SIYWPIEQVE ANWKSIPYGK PLANQTYYVL NYDQKMCPVG VIGDLYIGGA GLAQGYLND DQKTKDAFIM HPEFGPIYKT GDCGRMRPEG YIEFLGRQDY QVKIQGYRVE LEEISHCLLT YPDVDQAVVI DQTDERGMKF LVGYVVAQQ EIDEKALRKH LMEHLPEYMI PAHLVHLEQL PLTPNGKLDR KALPVPKKQR NAEKFVAPQA GLEKILASVW Q EVLNVEQI GANDHFFALG GDSIKAIQVS ARLFVQGYHL DTKSLFEFPV LRDVARTIKK LAAAENLYFQ |
-分子 #3: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 109.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X