+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22990 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the H-lobe of yeast CKM | |||||||||
 マップデータ マップデータ | Structure of the H-lobe of yeast CKM | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mediator /  Transcription (転写 (生物学)) / Transcription (転写 (生物学)) /  Cdk8 / Cdk8 /  Med13 / Med13 /  Med12 / CycC / CDK / Med12 / CycC / CDK /  Argonaute (アルゴノート (タンパク質)) / Argonaute (アルゴノート (タンパク質)) /  RNA Polymerase II (RNAポリメラーゼII) / RNA Polymerase II (RNAポリメラーゼII) /  PIWI PIWI | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of transcription by galactose / CKM complex /  mediator complex / mediator complex /  transcription coactivator activity / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II transcription coactivator activity / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae S288C (パン酵母) / Saccharomyces cerevisiae S288C (パン酵母) /   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) | |||||||||
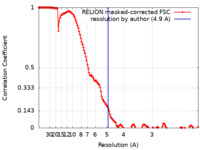
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.9 Å クライオ電子顕微鏡法 / 解像度: 4.9 Å | |||||||||
 データ登録者 データ登録者 | Li YC / Chao TC | |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Structure and noncanonical Cdk8 activation mechanism within an Argonaute-containing Mediator kinase module. 著者: Yi-Chuan Li / Ti-Chun Chao / Hee Jong Kim / Timothy Cholko / Shin-Fu Chen / Guojie Li / Laura Snyder / Kotaro Nakanishi / Chia-En Chang / Kenji Murakami / Benjamin A Garcia / Thomas G Boyer / Kuang-Lei Tsai /  要旨: The Cdk8 kinase module (CKM) in Mediator, comprising Med13, Med12, CycC, and Cdk8, regulates RNA polymerase II transcription through kinase-dependent and -independent functions. Numerous pathogenic ...The Cdk8 kinase module (CKM) in Mediator, comprising Med13, Med12, CycC, and Cdk8, regulates RNA polymerase II transcription through kinase-dependent and -independent functions. Numerous pathogenic mutations causative for neurodevelopmental disorders and cancer congregate in CKM subunits. However, the structure of the intact CKM and the mechanism by which Cdk8 is non-canonically activated and functionally affected by oncogenic CKM alterations are poorly understood. Here, we report a cryo-electron microscopy structure of CKM that redefines prior CKM structural models and explains the mechanism of Med12-dependent Cdk8 activation. Med12 interacts extensively with CycC and activates Cdk8 by stabilizing its activation (T-)loop through conserved Med12 residues recurrently mutated in human tumors. Unexpectedly, Med13 has a characteristic Argonaute-like bi-lobal architecture. These findings not only provide a structural basis for understanding CKM function and pathological dysfunction, but also further impute a previously unknown regulatory mechanism of Mediator in transcriptional modulation through its Med13 Argonaute-like features. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22990.map.gz emd_22990.map.gz | 9.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22990-v30.xml emd-22990-v30.xml emd-22990.xml emd-22990.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22990_fsc.xml emd_22990_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22990.png emd_22990.png | 46.5 KB | ||
| Filedesc metadata |  emd-22990.cif.gz emd-22990.cif.gz | 6.2 KB | ||
| その他 |  emd_22990_half_map_1.map.gz emd_22990_half_map_1.map.gz emd_22990_half_map_2.map.gz emd_22990_half_map_2.map.gz | 171 MB 171 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22990 http://ftp.pdbj.org/pub/emdb/structures/EMD-22990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22990 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22990.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22990.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the H-lobe of yeast CKM | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half-volume 1
| ファイル | emd_22990_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-volume 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-volume 2
| ファイル | emd_22990_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-volume 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : yeast CDK8 complex
| 全体 | 名称: yeast CDK8 complex Cyclin-dependent kinase 8 Cyclin-dependent kinase 8 |
|---|---|
| 要素 |
|
-超分子 #1: yeast CDK8 complex
| 超分子 | 名称: yeast CDK8 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母) |
| 分子量 | 理論値: 430 KDa |
-分子 #1: Mediator of RNA polymerase II transcription subunit 12
| 分子 | 名称: Mediator of RNA polymerase II transcription subunit 12 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 167.049812 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母) |
| 配列 | 文字列: MNNGSGRYLL TPPDDLHPYV PSSKPQEQVY PDFKPWEHTA AEDQILANFV AKGFYHTPMV NFESISARSS VHESLVTQSN ILSQQFDKI IKIREDHINK IPSNSTTTLH GPGFQLPNRI TLTDHRKETW LHELSSSHTS LVKIGKFIPH GLKRRQVIEQ C YLKFIPLK ...文字列: MNNGSGRYLL TPPDDLHPYV PSSKPQEQVY PDFKPWEHTA AEDQILANFV AKGFYHTPMV NFESISARSS VHESLVTQSN ILSQQFDKI IKIREDHINK IPSNSTTTLH GPGFQLPNRI TLTDHRKETW LHELSSSHTS LVKIGKFIPH GLKRRQVIEQ C YLKFIPLK RAIWLIKCCY FIEWKSNHKK KRSNAAGADD AISMHLLKDW TDTFVYILEK LIFDMTNHYN DSQQLRTWKR QI SYFLKLL GNCYSLRLIN KEIFHHWLVE FINKMENFEF LPLSLHILMI FWNDICQIDT NAPVAATITS SQKEPFFLVT KIT DMLLHK YYIVSSSKSM INDENYIIND IKKNNKIKLN ILKILSSLIL KIFQEQSLEV FIFPTSNWEI YKPLLFEIVS NADT NQNSD MKKKLELISY RNESLKNNSS IRNVIMSASN ANDFQLTIVT CKQFPKLSCI QLNCIDTQFT KLLDDNPTEF DWPTY VDQN PLTMHKIIQL ILWSIHPSRQ FDHYESNQLV AKLLLLRINS TDEDLHEFQI EDAIWSLVFQ LAKNFSAQKR VVSYMM PSL YRLLNILITY GIIKVPTYIR KLISSGLLYL QDSNDKFVHV QLLINLKISP LMKSQYNMVL RNVMEYDVKF YEIFNFD QL VEITEQIKMR ILSNDITNLQ LSKTPLSIKI MVAEWYLSHL CSGILSSVNR TVLLKIFKIF CIDLEVFHHF FKWIEFIV Y HQLLSDIESL EALMDILLCY QKLFSQFIND HILFTKTFIF IYKKVLKEKD VPAYNVTSFM PFWKFFMKNF PFVLKVDND LRIELQSVYN DEKLKTEKLK NDKSEVLKVY SMINNSNQAV GQTWNFPEVF QVNIRFLLHN SEIIDTNTSK QFQKARNNVM LLIATNLKE YNKFMSIFLK RKDFTNKNLI QLISLKLLTF EVTQNVLGLE YIIRLLPINL ENNDGSYGLF LKYHKEQFIK S NFEKILLT CYELEKKYHG NECEINYYEI LLKILITYGS SPKLLATSTK IIMLLLNDSV ENSSNILEDI LYYSTCPSET DL NDIPLGS GQPDNDTVVT NDDKSDDDDH TVDEIDHVEY YVMMDFANLW VFQAFTCFCI KKIMENNEPA MAMEDLKNFI FQI IEITNS NDLCSQIFDQ LKDMQTIEMI TQIVEKDFCT SCLQNNNQKI DDNYIVVVIE IITSLSMRFQ RETSGMIVIS MENY HLLIK IIRQLSELNE GNLSKREIQI DAVLKIFSFH QDSIFQRIIA DLSADKPTSP FIDSICKLFD KISFNLRLKL FLYEI LSSL KSFAIYSSTI DAPAFHTSGK VELPKKLLNL PPFQVSSFVK ETKLHSGDYG EEEDADQEES FSLNLGIGIV EIAHEN EQK WLIYDKKDHK YVCTFSMEPY HFISNYNTKY TDDMATGSND TTAFNDSCVN LSLFDARFER KNPH UniProtKB: Mediator of RNA polymerase II transcription subunit 12 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 25 mM Hepes pH 7.4, 200 mM NaCl, and 0.005% NP-40 |
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 実像数: 15075 / 平均電子線量: 65.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X