+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22191 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
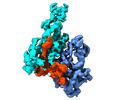
| タイトル | Yeast TFIIK (Kin28/Ccl1/Tfb3) Complex | |||||||||
 マップデータ マップデータ | main map of TFIIK | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / : / positive regulation of Atg1/ULK1 kinase complex assembly / : / : / transcription factor TFIIK complex / cellular response to nitrogen starvation / transcription factor TFIIH holo complex / cyclin-dependent protein serine/threonine kinase activator activity ...: / : / : / positive regulation of Atg1/ULK1 kinase complex assembly / : / : / transcription factor TFIIK complex / cellular response to nitrogen starvation / transcription factor TFIIH holo complex / cyclin-dependent protein serine/threonine kinase activator activity / [RNA-polymerase]-subunit kinase / cyclin-dependent protein serine/threonine kinase regulator activity / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / mRNA Capping / Formation of TC-NER Pre-Incision Complex / RNA polymerase II transcribes snRNA genes / RNA Polymerase I Promoter Escape / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / RNA Polymerase II Pre-transcription Events / Dual incision in TC-NER / regulation of cyclin-dependent protein serine/threonine kinase activity / 7-methylguanosine mRNA capping / Gap-filling DNA repair synthesis and ligation in TC-NER / transcription by RNA polymerase I / cyclin-dependent protein serine/threonine kinase activity / positive regulation of autophagy / RNA polymerase II CTD heptapeptide repeat kinase activity /  transcription initiation at RNA polymerase II promoter / nucleotide-excision repair / transcription by RNA polymerase II / transcription initiation at RNA polymerase II promoter / nucleotide-excision repair / transcription by RNA polymerase II /  protein kinase activity / protein kinase activity /  細胞周期 / 細胞周期 /  細胞分裂 / 細胞分裂 /  protein phosphorylation / protein phosphorylation /  DNA修復 / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA修復 / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  ミトコンドリア / ミトコンドリア /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) / Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) /   Baker's yeast (パン酵母) Baker's yeast (パン酵母) | |||||||||
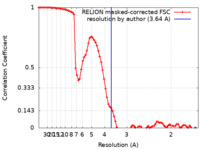
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.64 Å クライオ電子顕微鏡法 / 解像度: 3.64 Å | |||||||||
 データ登録者 データ登録者 | van Eeuwen T / Murakami K / Li T / Tsai KL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Structure of TFIIK for phosphorylation of CTD of RNA polymerase II. 著者: Trevor van Eeuwen / Tao Li / Hee Jong Kim / Jose J Gorbea Colón / Mitchell I Parker / Roland L Dunbrack / Benjamin A Garcia / Kuang-Lei Tsai / Kenji Murakami /  要旨: During transcription initiation, the general transcription factor TFIIH marks RNA polymerase II by phosphorylating Ser5 of the carboxyl-terminal domain (CTD) of Rpb1, which is followed by extensive ...During transcription initiation, the general transcription factor TFIIH marks RNA polymerase II by phosphorylating Ser5 of the carboxyl-terminal domain (CTD) of Rpb1, which is followed by extensive modifications coupled to transcription elongation, mRNA processing, and histone dynamics. We have determined a 3.5-Å resolution cryo-electron microscopy (cryo-EM) structure of the TFIIH kinase module (TFIIK in yeast), which is composed of Kin28, Ccl1, and Tfb3, yeast homologs of CDK7, cyclin H, and MAT1, respectively. The carboxyl-terminal region of Tfb3 was lying at the edge of catalytic cleft of Kin28, where a conserved Tfb3 helix served to stabilize the activation loop in its active conformation. By combining the structure of TFIIK with the previous cryo-EM structure of the preinitiation complex, we extend the previously proposed model of the CTD path to the active site of TFIIK. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22191.map.gz emd_22191.map.gz | 37.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22191-v30.xml emd-22191-v30.xml emd-22191.xml emd-22191.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22191_fsc.xml emd_22191_fsc.xml | 8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22191.png emd_22191.png | 70.1 KB | ||
| その他 |  emd_22191_half_map_1.map.gz emd_22191_half_map_1.map.gz emd_22191_half_map_2.map.gz emd_22191_half_map_2.map.gz | 33.1 MB 33.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22191 http://ftp.pdbj.org/pub/emdb/structures/EMD-22191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22191 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22191.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22191.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map of TFIIK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half-volume 2
| ファイル | emd_22191_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-volume 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-volume 2
| ファイル | emd_22191_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-volume 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae.
| 全体 | 名称: Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae. |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae.
| 超分子 | 名称: Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: the yeast Cdk7 complex, that phosphorylates the RNA pol II C-terminal domain (CTD) in transcription initiation. |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
| 分子量 | 実験値: 73 KDa |
-分子 #1: RNA polymerase II transcription factor B subunit 3
| 分子 | 名称: RNA polymerase II transcription factor B subunit 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 7.19103 KDa |
| 配列 | 文字列: PFNGDREAHP PFTLKGSVYN DPFIKDLEHR KEFIASGFNT NYAYERVLTE AFMGLGCVIS EEL |
-分子 #2: Serine/threonine-protein kinase KIN28
| 分子 | 名称: Serine/threonine-protein kinase KIN28 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: [RNA-polymerase]-subunit kinase |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 34.725043 KDa |
| 配列 | 文字列: VNMEYTKEKK VGEGTYAVVY LGCQHSTGRK IAIKEIKTSE FKDGLDMSAI REVKYLQEMQ HPNVIELIDI FMAYDNLNLV LEFLPTDLE VVIKDKSILF TPADIKAWML MTLRGVYHCH RNFILHRDLK PNNLLFSPDG QIKVADFGLA RAIPAPHEIL (TPO) ...文字列: VNMEYTKEKK VGEGTYAVVY LGCQHSTGRK IAIKEIKTSE FKDGLDMSAI REVKYLQEMQ HPNVIELIDI FMAYDNLNLV LEFLPTDLE VVIKDKSILF TPADIKAWML MTLRGVYHCH RNFILHRDLK PNNLLFSPDG QIKVADFGLA RAIPAPHEIL (TPO)SNVVTRWY RAPELLFGAK HYTSAIDIWS VGVIFAELML RIPYLPGQND VDQMEVTFRA LGTPTDRDWP EVSSFMT YN KLQIYPPPSR DELRKRFIAA SEYALDFMCG MLTMNPQKRW TAVQCLESDY FKELPPPSDP SSIK |
-分子 #3: Cyclin CCL1
| 分子 | 名称: Cyclin CCL1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 37.552715 KDa |
| 配列 | 文字列: DLYRHSSQYR MWSYTKDQLQ EKRVDTNARA IAYIEENLLK FREAHNLTEE EIKVLEAKAI PLTMEEELDL VNFYAKKVQV IAQHLNLPT EVVATAISFF RRFFLENSVM QIDPKSIVHT TIFLACKSEN YFISVDSFAQ KAKSTRDSVL KFEFKLLESL K FSLLNHHP ...文字列: DLYRHSSQYR MWSYTKDQLQ EKRVDTNARA IAYIEENLLK FREAHNLTEE EIKVLEAKAI PLTMEEELDL VNFYAKKVQV IAQHLNLPT EVVATAISFF RRFFLENSVM QIDPKSIVHT TIFLACKSEN YFISVDSFAQ KAKSTRDSVL KFEFKLLESL K FSLLNHHP YKPLHGFFLD IQNVLYGKVD LNYMGQIYDR CKKRITAALL TDVVYFYTPP QITLATLLIE DEALVTRYLE TK FPSREGS QESVPGNEKE EPQNDASTTE KNKEKSTESE EYSIDSAKLL TIIRECKSII EDCKPPSTEE AKKIAAKNYY CQN PSTL |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: ALUMINUM FLUORIDE
| 分子 | 名称: ALUMINUM FLUORIDE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: AF3 |
|---|---|
| 分子量 | 理論値: 83.977 Da |
| Chemical component information |  ChemComp-AF3: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.08 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 0 % / チャンバー内温度: 293 K / 装置: LEICA EM CPC 詳細: blotted for 2 seconds with Whatman 41 ashless filter paper. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4620 / 平均露光時間: 2.24 sec. / 平均電子線量: 45.0 e/Å2 詳細: Images collected in super-resolution mode. Movies were 35 frames. Imaging 1 image/hole, image shift between 4 holes. Focus once per image shift |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























 Z
Z Y
Y X
X