+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22074 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of NLRP1-DPP9 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  NLRP1 / NLRP1 /  DPP9 / DPP9 /  inflammasome (インフラマソーム) / Val-boroPro (VbP) / talabostat / inflammasome (インフラマソーム) / Val-boroPro (VbP) / talabostat /  innate immunity (自然免疫系) / innate immunity (自然免疫系) /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NLRP1 inflammasome complex assembly / cysteine-type endopeptidase activator activity / NLRP1 inflammasome complex / canonical inflammasome complex / The NLRP1 inflammasome /  DPP-4 / self proteolysis / dipeptidyl-peptidase activity / negative regulation of programmed cell death / DPP-4 / self proteolysis / dipeptidyl-peptidase activity / negative regulation of programmed cell death /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 ...NLRP1 inflammasome complex assembly / cysteine-type endopeptidase activator activity / NLRP1 inflammasome complex / canonical inflammasome complex / The NLRP1 inflammasome / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 ...NLRP1 inflammasome complex assembly / cysteine-type endopeptidase activator activity / NLRP1 inflammasome complex / canonical inflammasome complex / The NLRP1 inflammasome /  DPP-4 / self proteolysis / dipeptidyl-peptidase activity / negative regulation of programmed cell death / DPP-4 / self proteolysis / dipeptidyl-peptidase activity / negative regulation of programmed cell death /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 /  pattern recognition receptor activity / pattern recognition receptor signaling pathway / cellular response to UV-B / stress-activated protein kinase signaling cascade / pyroptotic inflammatory response / cell leading edge / cysteine-type endopeptidase activator activity involved in apoptotic process / antiviral innate immune response / response to muramyl dipeptide / signaling adaptor activity / pattern recognition receptor activity / pattern recognition receptor signaling pathway / cellular response to UV-B / stress-activated protein kinase signaling cascade / pyroptotic inflammatory response / cell leading edge / cysteine-type endopeptidase activator activity involved in apoptotic process / antiviral innate immune response / response to muramyl dipeptide / signaling adaptor activity /  aminopeptidase activity / serine-type peptidase activity / molecular condensate scaffold activity / positive regulation of interleukin-1 beta production / aminopeptidase activity / serine-type peptidase activity / molecular condensate scaffold activity / positive regulation of interleukin-1 beta production /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / protein homooligomerization / positive regulation of inflammatory response / activation of cysteine-type endopeptidase activity involved in apoptotic process / : / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / protein homooligomerization / positive regulation of inflammatory response / activation of cysteine-type endopeptidase activity involved in apoptotic process / : /  double-stranded RNA binding / double-stranded RNA binding /  peptidase activity / peptidase activity /  regulation of inflammatory response / regulation of inflammatory response /  double-stranded DNA binding / neuron apoptotic process / regulation of apoptotic process / defense response to virus / double-stranded DNA binding / neuron apoptotic process / regulation of apoptotic process / defense response to virus /  微小管 / defense response to bacterium / protein domain specific binding / apoptotic process / 微小管 / defense response to bacterium / protein domain specific binding / apoptotic process /  核小体 / 核小体 /  enzyme binding / enzyme binding /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  核質 / 核質 /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
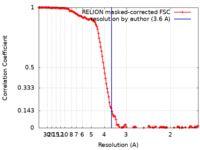
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Hollingsworth LR / Sharif H / Griswold AR / Fontana P / Mintseris J / Dagbay KB / Paulo JA / Gygi SP / Bachovchin DA / Wu H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: DPP9 sequesters the C terminus of NLRP1 to repress inflammasome activation. 著者: L Robert Hollingsworth / Humayun Sharif / Andrew R Griswold / Pietro Fontana / Julian Mintseris / Kevin B Dagbay / Joao A Paulo / Steven P Gygi / Daniel A Bachovchin / Hao Wu /  要旨: Nucleotide-binding domain and leucine-rich repeat pyrin-domain containing protein 1 (NLRP1) is an inflammasome sensor that mediates the activation of caspase-1 to induce cytokine maturation and ...Nucleotide-binding domain and leucine-rich repeat pyrin-domain containing protein 1 (NLRP1) is an inflammasome sensor that mediates the activation of caspase-1 to induce cytokine maturation and pyroptosis. Gain-of-function mutations of NLRP1 cause severe inflammatory diseases of the skin. NLRP1 contains a function-to-find domain that auto-proteolyses into noncovalently associated subdomains, and proteasomal degradation of the repressive N-terminal fragment of NLRP1 releases its inflammatory C-terminal fragment (NLRP1 CT). Cytosolic dipeptidyl peptidases 8 and 9 (hereafter, DPP8/DPP9) both interact with NLRP1, and small-molecule inhibitors of DPP8/DPP9 activate NLRP1 by mechanisms that are currently unclear. Here we report cryo-electron microscopy structures of the human NLRP1-DPP9 complex alone and with Val-boroPro (VbP), an inhibitor of DPP8/DPP9. The structures reveal a ternary complex that comprises DPP9, full-length NLRP1 and the NLRPT CT. The binding of the NLRP1 CT to DPP9 requires full-length NLRP1, which suggests that NLRP1 activation is regulated by the ratio of NLRP1 CT to full-length NLRP1. Activation of the inflammasome by ectopic expression of the NLRP1 CT is consistently rescued by co-expression of autoproteolysis-deficient full-length NLRP1. The N terminus of the NLRP1 CT inserts into the DPP9 active site, and VbP disrupts this interaction. Thus, VbP weakens the NLRP1-DPP9 interaction and accelerates degradation of the N-terminal fragment to induce inflammasome activation. Overall, these data demonstrate that DPP9 quenches low levels of NLRP1 CT and thus serves as a checkpoint for activation of the NLRP1 inflammasome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22074.map.gz emd_22074.map.gz | 15.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22074-v30.xml emd-22074-v30.xml emd-22074.xml emd-22074.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22074_fsc.xml emd_22074_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22074.png emd_22074.png | 161.7 KB | ||
| Filedesc metadata |  emd-22074.cif.gz emd-22074.cif.gz | 7.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22074 http://ftp.pdbj.org/pub/emdb/structures/EMD-22074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22074 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6x6aMC  6x6cC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10594 (タイトル: Human NLRP1-DPP9 complex / Data size: 2.3 TB EMPIAR-10594 (タイトル: Human NLRP1-DPP9 complex / Data size: 2.3 TBData #1: Unaligned multi frame micographs of NLRP1-DPP9-Apo-noTILT dataset [micrographs - multiframe] Data #2: Unaligned multi frame micographs of NLRP1-DPP9-Apo-TILT dataset [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22074.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22074.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DPP9-NLRP1 complex
| 全体 | 名称: DPP9-NLRP1 complex |
|---|---|
| 要素 |
|
-超分子 #1: DPP9-NLRP1 complex
| 超分子 | 名称: DPP9-NLRP1 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Dipeptidyl peptidase 9
| 分子 | 名称: Dipeptidyl peptidase 9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  DPP-4 DPP-4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 98.38432 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MATTGTPTAD RGDAAATDDP AARFQVQKHS WDGLRSIIHG SRKYSGLIVN KAPHDFQFVQ KTDESGPHSH RLYYLGMPYG SRENSLLYS EIPKKVRKEA LLLLSWKQML DHFQATPHHG VYSREEELLR ERKRLGVFGI TSYDFHSESG LFLFQASNSL F HCRDGGKN ...文字列: MATTGTPTAD RGDAAATDDP AARFQVQKHS WDGLRSIIHG SRKYSGLIVN KAPHDFQFVQ KTDESGPHSH RLYYLGMPYG SRENSLLYS EIPKKVRKEA LLLLSWKQML DHFQATPHHG VYSREEELLR ERKRLGVFGI TSYDFHSESG LFLFQASNSL F HCRDGGKN GFMVSPMKPL EIKTQCSGPR MDPKICPADP AFFSFINNSD LWVANIETGE ERRLTFCHQG LSNVLDDPKS AG VATFVIQ EEFDRFTGYW WCPTASWEGS EGLKTLRILY EEVDESEVEV IHVPSPALEE RKTDSYRYPR TGSKNPKIAL KLA EFQTDS QGKIVSTQEK ELVQPFSSLF PKVEYIARAG WTRDGKYAWA MFLDRPQQWL QLVLLPPALF IPSTENEEQR LASA RAVPR NVQPYVVYEE VTNVWINVHD IFYPFPQSEG EDELCFLRAN ECKTGFCHLY KVTAVLKSQG YDWSEPFSPG EDEFK CPIK EEIALTSGEW EVLARHGSKI WVNEETKLVY FQGTKDTPLE HHLYVVSYEA AGEIVRLTTP GFSHSCSMSQ NFDMFV SHY SSVSTPPCVH VYKLSGPDDD PLHKQPRFWA SMMEAASCPP DYVPPEIFHF HTRSDVRLYG MIYKPHALQP GKKHPTV LF VYGGPQVQLV NNSFKGIKYL RLNTLASLGY AVVVIDGRGS CQRGLRFEGA LKNQMGQVEI EDQVEGLQFV AEKYGFID L SRVAIHGWSY GGFLSLMGLI HKPQVFKVAI AGAPVTVWMA YDTGYTERYM DVPENNQHGY EAGSVALHVE KLPNEPNRL LILHGFLDEN VHFFHTNFLV SQLIRAGKPY QLQIYPNERH SIRCPESGEH YEVTLLHFLQ EYL UniProtKB:  Dipeptidyl peptidase 9 Dipeptidyl peptidase 9 |
-分子 #2: NACHT, LRR and PYD domains-containing protein 1
| 分子 | 名称: NACHT, LRR and PYD domains-containing protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 136.327344 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAGGAWGRLA CYLEFLKKEE LKEFQLLLAN KAHSRSSSGE TPAQPEKTSG MEVASYLVAQ YGEQRAWDLA LHTWEQMGLR SLCAQAQEG AGHSPSFPYS PSEPHLGSPS QPTSTAVLMP WIHELPAGCT QGSERRVLRQ LPDTSGRRWR EISASLLYQA L PSSPDHES ...文字列: MAGGAWGRLA CYLEFLKKEE LKEFQLLLAN KAHSRSSSGE TPAQPEKTSG MEVASYLVAQ YGEQRAWDLA LHTWEQMGLR SLCAQAQEG AGHSPSFPYS PSEPHLGSPS QPTSTAVLMP WIHELPAGCT QGSERRVLRQ LPDTSGRRWR EISASLLYQA L PSSPDHES PSQESPNAPT STAVLGSWGS PPQPSLAPRE QEAPGTQWPL DETSGIYYTE IREREREKSE KGRPPWAAVV GT PPQAHTS LQPHHHPWEP SVRESLCSTW PWKNEDFNQK FTQLLLLQRP HPRSQDPLVK RSWPDYVEEN RGHLIEIRDL FGP GLDTQE PRIVILQGAA GIGKSTLARQ VKEAWGRGQL YGDRFQHVFY FSCRELAQSK VVSLAELIGK DGTATPAPIR QILS RPERL LFILDGVDEP GWVLQEPSSE LCLHWSQPQP ADALLGSLLG KTILPEASFL ITARTTALQN LIPSLEQARW VEVLG FSES SRKEYFYRYF TDERQAIRAF RLVKSNKELW ALCLVPWVSW LACTCLMQQM KRKEKLTLTS KTTTTLCLHY LAQALQ AQP LGPQLRDLCS LAAEGIWQKK TLFSPDDLRK HGLDGAIIST FLKMGILQEH PIPLSYSFIH LCFQEFFAAM SYVLEDE KG RGKHSNCIID LEKTLEAYGI HGLFGASTTR FLLGLLSDEG EREMENIFHC RLSQGRNLMQ WVPSLQLLLQ PHSLESLH C LYETRNKTFL TQVMAHFEEM GMCVETDMEL LVCTFCIKFS RHVKKLQLIE GRQHRSTWSP TMVVLFRWVP VTDAYWQIL FSVLKVTRNL KELDLSGNSL SHSAVKSLCK TLRRPRCLLE TLRLAGCGLT AEDCKDLAFG LRANQTLTEL DLSFNVLTDA GAKHLCQRL RQPSCKLQRL QLVSCGLTSD CCQDLASVLS ASPSLKELDL QQNNLDDVGV RLLCEGLRHP ACKLIRLGLD Q TTLSDEMR QELRALEQEK PQLLIFSRRK PSVMTPTEGL DTGEMSNSTS SLKRQRLGSE RAASHVAQAN LKLLDVSKIF PI AEIAEES SPEVVPVELL CVPSPASQGD LHTKPLGTDD DFWGPTGPVA TEVVDKEKNL YRVHFPVAGS YRWPNTGLCF VMR EAVTVE IEFCVWDQFL GEINPQHSWM VAGPLLDIKA EPGAVEAVHL PHFVALQGGH VDTSLFQMAH FKEEGMLLEK PARV ELHHI VLENPSF UniProtKB: NACHT, LRR and PYD domains-containing protein 1 |
-分子 #3: NACHT, LRR and PYD domains-containing protein 1
| 分子 | 名称: NACHT, LRR and PYD domains-containing protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.76074 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SPLGVLLKMI HNALRFIPVT SVVLLYHRVH PEEVTFHLYL IPSDCSIRKA IDDLEMKFQF VRIHKPPPLT PLYMGCRYTV SGSGSGMLE ILPKELELCY RSPGEDQLFS EFYVGHLGSG IRLQVKDKKD ETLVWEALVK PGDLMPATTL IPPARIAVPS P LDAPQLLH ...文字列: SPLGVLLKMI HNALRFIPVT SVVLLYHRVH PEEVTFHLYL IPSDCSIRKA IDDLEMKFQF VRIHKPPPLT PLYMGCRYTV SGSGSGMLE ILPKELELCY RSPGEDQLFS EFYVGHLGSG IRLQVKDKKD ETLVWEALVK PGDLMPATTL IPPARIAVPS P LDAPQLLH FVDQYREQLI ARVTSVEVVL DKLHGQVLSQ EQYERVLAEN TRPSQMRKLF SLSQSWDRKC KDGLYQALKE TH PHLIMEL WEKGSKKGLL PLSS UniProtKB: NACHT, LRR and PYD domains-containing protein 1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 25 mM Tris pH 7.5, 150 mM NaCl, 1 mM TCEP |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源: OTHER |
| 電子光学系 | 倍率(補正後): 10500 / 照射モード: SPOT SCAN / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): -1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #0 - 撮影したグリッド数: 7 / #0 - 実像数: 7840 / #0 - 平均露光時間: 1.8 sec. / #0 - 平均電子線量: 67.54 e/Å2 / #0 - 詳細: stage tilt 0 degrees / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #1 - 撮影したグリッド数: 4 / #1 - 実像数: 1916 / #1 - 平均露光時間: 2.6 sec. / #1 - 平均電子線量: 67.6 e/Å2 / #1 - 詳細: stage tilt 37 degrees |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー