+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20828 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
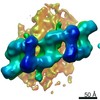
| タイトル | Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol | |||||||||
 マップデータ マップデータ | Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Watanabe R / Villa E | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: The In Situ Structure of Parkinson's Disease-Linked LRRK2. 著者: Reika Watanabe / Robert Buschauer / Jan Böhning / Martina Audagnotto / Keren Lasker / Tsan-Wen Lu / Daniela Boassa / Susan Taylor / Elizabeth Villa /  要旨: Mutations in leucine-rich repeat kinase 2 (LRRK2) are the most frequent cause of familial Parkinson's disease. LRRK2 is a multi-domain protein containing a kinase and GTPase. Using correlative light ...Mutations in leucine-rich repeat kinase 2 (LRRK2) are the most frequent cause of familial Parkinson's disease. LRRK2 is a multi-domain protein containing a kinase and GTPase. Using correlative light and electron microscopy, in situ cryo-electron tomography, and subtomogram analysis, we reveal a 14-Å structure of LRRK2 bearing a pathogenic mutation that oligomerizes as a right-handed double helix around microtubules, which are left-handed. Using integrative modeling, we determine the architecture of LRRK2, showing that the GTPase and kinase are in close proximity, with the GTPase closer to the microtubule surface, whereas the kinase is exposed to the cytoplasm. We identify two oligomerization interfaces mediated by non-catalytic domains. Mutation of one of these abolishes LRRK2 microtubule-association. Our work demonstrates the power of cryo-electron tomography to generate models of previously unsolved structures in their cellular environment. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20828.map.gz emd_20828.map.gz | 129.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20828-v30.xml emd-20828-v30.xml emd-20828.xml emd-20828.xml | 8.6 KB 8.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20828.png emd_20828.png | 141.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20828 http://ftp.pdbj.org/pub/emdb/structures/EMD-20828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20828 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6xr4C C: 同じ文献を引用 ( |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10377 (タイトル: Frame-aligned tilt series of cryo-FIB-milled HEK293 cells treated with taxol EMPIAR-10377 (タイトル: Frame-aligned tilt series of cryo-FIB-milled HEK293 cells treated with taxolData size: 2.1 Data #1: Frame-aligned tilt series of cryo-FIB-milled HEK293 cells treated with taxol [tilt series]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20828.map.gz / 形式: CCP4 / 大きさ: 158.9 MB / タイプ: IMAGE STORED AS SIGNED BYTE ダウンロード / ファイル: emd_20828.map.gz / 形式: CCP4 / 大きさ: 158.9 MB / タイプ: IMAGE STORED AS SIGNED BYTE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 10.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol
| 全体 | 名称: Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol
| 超分子 | 名称: Cryo-electron tomogram of FIB-milled HEK293 cells treated with Taxol タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 細胞: HEK293 Homo sapiens (ヒト) / 細胞: HEK293 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE-PROPANE |
| 切片作成 | 集束イオンビーム - 装置: OTHER / 集束イオンビーム - イオン: OTHER / 集束イオンビーム - 電圧: 30 kV / 集束イオンビーム - 電流: 0.001 nA / 集束イオンビーム - 時間: 50 sec. / 集束イオンビーム - 温度: 70 K / 集束イオンビーム - Initial thickness: 4000 nm / 集束イオンビーム - 最終 厚さ: 80 nm 集束イオンビーム - 詳細: The value given for _emd_sectioning_focused_ion_beam.instrument is Aquilos. This is not in a list of allowed values set(['DB235', 'OTHER']) so OTHER is written into the XML file. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 3.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / ソフトウェア - 名称:  IMOD / 使用した粒子像数: 39 IMOD / 使用した粒子像数: 39 |
|---|
 ムービー
ムービー コントローラー
コントローラー