+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1949 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human dynamin 1 deltaPRD polymer stabilized with GMPPCP | |||||||||
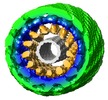
 マップデータ マップデータ | Three-dimensional volume of deltaPRD human dynamin 1 polymer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  dynamin / dynamin /  endocytosis (エンドサイトーシス) / GTP hydrolysis / membrane remodeling endocytosis (エンドサイトーシス) / GTP hydrolysis / membrane remodeling | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報clathrin coat assembly involved in endocytosis / vesicle scission / synaptic vesicle budding from presynaptic endocytic zone membrane / presynaptic endocytic zone membrane / interleukin-27-mediated signaling pathway /  dynamin GTPase / dynamin GTPase /  クロム親和性細胞 / regulation of vesicle size / photoreceptor ribbon synapse / Toll Like Receptor 4 (TLR4) Cascade ...clathrin coat assembly involved in endocytosis / vesicle scission / synaptic vesicle budding from presynaptic endocytic zone membrane / presynaptic endocytic zone membrane / interleukin-27-mediated signaling pathway / クロム親和性細胞 / regulation of vesicle size / photoreceptor ribbon synapse / Toll Like Receptor 4 (TLR4) Cascade ...clathrin coat assembly involved in endocytosis / vesicle scission / synaptic vesicle budding from presynaptic endocytic zone membrane / presynaptic endocytic zone membrane / interleukin-27-mediated signaling pathway /  dynamin GTPase / dynamin GTPase /  クロム親和性細胞 / regulation of vesicle size / photoreceptor ribbon synapse / Toll Like Receptor 4 (TLR4) Cascade / Retrograde neurotrophin signalling / Formation of annular gap junctions / endosome organization / Gap junction degradation / membrane coat / response to type I interferon / negative regulation of viral genome replication / Recycling pathway of L1 / phosphatidylinositol-3,4,5-trisphosphate binding / antiviral innate immune response / synaptic vesicle endocytosis / endocytic vesicle / EPH-ephrin mediated repulsion of cells / クロム親和性細胞 / regulation of vesicle size / photoreceptor ribbon synapse / Toll Like Receptor 4 (TLR4) Cascade / Retrograde neurotrophin signalling / Formation of annular gap junctions / endosome organization / Gap junction degradation / membrane coat / response to type I interferon / negative regulation of viral genome replication / Recycling pathway of L1 / phosphatidylinositol-3,4,5-trisphosphate binding / antiviral innate immune response / synaptic vesicle endocytosis / endocytic vesicle / EPH-ephrin mediated repulsion of cells /  クラスリン / クラスリン /  phosphatidylinositol-4,5-bisphosphate binding / MHC class II antigen presentation / photoreceptor inner segment / phosphatidylinositol-4,5-bisphosphate binding / MHC class II antigen presentation / photoreceptor inner segment /  receptor-mediated endocytosis / cell projection / response to virus / modulation of chemical synaptic transmission / defense response / protein homooligomerization / receptor-mediated endocytosis / cell projection / response to virus / modulation of chemical synaptic transmission / defense response / protein homooligomerization /  receptor internalization / ISG15 antiviral mechanism / receptor internalization / ISG15 antiviral mechanism /  エンドサイトーシス / GDP binding / : / Interferon alpha/beta signaling / presynapse / エンドサイトーシス / GDP binding / : / Interferon alpha/beta signaling / presynapse /  Clathrin-mediated endocytosis / Clathrin-mediated endocytosis /  microtubule binding / protein homotetramerization / defense response to virus / microtubule binding / protein homotetramerization / defense response to virus /  核膜 / 核膜 /  微小管 / 微小管 /  自然免疫系 / 自然免疫系 /  GTPase activity / glutamatergic synapse / apoptotic process / endoplasmic reticulum membrane / GTP binding / GTPase activity / glutamatergic synapse / apoptotic process / endoplasmic reticulum membrane / GTP binding /  protein kinase binding / perinuclear region of cytoplasm / protein kinase binding / perinuclear region of cytoplasm /  シグナル伝達 / protein homodimerization activity / シグナル伝達 / protein homodimerization activity /  RNA binding / extracellular exosome / RNA binding / extracellular exosome /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 12.2 Å クライオ電子顕微鏡法 / 解像度: 12.2 Å | |||||||||
 データ登録者 データ登録者 | Chappie JS / Mears JA / Fang S / Leonard M / Schmid SL / Milligan RA / Hinshaw JE / Dyda F | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2011 ジャーナル: Cell / 年: 2011タイトル: A pseudoatomic model of the dynamin polymer identifies a hydrolysis-dependent powerstroke. 著者: Joshua S Chappie / Jason A Mears / Shunming Fang / Marilyn Leonard / Sandra L Schmid / Ronald A Milligan / Jenny E Hinshaw / Fred Dyda /  要旨: The GTPase dynamin catalyzes membrane fission by forming a collar around the necks of clathrin-coated pits, but the specific structural interactions and conformational changes that drive this process ...The GTPase dynamin catalyzes membrane fission by forming a collar around the necks of clathrin-coated pits, but the specific structural interactions and conformational changes that drive this process remain a mystery. We present the GMPPCP-bound structures of the truncated human dynamin 1 helical polymer at 12.2 Å and a fusion protein, GG, linking human dynamin 1's catalytic G domain to its GTPase effector domain (GED) at 2.2 Å. The structures reveal the position and connectivity of dynamin fragments in the assembled structure, showing that G domain dimers only form between tetramers in sequential rungs of the dynamin helix. Using chemical crosslinking, we demonstrate that dynamin tetramers are made of two dimers, in which the G domain of one molecule interacts in trans with the GED of another. Structural comparison of GG(GMPPCP) to the GG transition-state complex identifies a hydrolysis-dependent powerstroke that may play a role in membrane-remodeling events necessary for fission. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1949.map.gz emd_1949.map.gz | 7.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1949-v30.xml emd-1949-v30.xml emd-1949.xml emd-1949.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_1949.tif emd_1949.tif | 360.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1949 http://ftp.pdbj.org/pub/emdb/structures/EMD-1949 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1949 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1949 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1949.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1949.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Three-dimensional volume of deltaPRD human dynamin 1 polymer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.26 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : GMPPCP-stablized human dynamin 1 delta PRD polymer
| 全体 | 名称: GMPPCP-stablized human dynamin 1 delta PRD polymer |
|---|---|
| 要素 |
|
-超分子 #1000: GMPPCP-stablized human dynamin 1 delta PRD polymer
| 超分子 | 名称: GMPPCP-stablized human dynamin 1 delta PRD polymer / タイプ: sample / ID: 1000 / 集合状態: Helical assembly of dynamin tetramers / Number unique components: 3 |
|---|
-分子 #1: Human dynamin 1 delta PRD
| 分子 | 名称: Human dynamin 1 delta PRD / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Human dynamin 1 delta PRD / 詳細: contains bound GMPPCP / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane and cytosol Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane and cytosol |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: PMALC2XP5D Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: PMALC2XP5D |
-分子 #2: Human dynamin 1 pleckstrin homology domain
| 分子 | 名称: Human dynamin 1 pleckstrin homology domain / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Human dynamin 1 pleckstrin homology domain / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane and cytosol Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane and cytosol |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: PSKB-LNB Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: PSKB-LNB |
-分子 #3: Interferon-induced GTP-binding protein Mx1
| 分子 | 名称: Interferon-induced GTP-binding protein Mx1 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: Interferon-induced GTP-binding protein Mx1 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換プラスミド: PET11A Escherichia coli (大腸菌) / 組換プラスミド: PET11A |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Hepes pH 7.5, 50 mM NaCl, 2 mM EGTA, 4 mM MgCl2, 1 mM DTT, 1 mg/ml 0.4 um 1,2-dioleoyl-sn-glycero-3-phospho-L-serine (DOPS) liposomes, 2 mM GMPPCP |
| グリッド | 詳細: 400 mesh C-flat grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 93 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Manual 手法: Absorbed samples to grids, blotted, washed with 20 mM Hepes pH 7.5, blotted and plunged. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Gatan 626 side entry cryo-stage / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 最低: 93 K / 最高: 95 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 100,000X magnification |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 10 e/Å2 / 詳細: Images were collected on CCD |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each image using ACE2 |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 7.52 Å 想定した対称性 - らせんパラメータ - ΔΦ: 27.3 ° アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.2 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider 詳細: A total of 4,814 helical segments were incorporated by the IHRSR algorithm into the final reconstruction after 50 cycles |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: YUP |
| 詳細 | PDBEntryID_givenInChain. Protocol: Manual and Flexible fitting. Models were initially placed manually and initial positions were refined using the YUP.SCX method of the YUP software package |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3zys: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: YUP |
| 詳細 | PDBEntryID_givenInChain. Protocol: Manual and Flexible fitting. Models were initially placed manually and initial positions were refined using the YUP.SCX method of the YUP software package |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3zys: |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: YUP |
| 詳細 | PDBEntryID_givenInChain. Protocol: Manual and Flexible fitting. Models were initially placed manually and initial positions were refined using the YUP.SCX method of the YUP software package |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3zys: |
 ムービー
ムービー コントローラー
コントローラー