+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RPA tetrameric supercomplex from Pyrococcus abyssi | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報macromolecule metabolic process / intracellular organelle / primary metabolic process / : /  nucleic acid binding / nucleic acid binding /  DNA binding DNA binding類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus abyssi (古細菌) Pyrococcus abyssi (古細菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.35 Å クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Madru C / Martinez-Carranza M / Legrand P / Sauguet L | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: DNA-binding mechanism and evolution of replication protein A. 著者: Clément Madru / Markel Martínez-Carranza / Sébastien Laurent / Alessandra C Alberti / Maelenn Chevreuil / Bertrand Raynal / Ahmed Haouz / Rémy A Le Meur / Marc Delarue / Ghislaine Henneke ...著者: Clément Madru / Markel Martínez-Carranza / Sébastien Laurent / Alessandra C Alberti / Maelenn Chevreuil / Bertrand Raynal / Ahmed Haouz / Rémy A Le Meur / Marc Delarue / Ghislaine Henneke / Didier Flament / Mart Krupovic / Pierre Legrand / Ludovic Sauguet /  要旨: Replication Protein A (RPA) is a heterotrimeric single stranded DNA-binding protein with essential roles in DNA replication, recombination and repair. Little is known about the structure of RPA in ...Replication Protein A (RPA) is a heterotrimeric single stranded DNA-binding protein with essential roles in DNA replication, recombination and repair. Little is known about the structure of RPA in Archaea, the third domain of life. By using an integrative structural, biochemical and biophysical approach, we extensively characterize RPA from Pyrococcus abyssi in the presence and absence of DNA. The obtained X-ray and cryo-EM structures reveal that the trimerization core and interactions promoting RPA clustering on ssDNA are shared between archaea and eukaryotes. However, we also identified a helical domain named AROD (Acidic Rpa1 OB-binding Domain), and showed that, in Archaea, RPA forms an unanticipated tetrameric supercomplex in the absence of DNA. The four RPA molecules clustered within the tetramer could efficiently coat and protect stretches of ssDNA created by the advancing replisome. Finally, our results provide insights into the evolution of this primordial replication factor in eukaryotes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16444.map.gz emd_16444.map.gz | 203.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16444-v30.xml emd-16444-v30.xml emd-16444.xml emd-16444.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
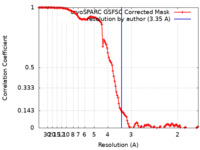
| FSC (解像度算出) |  emd_16444_fsc.xml emd_16444_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16444.png emd_16444.png | 83 KB | ||
| マスクデータ |  emd_16444_msk_1.map emd_16444_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16444_half_map_1.map.gz emd_16444_half_map_1.map.gz emd_16444_half_map_2.map.gz emd_16444_half_map_2.map.gz | 200 MB 200 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16444 http://ftp.pdbj.org/pub/emdb/structures/EMD-16444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16444 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c5yMC  8aa9C  8aajC  8aasC  8c5zC  8oejC  8oelC  15300 M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16444.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16444.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16444_msk_1.map emd_16444_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16444_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16444_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RPA tetrameric supercomplex
| 全体 | 名称: RPA tetrameric supercomplex |
|---|---|
| 要素 |
|
-超分子 #1: RPA tetrameric supercomplex
| 超分子 | 名称: RPA tetrameric supercomplex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi (古細菌) Pyrococcus abyssi (古細菌) |
| 分子量 | 理論値: 345 KDa |
-分子 #1: Replication factor A
| 分子 | 名称: Replication factor A / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi (古細菌) / 株: GE5 / ORSAY Pyrococcus abyssi (古細菌) / 株: GE5 / ORSAY |
| 分子量 | 理論値: 20.234057 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: VATYTRKKIK DIEAGDRFVE VRGTIAKVYR VLTYDACPEC KKKVDYDEGL GVWICPEHGE VQPIKMTILD FGLDDGTGYI RVTLFGDDA EELLGVSPEE IAEKIKELEE SGLTTKEAAR KLAEDEFYNI IGREIVVRGN VIEDRFLGLI LRASSWEDVD Y RREIERIK EELEKLGVM |
-分子 #2: RPA32 subunit of the hetero-oligomeric complex involved in homolo...
| 分子 | 名称: RPA32 subunit of the hetero-oligomeric complex involved in homologous recombination タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi (古細菌) / 株: GE5 / ORSAY Pyrococcus abyssi (古細菌) / 株: GE5 / ORSAY |
| 分子量 | 理論値: 20.930475 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: KKRMPATRLY IKDILEGYFV KSEGDFEPNY LITKYARKVY RAKIVGTVVR EPLIAEDETY GKFQVDDGTG VIWVLGFRDD TKFAKLVRK GDLVQVIGKI AEWRDDKQIL VEGVSKVHPN MWILHRYETL KEKIEHIKKA KIALEIYNQY GITAKSKVIA K NKGIEEEL LEVIDELYGI MM |
-分子 #3: RPA14 subunit of the hetero-oligomeric complex involved in homolo...
| 分子 | 名称: RPA14 subunit of the hetero-oligomeric complex involved in homologous recombination タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi (古細菌) / 株: GE5 / ORSAY Pyrococcus abyssi (古細菌) / 株: GE5 / ORSAY |
| 分子量 | 理論値: 13.078994 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: RRRKPAVERK ISEIREEDTR VSLIGRVIKV DKMDYMFWLD DGTGVAIIES ESDLPKVGQV VRVIGRIIRN EEGIHIYAEV IQDFSDADL EALEEIRELE RKLLPRLEGE IVW |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)