+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SARS-CoV-2 spike (Omicron BA.1 variant) in complex with nanobody W25 (map 4, one RBD up) | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  SARS-CoV-2 (SARSコロナウイルス2) / spike / SARS-CoV-2 (SARSコロナウイルス2) / spike /  Omicron (Ο) / Omicron (Ο) /  BA.1 (SARSコロナウイルス2-オミクロン株) / BA.1 (SARSコロナウイルス2-オミクロン株) /  nanobody (ナノボディ) / W25 / nanobody (ナノボディ) / W25 /  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.97 Å クライオ電子顕微鏡法 / 解像度: 4.97 Å | |||||||||
 データ登録者 データ登録者 | Modhiran N / Lauer S / Spahn CMT / Watterson D / Schwefel D | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of SARS-CoV-2 spike (Omicron BA.1 variant) in complex with nanobody W25 (map 4, one RBD up) 著者: Schwefel D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16028.map.gz emd_16028.map.gz | 48.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16028-v30.xml emd-16028-v30.xml emd-16028.xml emd-16028.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16028.png emd_16028.png | 74.2 KB | ||
| マスクデータ |  emd_16028_msk_1.map emd_16028_msk_1.map | 51.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16028.cif.gz emd-16028.cif.gz | 5.9 KB | ||
| その他 |  emd_16028_half_map_1.map.gz emd_16028_half_map_1.map.gz emd_16028_half_map_2.map.gz emd_16028_half_map_2.map.gz | 47.8 MB 47.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16028 http://ftp.pdbj.org/pub/emdb/structures/EMD-16028 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16028 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16028 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16028.map.gz / 形式: CCP4 / 大きさ: 51.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16028.map.gz / 形式: CCP4 / 大きさ: 51.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16028_msk_1.map emd_16028_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
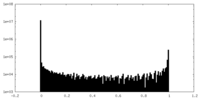
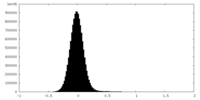
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_16028_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
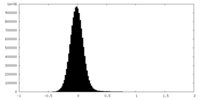
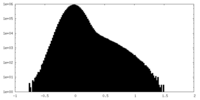
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_16028_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
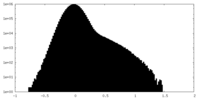
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike (Omicron BA.1 variant) in complex with nanobody W25
| 全体 | 名称: SARS-CoV-2 spike (Omicron BA.1 variant) in complex with nanobody W25 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike (Omicron BA.1 variant) in complex with nanobody W25
| 超分子 | 名称: SARS-CoV-2 spike (Omicron BA.1 variant) in complex with nanobody W25 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 100 KDa |
-分子 #1: SARS-CoV-2 spike protein (Omicron BA.1 variant)
| 分子 | 名称: SARS-CoV-2 spike protein (Omicron BA.1 variant) / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHVISG TNGTKRFDN PVLPFNDGVY FASIEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL D HKNNKSWM ESEFRVYSSA NNCTFEYVSQ ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHVISG TNGTKRFDN PVLPFNDGVY FASIEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL D HKNNKSWM ESEFRVYSSA NNCTFEYVSQ PFLMDLEGKQ GNFKNLREFV FKNIDGYFKI YSKHTPIIVR EP EDLPQGF SALEPLVDLP IGINITRFQT LLALHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN ENG TITDAV DCALDPLSET KCTLKSFTVE KGIYQTSNFR VQPTESIVRF PNITNLCPFD EVFNATRFAS VYAW NRKRI SNCVADYSVL YNLAPFFTFK CYGVSPTKLN DLCFTNVYAD SFVIRGDEVR QIAPGQTGNI ADYNY KLPD DFTGCVIAWN SNKLDSKVSG NYNYLYRLFR KSNLKPFERD ISTEIYQAGN KPCNGVAGFN CYFPLR SYS FRPTYGVGHQ PYRVVVLSFE LLHAPATVCG PKKSTNLVKN KCVNFNFNGL KGTGVLTESN KKFLPFQ QF GRDIADTTDA VRDPQTLEIL DITPCSFGGV SVITPGTNTS NQVAVLYQGV NCTEVPVAIH ADQLTPTW R VYSTGSNVFQ TRAGCLIGAE YVNNSYECDI PIGAGICASY QTQTKSHRRA RSVASQSIIA YTMSLGAEN SVAYSNNSIA IPTNFTISVT TEILPVSMTK TSVDCTMYIC GDSTECSNLL LQYGSFCTQL KRALTGIAVE QDKNTQEVF AQVKQIYKTP PIKYFGGFNF SQILPDPSKP SKRSFIEDLL FNKVTLADAG FIKQYGDCLG D IAARDLIC AQKFKGLTVL PPLLTDEMIA QYTSALLAGT ITSGWTFGAG AALQIPFAMQ MAYRFNGIGV TQ NVLYENQ KLIANQFNSA IGKIQDSLSS TASALGKLQD VVNHNAQALN TLVKQLSSKF GAISSVLNDI FSR LDKVEA EVQIDRLITG RLQSLQTYVT QQLIRAAEIR ASANLAATKM SECVLGQSKR VDFCGKGYHL MSFP QSAPH GVVFLHVTYV PAQEKNFTTA PAICHDGKAH FPREGVFVSN GTHWFVTQRN FYEPQIITTD NTFVS GNCD VVIGIVNNTV YDPLQPELDS FKEELDKYFK NHTSPDVDLG DISGINASVV NIQKEIDRLN EVAKNL NES LIDLQELGKY EQYIKWPWYI WLGFIAGLIA IVMVTIMLCC MTSCCSCLKG CCSCGSCCKF DEDDSEP VL KGVKLHYT GENBANK:  GENBANK: UFO69279.1 GENBANK: UFO69279.1 |
-分子 #2: Nanobody W25
| 分子 | 名称: Nanobody W25 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MKYLLPTAAA GLLLLAAQP A MAQVQLVE SG GGLVQPG ESL RLSCAA SGSI FGIYA VHWFR MAPG KEREFT AGF GSHGSTN YA ASVKGRFT M SRDNAKNTT YLQMNSLKPA DTAVYYCHA L IKNELGFL DY WGPGTQV TVS SAAAHH HHHH GAAEQ KLISE EDLN GAA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 9125 / 平均露光時間: 3.3 sec. / 平均電子線量: 40.0 e/Å2 |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 2458527 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 3次元分類 | クラス数: 3 / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.97 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) / 使用した粒子像数: 32512 |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)