+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Direct observation of the open conformational state of formin mDia1 at actin filament barbed end | |||||||||
 マップデータ マップデータ | Actin barbed-end - Formin "Open State" | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Actin binding protein /  Complex / barbed-end / Complex / barbed-end /  formin / formin /  MOTOR PROTEIN (モータータンパク質) MOTOR PROTEIN (モータータンパク質) | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) / Mus musculus (ハツカネズミ) /   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) | |||||||||
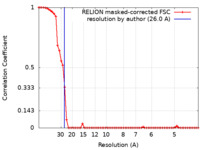
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 26.0 Å ネガティブ染色法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Maufront J / Guichard B / Cao L / Di Cicco A / Jegou A / Romet-Lemonne G / Bertin A | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Biol Cell / 年: 2023 ジャーナル: Mol Biol Cell / 年: 2023タイトル: Direct observation of the conformational states of formin mDia1 at actin filament barbed ends and along the filament. 著者: Julien Maufront / Bérengère Guichard / Lu-Yan Cao / Aurélie Di Cicco / Antoine Jégou / Guillaume Romet-Lemonne / Aurélie Bertin /  要旨: The fine regulation of actin polymerization is essential to control cell motility and architecture and to perform essential cellular functions. Formins are key regulators of actin filament assembly, ...The fine regulation of actin polymerization is essential to control cell motility and architecture and to perform essential cellular functions. Formins are key regulators of actin filament assembly, known to processively elongate filament barbed ends and increase their polymerization rate. Different models have been extrapolated to describe the molecular mechanism governing the processive motion of formin FH2 domains at polymerizing barbed ends. Using negative stain electron microscopy, we directly identified for the first time two conformations of the mDia1 formin FH2 domains in interaction with the barbed ends of actin filaments. These conformations agree with the speculated open and closed conformations of the "stair-stepping" model. We observed the FH2 dimers to be in the open conformation for 79% of the data, interacting with the two terminal actin subunits of the barbed end while they interact with three actin subunits in the closed conformation. In addition, we identified and characterized the structure of single FH2 dimers encircling the core of actin filaments, and reveal their ability to spontaneously depart from barbed ends. #1:  ジャーナル: BiorXiv / 年: 2022 ジャーナル: BiorXiv / 年: 2022タイトル: Direct observation of the open conformational state of formin mDia1 at actin filament barbed end 著者: Maufront J / Bertin A | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15947.map.gz emd_15947.map.gz | 1.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15947-v30.xml emd-15947-v30.xml emd-15947.xml emd-15947.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15947_fsc.xml emd_15947_fsc.xml | 6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15947.png emd_15947.png | 39.3 KB | ||
| マスクデータ |  emd_15947_msk_1.map emd_15947_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15947.cif.gz emd-15947.cif.gz | 4.4 KB | ||
| その他 |  emd_15947_half_map_1.map.gz emd_15947_half_map_1.map.gz emd_15947_half_map_2.map.gz emd_15947_half_map_2.map.gz | 17 MB 17 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15947 http://ftp.pdbj.org/pub/emdb/structures/EMD-15947 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15947 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15947 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15947.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15947.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Actin barbed-end - Formin "Open State" | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.13 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15947_msk_1.map emd_15947_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
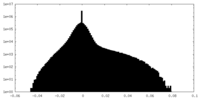
| 密度ヒストグラム |
-ハーフマップ: Actin barbed-end - Formin "Open State"
| ファイル | emd_15947_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Actin barbed-end - Formin "Open State" | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Actin barbed-end - Formin "Open State"
| ファイル | emd_15947_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Actin barbed-end - Formin "Open State" | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Actin barbed-end bound by formin mDia1 dimer in his "open state"
| 全体 | 名称: Actin barbed-end bound by formin mDia1 dimer in his "open state" |
|---|---|
| 要素 |
|
-超分子 #1: Actin barbed-end bound by formin mDia1 dimer in his "open state"
| 超分子 | 名称: Actin barbed-end bound by formin mDia1 dimer in his "open state" タイプ: complex / ID: 1 / 親要素: 0 |
|---|
-超分子 #2: Formin mDia1 dimer
| 超分子 | 名称: Formin mDia1 dimer / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-超分子 #3: Alpha Actin subunits assembled in helicoidal F-actin filament
| 超分子 | 名称: Alpha Actin subunits assembled in helicoidal F-actin filament タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製 #1
試料調製 #1
| Preparation ID | 1 |
|---|---|
| 濃度 | 0.04 mg/mL |
| 緩衝液 | pH: 7.8 |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate |
- 試料調製 #2
試料調製 #2
| Preparation ID | 2 |
|---|---|
| 濃度 | 0.01 mg/mL |
| 緩衝液 | pH: 7.8 |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 平均電子線量: 20.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X