+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Outer membrane secretin pore of the type 3 secretion system of Shigella flexneri | |||||||||
 マップデータ マップデータ | Main map sharpened and masked | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 type III protein secretion system complex / type III protein secretion system complex /  protein secretion by the type III secretion system / cell outer membrane protein secretion by the type III secretion system / cell outer membrane類似検索 - 分子機能 | |||||||||
| 生物種 |   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.42 Å クライオ電子顕微鏡法 / 解像度: 3.42 Å | |||||||||
 データ登録者 データ登録者 | Lunelli M | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Protein Sci / 年: 2023 ジャーナル: Protein Sci / 年: 2023タイトル: Integrative structural analysis of the type III secretion system needle complex from Shigella flexneri. 著者: Lara Flacht / Michele Lunelli / Karol Kaszuba / Zhuo Angel Chen / Francis J O' Reilly / Juri Rappsilber / Jan Kosinski / Michael Kolbe /   要旨: The type III secretion system (T3SS) is a large, transmembrane protein machinery used by various pathogenic gram-negative bacteria to transport virulence factors into the host cell during infection. ...The type III secretion system (T3SS) is a large, transmembrane protein machinery used by various pathogenic gram-negative bacteria to transport virulence factors into the host cell during infection. Understanding the structure of T3SSs is crucial for future developments of therapeutics that could target this system. However, much of the knowledge about the structure of T3SS is available only for Salmonella, and it is unclear how this large assembly is conserved across species. Here, we combined cryo-electron microscopy, cross-linking mass spectrometry, and integrative modeling to determine the structure of the T3SS needle complex from Shigella flexneri. We show that the Shigella T3SS exhibits unique features distinguishing it from other structurally characterized T3SSs. The secretin pore complex adopts a new fold of its C-terminal S domain and the pilotin MxiM[SctG] locates around the outer surface of the pore. The export apparatus structure exhibits a conserved pseudohelical arrangement but includes the N-terminal domain of the SpaS[SctU] subunit, which was not present in any of the previously published virulence-related T3SS structures. Similar to other T3SSs, however, the apparatus is anchored within the needle complex by a network of flexible linkers that either adjust conformation to connect to equivalent patches on the secretin oligomer or bind distinct surface patches at the same height of the export apparatus. The conserved and unique features delineated by our analysis highlight the necessity to analyze T3SS in a species-specific manner, in order to fully understand the underlying molecular mechanisms of these systems. The structure of the type III secretion system from Shigella flexneri delineates conserved and unique features, which could be used for the development of broad-range therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15701.map.gz emd_15701.map.gz | 24.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15701-v30.xml emd-15701-v30.xml emd-15701.xml emd-15701.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15701_fsc.xml emd_15701_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15701.png emd_15701.png | 155.3 KB | ||
| マスクデータ |  emd_15701_msk_1.map emd_15701_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15701_additional_1.map.gz emd_15701_additional_1.map.gz emd_15701_half_map_1.map.gz emd_15701_half_map_1.map.gz emd_15701_half_map_2.map.gz emd_15701_half_map_2.map.gz | 330.6 MB 335.2 MB 335.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15701 http://ftp.pdbj.org/pub/emdb/structures/EMD-15701 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15701 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15701 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8axlMC  8axkC  8axnC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15701.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15701.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map sharpened and masked | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38369 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15701_msk_1.map emd_15701_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
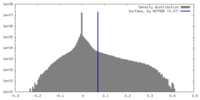
| 密度ヒストグラム |
-追加マップ: Main map unsharpened and unmasked
| ファイル | emd_15701_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map unsharpened and unmasked | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
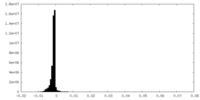
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15701_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
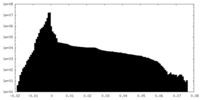
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15701_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
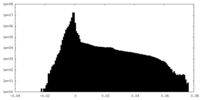
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Needle complex of the type 3 secretion system
| 全体 | 名称: Needle complex of the type 3 secretion system |
|---|---|
| 要素 |
|
-超分子 #1: Needle complex of the type 3 secretion system
| 超分子 | 名称: Needle complex of the type 3 secretion system / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
-超分子 #2: Outer membrane secretin pore of the type 3 secretion system
| 超分子 | 名称: Outer membrane secretin pore of the type 3 secretion system タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: all 詳細: Pore formed by the oligomerization of the domains N3, Secretin and S of MxiD |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
-分子 #1: Outer membrane protein MxiD
| 分子 | 名称: Outer membrane protein MxiD / タイプ: protein_or_peptide / ID: 1 / コピー数: 15 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 分子量 | 理論値: 63.230414 KDa |
| 配列 | 文字列: MKKFNIKSLT LLIVLLPLIV NANNIDSHLL EQNDIAKYVA QSDTVGSFFE RFSALLNYPI VVSKQAAKKR ISGEFDLSNP EEMLEKLTL LVGLIWYKDG NALYIYDSGE LISKVILLEN ISLNYLIQYL KDANLYDHRY PIRGNISDKT FYISGPPALV E LVANTATL ...文字列: MKKFNIKSLT LLIVLLPLIV NANNIDSHLL EQNDIAKYVA QSDTVGSFFE RFSALLNYPI VVSKQAAKKR ISGEFDLSNP EEMLEKLTL LVGLIWYKDG NALYIYDSGE LISKVILLEN ISLNYLIQYL KDANLYDHRY PIRGNISDKT FYISGPPALV E LVANTATL LDKQVSSIGT DKVNFGVIKL KNTFVSDRTY NMRGEDIVIP GVATVVERLL NNGKALSNRQ AQNDPMPPFN IT QKVSEDS NDFSFSSVTN SSILEDVSLI AYPETNSILV KGNDQQIQII RDIITQLDVA KRHIELSLWI IDIDKSELNN LGV NWQGTA SFGDSFGASF NMSSSASIST LDGNKFIASV MALNQKKKAN VVSRPVILTQ ENIPAIFDNN RTFYVSLVGE RNSS LEHVT YGTLINVIPR FSSRGQIEMS LTIEDGTGNS QSNYNYNNEN TSVLPEVGRT KISTIARVPQ GKSLLIGGYT HETNS NEII SIPFLSSIPV IGNVFKYKTS NISNIVRVFL IQPREIKESS YYNTAEYKSL ISEREIQKTT QIIPSETTLL EDEKSL VSY LNY |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 101179 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 101179 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 1-6 / 撮影したグリッド数: 1 / 実像数: 5238 / 平均露光時間: 1.5 sec. / 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 72.8 |
|---|---|
| 得られたモデル |  PDB-8axl: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)