+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of shCas12k-sgRNA-dsDNA ternary complex (type V-K CRISPR-associated transposon) | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  CRISPR (CRISPR) / CRISPR (CRISPR) /  complex / transposition / complex / transposition /  DNA BINDING PROTEIN (DNA結合タンパク質) DNA BINDING PROTEIN (DNA結合タンパク質) | |||||||||||||||
| 機能・相同性 | Cas12k 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) | |||||||||||||||
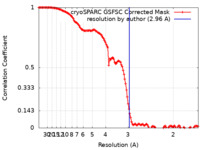
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.96 Å クライオ電子顕微鏡法 / 解像度: 2.96 Å | |||||||||||||||
 データ登録者 データ登録者 | Tenjo-Castano F / Sofos N / Stella S / Fuglsang A / Pape T / Mesa P / Stutzke LS / Temperini P / Montoya G | |||||||||||||||
| 資金援助 |  デンマーク, 4件 デンマーク, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Conformational Landscape of the Type V-K CRISPR-associated Transposon Integration Assembly 著者: Tenjo-Castano F / Sofos N / Stella S / Fuglsang A / Pape T / Mesa P / Stutzke LS / Temperini P / Montoya G | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15697.map.gz emd_15697.map.gz | 108.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15697-v30.xml emd-15697-v30.xml emd-15697.xml emd-15697.xml | 25.6 KB 25.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15697_fsc.xml emd_15697_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15697.png emd_15697.png | 53.7 KB | ||
| マスクデータ |  emd_15697_msk_1.map emd_15697_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15697.cif.gz emd-15697.cif.gz | 7 KB | ||
| その他 |  emd_15697_additional_1.map.gz emd_15697_additional_1.map.gz emd_15697_half_map_1.map.gz emd_15697_half_map_1.map.gz emd_15697_half_map_2.map.gz emd_15697_half_map_2.map.gz | 195.4 MB 200.7 MB 200.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15697 http://ftp.pdbj.org/pub/emdb/structures/EMD-15697 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15697 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15697 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8axaMC  19075  19282  19283  19284  19286 M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15697.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15697.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15697_msk_1.map emd_15697_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: DeepEMhancer map used for model building
| ファイル | emd_15697_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer map used for model building | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15697_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15697_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of ShCas12k bound to sgRNA and dsDNA.
| 全体 | 名称: Ternary complex of ShCas12k bound to sgRNA and dsDNA. |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of ShCas12k bound to sgRNA and dsDNA.
| 超分子 | 名称: Ternary complex of ShCas12k bound to sgRNA and dsDNA. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 分子量 | 理論値: 193 KDa |
-分子 #1: Cas12k
| 分子 | 名称: Cas12k / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 分子量 | 理論値: 74.738258 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSQITIQARL ISFESNRQQL WKLMADLNTP LINELLCQLG QHPDFEKWQQ KGKLPSTVVS QLCQPLKTDP RFAGQPSRLY MSAIHIVDY IYKSWLAIQK RLQQQLDGKT RWLEMLNSDA ELVELSGDTL EAIRVKAAEI LAIAMPASES DSASPKGKKG K KEKKPSSS ...文字列: MSQITIQARL ISFESNRQQL WKLMADLNTP LINELLCQLG QHPDFEKWQQ KGKLPSTVVS QLCQPLKTDP RFAGQPSRLY MSAIHIVDY IYKSWLAIQK RLQQQLDGKT RWLEMLNSDA ELVELSGDTL EAIRVKAAEI LAIAMPASES DSASPKGKKG K KEKKPSSS SPKRSLSKTL FDAYQETEDI KSRSAISYLL KNGCKLTDKE EDSEKFAKRR RQVEIQIQRL TEKLISRMPK GR DLTNAKW LETLLTATTT VAEDNAQAKR WQDILLTRSS SLPFPLVFET NEDMVWSKNQ KGRLCVHFNG LSDLIFEVYC GNR QLHWFQ RFLEDQQTKR KSKNQHSSGL FTLRNGHLVW LEGEGKGEPW NLHHLTLYCC VDNRLWTEEG TEIVRQEKAD EITK FITNM KKKSDLSDTQ QALIQRKQST LTRINNSFER PSQPLYQGQS HILVGVSLGL EKPATVAVVD AIANKVLAYR SIKQL LGDN YELLNRQRRQ QQYLSHERHK AQKNFSPNQF GASELGQHID RLLAKAIVAL ARTYKAGSIV LPKLGDMREV VQSEIQ AIA EQKFPGYIEG QQKYAKQYRV NVHRWSYGRL IQSIQSKAAQ TGIVIEEGKQ PIRGSPHDKA KELALSAYNL RLTRRSG SE FELENLYFQ UniProtKB: Cas12k |
-分子 #2: sgRNA
| 分子 | 名称: sgRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 分子量 | 理論値: 84.006516 KDa |
| 配列 | 文字列: GGAUAUUAAU AGCGCCGCAA UUCAUGCUGC UUGCAGCCUC UGAAUUUUGU UAAAUGAGGG UUAGUUUGAC UGUAUAAAUA CAGUCUUGC UUUCUGACCC UGGUAGCUGC UCACCCUGAU GCUGCUGUCA AUAGACAGGA UAGGUGCGCU CCCAGCAAUA A GGGCGCGG ...文字列: GGAUAUUAAU AGCGCCGCAA UUCAUGCUGC UUGCAGCCUC UGAAUUUUGU UAAAUGAGGG UUAGUUUGAC UGUAUAAAUA CAGUCUUGC UUUCUGACCC UGGUAGCUGC UCACCCUGAU GCUGCUGUCA AUAGACAGGA UAGGUGCGCU CCCAGCAAUA A GGGCGCGG AUGUACUGCU GUAGUGGCUA CUGAAUCACC CCCGAUCAAG GGGGAACCCU CCAAAAGGUG GGUUGAAAGG AG AAGUCAU UUAAUAAGGC CACU |
-分子 #3: DNA target strand
| 分子 | 名称: DNA target strand / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 分子量 | 理論値: 16.858855 KDa |
| 配列 | 文字列: (DC)(DT)(DA)(DG)(DA)(DA)(DA)(DG)(DA)(DC) (DT)(DT)(DT)(DT)(DA)(DA)(DC)(DA)(DG)(DT) (DG)(DG)(DC)(DC)(DT)(DT)(DA)(DT)(DT) (DA)(DA)(DA)(DT)(DG)(DA)(DC)(DT)(DT)(DC) (DT) (DC)(DA)(DA)(DC)(DC) ...文字列: (DC)(DT)(DA)(DG)(DA)(DA)(DA)(DG)(DA)(DC) (DT)(DT)(DT)(DT)(DA)(DA)(DC)(DA)(DG)(DT) (DG)(DG)(DC)(DC)(DT)(DT)(DA)(DT)(DT) (DA)(DA)(DA)(DT)(DG)(DA)(DC)(DT)(DT)(DC) (DT) (DC)(DA)(DA)(DC)(DC)(DA)(DT)(DC) (DT)(DT)(DG)(DC)(DT)(DG)(DA) |
-分子 #4: DNA non-target strand
| 分子 | 名称: DNA non-target strand / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 分子量 | 理論値: 17.027963 KDa |
| 配列 | 文字列: (DT)(DC)(DA)(DG)(DC)(DA)(DA)(DG)(DA)(DT) (DG)(DG)(DT)(DT)(DG)(DA)(DG)(DA)(DA)(DG) (DT)(DC)(DA)(DT)(DT)(DT)(DA)(DA)(DT) (DA)(DA)(DG)(DG)(DC)(DC)(DA)(DC)(DT)(DG) (DT) (DT)(DA)(DA)(DA)(DA) ...文字列: (DT)(DC)(DA)(DG)(DC)(DA)(DA)(DG)(DA)(DT) (DG)(DG)(DT)(DT)(DG)(DA)(DG)(DA)(DA)(DG) (DT)(DC)(DA)(DT)(DT)(DT)(DA)(DA)(DT) (DA)(DA)(DG)(DG)(DC)(DC)(DA)(DC)(DT)(DG) (DT) (DT)(DA)(DA)(DA)(DA)(DG)(DT)(DC) (DT)(DT)(DT)(DC)(DT)(DA)(DG) |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 3 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9.6 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 3 s blotting, 4 degrees celcius. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 96000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2231 / 平均露光時間: 45.0 sec. / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8axa: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)