+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle structure of Atg18-WT | |||||||||
 マップデータ マップデータ | sharp map from CryoSPARC softwarew | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  autophagy (オートファジー) / membrane remodeling / PIP binding / autophagy (オートファジー) / membrane remodeling / PIP binding /  PI3P / PI(3 / 5)P2 / lipid binding protein / PI3P / PI(3 / 5)P2 / lipid binding protein /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of phosphatidylinositol biosynthetic process / PAS complex / 1-phosphatidyl-1D-myo-inositol 3,5-bisphosphate metabolic process / phagophore /  vacuolar protein processing / positive regulation of vacuole organization / vacuolar protein processing / positive regulation of vacuole organization /  オートファジー / cytoplasm to vacuole targeting by the Cvt pathway / pexophagy / nucleophagy ...regulation of phosphatidylinositol biosynthetic process / PAS complex / 1-phosphatidyl-1D-myo-inositol 3,5-bisphosphate metabolic process / phagophore / オートファジー / cytoplasm to vacuole targeting by the Cvt pathway / pexophagy / nucleophagy ...regulation of phosphatidylinositol biosynthetic process / PAS complex / 1-phosphatidyl-1D-myo-inositol 3,5-bisphosphate metabolic process / phagophore /  vacuolar protein processing / positive regulation of vacuole organization / vacuolar protein processing / positive regulation of vacuole organization /  オートファジー / cytoplasm to vacuole targeting by the Cvt pathway / pexophagy / nucleophagy / protein localization to phagophore assembly site / piecemeal microautophagy of the nucleus / phagophore assembly site membrane / late endosome to vacuole transport / オートファジー / cytoplasm to vacuole targeting by the Cvt pathway / pexophagy / nucleophagy / protein localization to phagophore assembly site / piecemeal microautophagy of the nucleus / phagophore assembly site membrane / late endosome to vacuole transport /  phosphatidylinositol-3-phosphate binding / phagophore assembly site / fungal-type vacuole membrane / phosphatidylinositol-4-phosphate binding / phosphatidylinositol-3,5-bisphosphate binding / vacuolar membrane / phosphatidylinositol-3-phosphate binding / phagophore assembly site / fungal-type vacuole membrane / phosphatidylinositol-4-phosphate binding / phosphatidylinositol-3,5-bisphosphate binding / vacuolar membrane /  extrinsic component of membrane / extrinsic component of membrane /  autophagosome assembly / autophagosome assembly /  ubiquitin binding / cell periphery / ubiquitin binding / cell periphery /  オートファジー / オートファジー /  protein transport / endosome membrane / protein transport / endosome membrane /  エンドソーム / protein-containing complex / エンドソーム / protein-containing complex /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.8 Å クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Mann D / Fromm S / Martinez-Sanchez A / Gopaldass N / Mayer A / Sachse C | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structures of Atg18 oligomers reveal a tilted structural scaffold for Atg2 at the isolation membrane 著者: Mann D / Fromm SA / Martinez-Sanchez A / Gopaldass N / Mayer A / Sachse C | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15411.map.gz emd_15411.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15411-v30.xml emd-15411-v30.xml emd-15411.xml emd-15411.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15411_fsc.xml emd_15411_fsc.xml | 9.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15411.png emd_15411.png | 86.1 KB | ||
| マスクデータ |  emd_15411_msk_1.map emd_15411_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15411.cif.gz emd-15411.cif.gz | 5.6 KB | ||
| その他 |  emd_15411_half_map_1.map.gz emd_15411_half_map_1.map.gz emd_15411_half_map_2.map.gz emd_15411_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15411 http://ftp.pdbj.org/pub/emdb/structures/EMD-15411 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15411 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15411 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8afxMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15411.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15411.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharp map from CryoSPARC softwarew | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8389 Å | ||||||||||||||||||||||||||||||||||||
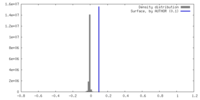
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15411_msk_1.map emd_15411_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
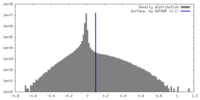
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_15411_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_15411_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Atg18
| 全体 | 名称: Atg18 |
|---|---|
| 要素 |
|
-超分子 #1: Atg18
| 超分子 | 名称: Atg18 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-分子 #1: Autophagy-related protein 18
| 分子 | 名称: Autophagy-related protein 18 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 54.62241 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SPTINFINFN QTGTCISLGT SKGFKIFNCE PFGKFYSEDS GGYAIVEMLF STSLLALVGI GDQPALSPRR LRIINTKKHS IICEVTFPT SILSVKMNKS RLVVLLQEQI YIYDINTMRL LHTIETNPNP RGLMAMSPSV ANSYLVYPSP PKVINSEIKA H ATTNNITL ...文字列: SPTINFINFN QTGTCISLGT SKGFKIFNCE PFGKFYSEDS GGYAIVEMLF STSLLALVGI GDQPALSPRR LRIINTKKHS IICEVTFPT SILSVKMNKS RLVVLLQEQI YIYDINTMRL LHTIETNPNP RGLMAMSPSV ANSYLVYPSP PKVINSEIKA H ATTNNITL SVGGNTETSF KRDQQDAGHS DISDLDQYSS FTKRDDADPT SSNGGNSSII KNGDVIVFNL ETLQPTMVIE AH KGEIAAM AISFDGTLMA TASDKGTIIR VFDIETGDKI YQFRRGTYAT RIYSISFSED SQYLAVTGSS KTVHIFKLGH SMS NNKLDS DDSNMEEAAA DDSSLDTTSI DALSDEENPT RLAREPYVDA SRKTMGRMIR YSSQKLSRRA ARTLGQIFPI KVTS LLESS RHFASLKLPV ETNSHVMTIS SIGSPIDIDT SEYPELFETG NSASTESYHE PVMKMVPIRV VSSDGYLYNF VMDPE RGGD CLILSQYSIL M UniProtKB: Autophagy-related protein 18 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / 実像数: 5117 / 平均電子線量: 70.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)