+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Sodium pumping NADH-quinone oxidoreductase with substrate Q2 | |||||||||
 マップデータ マップデータ | NQR with substrate Q2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  quinone (キノン) / quinone (キノン) /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 NADH:ユビキノン還元酵素 (ナトリウムイオン輸送型) / Gram-negative-bacterium-type cell wall / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor / sodium ion transport / respiratory electron transport chain / transmembrane transport / 2 iron, 2 sulfur cluster binding / FMN binding / NADH:ユビキノン還元酵素 (ナトリウムイオン輸送型) / Gram-negative-bacterium-type cell wall / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor / sodium ion transport / respiratory electron transport chain / transmembrane transport / 2 iron, 2 sulfur cluster binding / FMN binding /  electron transfer activity / electron transfer activity /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Vibrio cholerae (コレラ菌) Vibrio cholerae (コレラ菌) | |||||||||
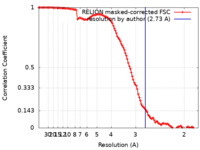
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.73 Å クライオ電子顕微鏡法 / 解像度: 2.73 Å | |||||||||
 データ登録者 データ登録者 | Hau J-L / Kaltwasser S / Vonck J / Fritz G / Steuber J | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Conformational coupling of redox-driven Na-translocation in Vibrio cholerae NADH:quinone oxidoreductase. 著者: Jann-Louis Hau / Susann Kaltwasser / Valentin Muras / Marco S Casutt / Georg Vohl / Björn Claußen / Wojtek Steffen / Alexander Leitner / Eckhard Bill / George E Cutsail / Serena DeBeer / ...著者: Jann-Louis Hau / Susann Kaltwasser / Valentin Muras / Marco S Casutt / Georg Vohl / Björn Claußen / Wojtek Steffen / Alexander Leitner / Eckhard Bill / George E Cutsail / Serena DeBeer / Janet Vonck / Julia Steuber / Günter Fritz /   要旨: In the respiratory chain, NADH oxidation is coupled to ion translocation across the membrane to build up an electrochemical gradient. In the human pathogen Vibrio cholerae, the sodium-pumping NADH: ...In the respiratory chain, NADH oxidation is coupled to ion translocation across the membrane to build up an electrochemical gradient. In the human pathogen Vibrio cholerae, the sodium-pumping NADH:quinone oxidoreductase (Na-NQR) generates a sodium gradient by a so far unknown mechanism. Here we show that ion pumping in Na-NQR is driven by large conformational changes coupling electron transfer to ion translocation. We have determined a series of cryo-EM and X-ray structures of the Na-NQR that represent snapshots of the catalytic cycle. The six subunits NqrA, B, C, D, E, and F of Na-NQR harbor a unique set of cofactors that shuttle the electrons from NADH twice across the membrane to quinone. The redox state of a unique intramembranous [2Fe-2S] cluster orchestrates the movements of subunit NqrC, which acts as an electron transfer switch. We propose that this switching movement controls the release of Na from a binding site localized in subunit NqrB. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15090.map.gz emd_15090.map.gz | 117.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15090-v30.xml emd-15090-v30.xml emd-15090.xml emd-15090.xml | 27.8 KB 27.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15090_fsc.xml emd_15090_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15090.png emd_15090.png | 218.3 KB | ||
| マスクデータ |  emd_15090_msk_1.map emd_15090_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15090.cif.gz emd-15090.cif.gz | 8.4 KB | ||
| その他 |  emd_15090_half_map_1.map.gz emd_15090_half_map_1.map.gz emd_15090_half_map_2.map.gz emd_15090_half_map_2.map.gz | 98.7 MB 98.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15090 http://ftp.pdbj.org/pub/emdb/structures/EMD-15090 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15090 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15090 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8a1vMC  8a1tC  8a1uC  8a1wC  8a1xC  8a1yC  8acwC  8acyC  8ad3C  8ad4C  8ad5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15090.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15090.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NQR with substrate Q2 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9125 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15090_msk_1.map emd_15090_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15090_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15090_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : NQR complex with substrate Q2
+超分子 #1: NQR complex with substrate Q2
+分子 #1: Na(+)-translocating NADH-quinone reductase subunit A
+分子 #2: Na(+)-translocating NADH-quinone reductase subunit B
+分子 #3: Na(+)-translocating NADH-quinone reductase subunit C
+分子 #4: Na(+)-translocating NADH-quinone reductase subunit D
+分子 #5: Na(+)-translocating NADH-quinone reductase subunit E
+分子 #6: Na(+)-translocating NADH-quinone reductase subunit F
+分子 #7: RIBOFLAVIN
+分子 #8: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #9: DODECYL-BETA-D-MALTOSIDE
+分子 #10: UBIQUINONE-2
+分子 #11: FLAVIN MONONUCLEOTIDE
+分子 #12: SODIUM ION
+分子 #13: FE2/S2 (INORGANIC) CLUSTER
+分子 #14: FLAVIN-ADENINE DINUCLEOTIDE
+分子 #15: water
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 |
| グリッド | モデル: C-flat-1.2/1.3 / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 276 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 191781 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 7 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 7647 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X