[日本語] English
 万見
万見- EMDB-14874: Cryo-EM Structure of Human Transferrin Receptor 1 bound to DNA Aptamer -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of Human Transferrin Receptor 1 bound to DNA Aptamer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 transferrin receptor activity / negative regulation of mitochondrial fusion / transferrin transport / Transferrin endocytosis and recycling / positive regulation of isotype switching / response to iron ion / response to copper ion / response to manganese ion / RND1 GTPase cycle / RND2 GTPase cycle ... transferrin receptor activity / negative regulation of mitochondrial fusion / transferrin transport / Transferrin endocytosis and recycling / positive regulation of isotype switching / response to iron ion / response to copper ion / response to manganese ion / RND1 GTPase cycle / RND2 GTPase cycle ... transferrin receptor activity / negative regulation of mitochondrial fusion / transferrin transport / Transferrin endocytosis and recycling / positive regulation of isotype switching / response to iron ion / response to copper ion / response to manganese ion / RND1 GTPase cycle / RND2 GTPase cycle / RHOB GTPase cycle / Golgi Associated Vesicle Biogenesis / RHOJ GTPase cycle / RHOC GTPase cycle / RHOQ GTPase cycle / RHOH GTPase cycle / CDC42 GTPase cycle / transport across blood-brain barrier / RHOG GTPase cycle / RHOA GTPase cycle / RAC2 GTPase cycle / RAC3 GTPase cycle / positive regulation of bone resorption / response to retinoic acid / positive regulation of B cell proliferation / transferrin receptor activity / negative regulation of mitochondrial fusion / transferrin transport / Transferrin endocytosis and recycling / positive regulation of isotype switching / response to iron ion / response to copper ion / response to manganese ion / RND1 GTPase cycle / RND2 GTPase cycle / RHOB GTPase cycle / Golgi Associated Vesicle Biogenesis / RHOJ GTPase cycle / RHOC GTPase cycle / RHOQ GTPase cycle / RHOH GTPase cycle / CDC42 GTPase cycle / transport across blood-brain barrier / RHOG GTPase cycle / RHOA GTPase cycle / RAC2 GTPase cycle / RAC3 GTPase cycle / positive regulation of bone resorption / response to retinoic acid / positive regulation of B cell proliferation /  クラスリン / クラスリン /  Hsp70 protein binding / positive regulation of T cell proliferation / RAC1 GTPase cycle / cellular response to leukemia inhibitory factor / osteoclast differentiation / response to nutrient / acute-phase response / positive regulation of protein-containing complex assembly / clathrin-coated endocytic vesicle membrane / HFE-transferrin receptor complex / Hsp70 protein binding / positive regulation of T cell proliferation / RAC1 GTPase cycle / cellular response to leukemia inhibitory factor / osteoclast differentiation / response to nutrient / acute-phase response / positive regulation of protein-containing complex assembly / clathrin-coated endocytic vesicle membrane / HFE-transferrin receptor complex /  receptor internalization / recycling endosome / positive regulation of protein localization to nucleus / recycling endosome membrane / receptor internalization / recycling endosome / positive regulation of protein localization to nucleus / recycling endosome membrane /  extracellular vesicle / extracellular vesicle /  メラノソーム / cellular response to xenobiotic stimulus / Cargo recognition for clathrin-mediated endocytosis / メラノソーム / cellular response to xenobiotic stimulus / Cargo recognition for clathrin-mediated endocytosis /  double-stranded RNA binding / virus receptor activity / positive regulation of peptidyl-serine phosphorylation / double-stranded RNA binding / virus receptor activity / positive regulation of peptidyl-serine phosphorylation /  Clathrin-mediated endocytosis / positive regulation of NF-kappaB transcription factor activity / iron ion transport / cytoplasmic vesicle / basolateral plasma membrane / positive regulation of canonical NF-kappaB signal transduction / blood microparticle / intracellular iron ion homeostasis / response to hypoxia / Clathrin-mediated endocytosis / positive regulation of NF-kappaB transcription factor activity / iron ion transport / cytoplasmic vesicle / basolateral plasma membrane / positive regulation of canonical NF-kappaB signal transduction / blood microparticle / intracellular iron ion homeostasis / response to hypoxia /  エンドソーム / endosome membrane / エンドソーム / endosome membrane /  エンドソーム / intracellular signal transduction / positive regulation of protein phosphorylation / external side of plasma membrane / intracellular membrane-bounded organelle / protein-containing complex binding / positive regulation of gene expression / negative regulation of apoptotic process / エンドソーム / intracellular signal transduction / positive regulation of protein phosphorylation / external side of plasma membrane / intracellular membrane-bounded organelle / protein-containing complex binding / positive regulation of gene expression / negative regulation of apoptotic process /  protein kinase binding / perinuclear region of cytoplasm / protein kinase binding / perinuclear region of cytoplasm /  細胞膜 / protein homodimerization activity / 細胞膜 / protein homodimerization activity /  extracellular space / extracellular space /  RNA binding / extracellular exosome / extracellular region / RNA binding / extracellular exosome / extracellular region /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / unidentified (未定義) Homo sapiens (ヒト) / unidentified (未定義) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.54 Å クライオ電子顕微鏡法 / 解像度: 2.54 Å | |||||||||
 データ登録者 データ登録者 | Wang T / Bansia H / Gutierrez D / des Georges A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2022 ジャーナル: J Am Chem Soc / 年: 2022タイトル: Discovery of a Transferrin Receptor 1-Binding Aptamer and Its Application in Cancer Cell Depletion for Adoptive T-Cell Therapy Manufacturing. 著者: Emmeline L Cheng / Ian I Cardle / Nataly Kacherovsky / Harsh Bansia / Tong Wang / Yunshi Zhou / Jai Raman / Albert Yen / Dominique Gutierrez / Stephen J Salipante / Amédée des Georges / ...著者: Emmeline L Cheng / Ian I Cardle / Nataly Kacherovsky / Harsh Bansia / Tong Wang / Yunshi Zhou / Jai Raman / Albert Yen / Dominique Gutierrez / Stephen J Salipante / Amédée des Georges / Michael C Jensen / Suzie H Pun /  要旨: The clinical manufacturing of chimeric antigen receptor (CAR) T cells includes cell selection, activation, gene transduction, and expansion. While the method of T-cell selection varies across ...The clinical manufacturing of chimeric antigen receptor (CAR) T cells includes cell selection, activation, gene transduction, and expansion. While the method of T-cell selection varies across companies, current methods do not actively eliminate the cancer cells in the patient's apheresis product from the healthy immune cells. Alarmingly, it has been found that transduction of a single leukemic B cell with the CAR gene can confer resistance to CAR T-cell therapy and lead to treatment failure. In this study, we report the identification of a novel high-affinity DNA aptamer, termed tJBA8.1, that binds transferrin receptor 1 (TfR1), a receptor broadly upregulated by cancer cells. Using competition assays, high resolution cryo-EM, and model building of the aptamer into the resulting electron density, we reveal that tJBA8.1 shares a binding site on TfR1 with holo-transferrin, the natural ligand of TfR1. We use tJBA8.1 to effectively deplete B lymphoma cells spiked into peripheral blood mononuclear cells with minimal impact on the healthy immune cell composition. Lastly, we present opportunities for affinity improvement of tJBA8.1. As TfR1 expression is broadly upregulated in many cancers, including difficult-to-treat T-cell leukemias and lymphomas, our work provides a facile, universal, and inexpensive approach for comprehensively removing cancerous cells from patient apheresis products for safe manufacturing of adoptive T-cell therapies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14874.map.gz emd_14874.map.gz | 32 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14874-v30.xml emd-14874-v30.xml emd-14874.xml emd-14874.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14874_fsc.xml emd_14874_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14874.png emd_14874.png | 101.4 KB | ||
| その他 |  emd_14874_half_map_1.map.gz emd_14874_half_map_1.map.gz emd_14874_half_map_2.map.gz emd_14874_half_map_2.map.gz | 59.2 MB 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14874 http://ftp.pdbj.org/pub/emdb/structures/EMD-14874 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14874 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14874 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zqsMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14874.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14874.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8465 Å | ||||||||||||||||||||||||||||||||||||
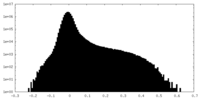
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_14874_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
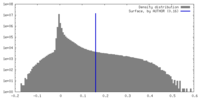
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14874_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
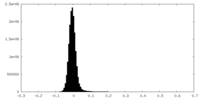
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human transferrin receptor 1 in complex with DNA aptamer
| 全体 | 名称: Human transferrin receptor 1 in complex with DNA aptamer |
|---|---|
| 要素 |
|
-超分子 #1: Human transferrin receptor 1 in complex with DNA aptamer
| 超分子 | 名称: Human transferrin receptor 1 in complex with DNA aptamer タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
| 分子量 | 理論値: 190 KDa |
-分子 #1: DNA (30-MER)
| 分子 | 名称: DNA (30-MER) / タイプ: dna / ID: 1 / 詳細: DNA aptamer / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 15.988172 KDa |
| 配列 | 文字列: (DG)(DC)(DA)(DG)(DC)(DA)(DG)(DC)(DG)(DT) (DA)(DA)(DA)(DG)(DG)(DG)(DG)(DG)(DT)(DG) (DT)(DT)(DT)(DG)(DT)(DG)(DC)(DG)(DG) (DT)(DG)(DT)(DG)(DG)(DA)(DG)(DT)(DG)(DC) (DG) (DC)(DG)(DT)(DG)(DC) ...文字列: (DG)(DC)(DA)(DG)(DC)(DA)(DG)(DC)(DG)(DT) (DA)(DA)(DA)(DG)(DG)(DG)(DG)(DG)(DT)(DG) (DT)(DT)(DT)(DG)(DT)(DG)(DC)(DG)(DG) (DT)(DG)(DT)(DG)(DG)(DA)(DG)(DT)(DG)(DC) (DG) (DC)(DG)(DT)(DG)(DC)(DT)(DG)(DC) (DT)(DG)(DC) |
-分子 #2: Transferrin receptor protein 1
| 分子 | 名称: Transferrin receptor protein 1 / タイプ: protein_or_peptide / ID: 2 / 詳細: Human Transferrin Receptor 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 84.967078 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MMDQARSAFS NLFGGEPLSY TRFSLARQVD GDNSHVEMKL AVDEEENADN NTKANVTKPK RCSGSICYGT IAVIVFFLIG FMIGYLGYC KGVEPKTECE RLAGTESPVR EEPGEDFPAA RRLYWDDLKR KLSEKLDSTD FTGTIKLLNE NSYVPREAGS Q KDENLALY ...文字列: MMDQARSAFS NLFGGEPLSY TRFSLARQVD GDNSHVEMKL AVDEEENADN NTKANVTKPK RCSGSICYGT IAVIVFFLIG FMIGYLGYC KGVEPKTECE RLAGTESPVR EEPGEDFPAA RRLYWDDLKR KLSEKLDSTD FTGTIKLLNE NSYVPREAGS Q KDENLALY VENQFREFKL SKVWRDQHFV KIQVKDSAQN SVIIVDKNGR LVYLVENPGG YVAYSKAATV TGKLVHANFG TK KDFEDLY TPVNGSIVIV RAGKITFAEK VANAESLNAI GVLIYMDQTK FPIVNAELSF FGHAHLGTGD PYTPGFPSFN HTQ FPPSRS SGLPNIPVQT ISRAAAEKLF GNMEGDCPSD WKTDSTCRMV TSESKNVKLT VSNVLKEIKI LNIFGVIKGF VEPD HYVVV GAQRDAWGPG AAKSGVGTAL LLKLAQMFSD MVLKDGFQPS RSIIFASWSA GDFGSVGATE WLEGYLSSLH LKAFT YINL DKAVLGTSNF KVSASPLLYT LIEKTMQNVK HPVTGQFLYQ DSNWASKVEK LTLDNAAFPF LAYSGIPAVS FCFCED TDY PYLGTTMDTY KELIERIPEL NKVARAAAEV AGQFVIKLTH DVELNLDYER YNSQLLSFVR DLNQYRADIK EMGLSLQ WL YSARGDFFRA TSRLTTDFGN AEKTDRFVMK KLNDRVMRVE YHFLSPYVSP KESPFRHVFW GSGSHTLPAL LENLKLRK Q NNGAFNETLF RNQLALATWT IQGAANALSG DVWDIDNEF |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: LACEY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: CARBON / 支持フィルム - #1 - トポロジー: CONTINUOUS 支持フィルム - #1 - Film thickness: 0.30000000000000004 nm 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 37000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 37000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 1397 / 平均露光時間: 4.0 sec. / 平均電子線量: 79.2 e/Å2 |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Ab-initio |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.54 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 216817 |
FSC曲線 (解像度の算出) | 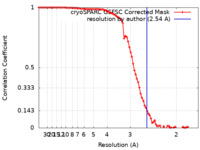 |
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7zqs: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)