+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | native KtrAB complex | |||||||||
 マップデータ マップデータ | KtrAB native complex of KtrB dimer with KtrA octamer ring | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | potassium transport /  membrane protein (膜タンパク質) / membrane protein (膜タンパク質) /  transporter (運搬体タンパク質) transporter (運搬体タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報potassium:chloride symporter activity / monoatomic cation transmembrane transporter activity / potassium ion transport /  ATP binding / ATP binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア) | |||||||||
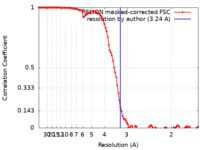
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.24 Å クライオ電子顕微鏡法 / 解像度: 3.24 Å | |||||||||
 データ登録者 データ登録者 | Vonck J / Stautz J | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: native KtrAB complex 著者: Stautz J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14859.map.gz emd_14859.map.gz | 92.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14859-v30.xml emd-14859-v30.xml emd-14859.xml emd-14859.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14859_fsc.xml emd_14859_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14859.png emd_14859.png | 228.2 KB | ||
| マスクデータ |  emd_14859_msk_1.map emd_14859_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14859.cif.gz emd-14859.cif.gz | 6.1 KB | ||
| その他 |  emd_14859_half_map_1.map.gz emd_14859_half_map_1.map.gz emd_14859_half_map_2.map.gz emd_14859_half_map_2.map.gz | 79.6 MB 79.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14859 http://ftp.pdbj.org/pub/emdb/structures/EMD-14859 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14859 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14859 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zpoMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14859.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14859.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | KtrAB native complex of KtrB dimer with KtrA octamer ring | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8371 Å | ||||||||||||||||||||||||||||||||||||
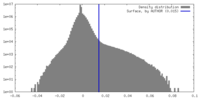
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14859_msk_1.map emd_14859_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
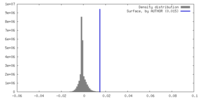
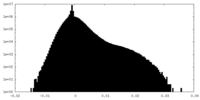
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14859_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
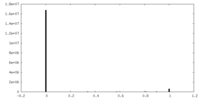
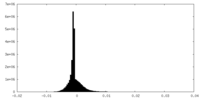
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14859_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
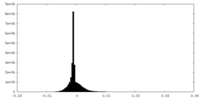
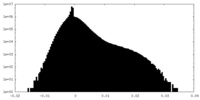
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : native KtrAB complex
| 全体 | 名称: native KtrAB complex |
|---|---|
| 要素 |
|
-超分子 #1: native KtrAB complex
| 超分子 | 名称: native KtrAB complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア) |
| 分子量 | 理論値: 390 KDa |
-分子 #1: Ktr system potassium uptake protein A
| 分子 | 名称: Ktr system potassium uptake protein A / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア) |
| 分子量 | 理論値: 23.83692 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MKTGDKQFAV IGLGRFGLAV CKELQDSGSQ VLAVDINEDR VKEAAGFVSQ AIVANCTHEE TVAELKLDDY DMVMIAIGAD VNASILATL IAKEAGVKSV WVKANDRFQA RVLQKIGADH IIMPERDMGI RVARKMLDKR VLEFHPLGSG LAMTEFVVGS R LMGKTLSD ...文字列: MKTGDKQFAV IGLGRFGLAV CKELQDSGSQ VLAVDINEDR VKEAAGFVSQ AIVANCTHEE TVAELKLDDY DMVMIAIGAD VNASILATL IAKEAGVKSV WVKANDRFQA RVLQKIGADH IIMPERDMGI RVARKMLDKR VLEFHPLGSG LAMTEFVVGS R LMGKTLSD LALCKVEGVQ VLGYKRGPEI IKAPDMSTTL EIGDLIIVVG PQDKLANKLK SL UniProtKB: Ktr system potassium uptake protein A |
-分子 #2: Ktr system potassium uptake protein B
| 分子 | 名称: Ktr system potassium uptake protein B / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア) |
| 分子量 | 理論値: 49.707715 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MTQFHQRGVF YVPDGKRDKA KGGEPRIILL SFLGVLLPSA VLLTLPVFSV SGLSITDALF TATSAISVTG LGVVDTGQHF TLAGKILLM CLMQIGGLGQ MTLSAVLLYM FGVRLSLRQQ ALAKEALGQE RQVNLRRLVK KIVTFALVAE AIGFVFLSYR W VPEMGWQT ...文字列: MTQFHQRGVF YVPDGKRDKA KGGEPRIILL SFLGVLLPSA VLLTLPVFSV SGLSITDALF TATSAISVTG LGVVDTGQHF TLAGKILLM CLMQIGGLGQ MTLSAVLLYM FGVRLSLRQQ ALAKEALGQE RQVNLRRLVK KIVTFALVAE AIGFVFLSYR W VPEMGWQT GMFYALFHSI SAFNNAGFAL FSDSMMSFVN DPLVSFTLAG LFIFGGLGFT VIGDVWRHWR KGFHFLHIHT KI MLIATPL LLLVGTVLFW LLERHNPNTM GSLTTGGQWL AAFFQSASAR TAGFNSVDLT QFTQPALLIM IVLMLIGAGS TST GGGIKV STFAVAFMAT WTFLRQKKHV VMFKRTVNWP TVTKSLAIIV VSGAILTTAM FLLMLTEKAS FDKVMFETIS AFAT VGLTA GLTAELSEPG KYIMIVVMII GRIGPLTLAY MLARPEPTLI KYPEDTVLTG UniProtKB: Ktr system potassium uptake protein B |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 8 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: DODECYL-BETA-D-MALTOSIDE
| 分子 | 名称: DODECYL-BETA-D-MALTOSIDE / タイプ: ligand / ID: 5 / コピー数: 4 / 式: LMT |
|---|---|
| 分子量 | 理論値: 510.615 Da |
| Chemical component information |  ChemComp-LMT: |
-分子 #6: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.2 µm / 倍率(補正後): 60168 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 実像数: 3663 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The model was refined by phenix.real-space-refine |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-7zpo: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)