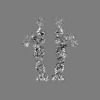+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | gp6/gp15/gp16 connector complex of bacteriophage SPP1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral DNA genome packaging, headful / symbiont genome ejection through host cell envelope, long flexible tail mechanism / viral portal complex / viral procapsid /  virion component virion component類似検索 - 分子機能 | |||||||||
| 生物種 |   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.7 Å クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Orlov I / Roche S / Tavares P / Orlova EV | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: CryoEM structure and assembly mechanism of a bacterial virus genome gatekeeper. 著者: Igor Orlov / Stéphane Roche / Sandrine Brasilès / Natalya Lukoyanova / Marie-Christine Vaney / Paulo Tavares / Elena V Orlova /   要旨: Numerous viruses package their dsDNA genome into preformed capsids through a portal gatekeeper that is subsequently closed. We report the structure of the DNA gatekeeper complex of bacteriophage SPP1 ...Numerous viruses package their dsDNA genome into preformed capsids through a portal gatekeeper that is subsequently closed. We report the structure of the DNA gatekeeper complex of bacteriophage SPP1 (gp6gp15gp16) in the post-DNA packaging state at 2.7 Å resolution obtained by single particle cryo-electron microscopy. Comparison of the native SPP1 complex with assembly-naïve structures of individual components uncovered the complex program of conformational changes leading to its assembly. After DNA packaging, gp15 binds via its C-terminus to the gp6 oligomer positioning gp15 subunits for oligomerization. Gp15 refolds its inner loops creating an intersubunit β-barrel that establishes different types of contacts with six gp16 subunits. Gp16 binding and oligomerization is accompanied by folding of helices that close the portal channel to keep the viral genome inside the capsid. This mechanism of assembly has broad functional and evolutionary implications for viruses of the prokaryotic tailed viruses-herpesviruses lineage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14509.map.gz emd_14509.map.gz | 351 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14509-v30.xml emd-14509-v30.xml emd-14509.xml emd-14509.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14509.png emd_14509.png | 170 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14509 http://ftp.pdbj.org/pub/emdb/structures/EMD-14509 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14509 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14509 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7z4wMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14509.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14509.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.69 Å | ||||||||||||||||||||||||||||||||||||
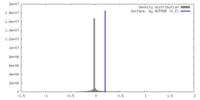
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : gp6(12) gp15(12) gp16(6) portal complex (connector) of SPP1 bacte...
| 全体 | 名称: gp6(12) gp15(12) gp16(6) portal complex (connector) of SPP1 bacteriophage |
|---|---|
| 要素 |
|
-超分子 #1: gp6(12) gp15(12) gp16(6) portal complex (connector) of SPP1 bacte...
| 超分子 | 名称: gp6(12) gp15(12) gp16(6) portal complex (connector) of SPP1 bacteriophage タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) / 株: YB886 Bacillus subtilis (枯草菌) / 株: YB886 |
| 分子量 | 理論値: 0.902 kDa/nm |
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
| 分子量 | 理論値: 57.390277 KDa |
| 配列 | 文字列: MADIYPLGKT HTEELNEIIV ESAKEIAEPD TTMIQKLIDE HNPEPLLKGV RYYMCENDIE KKRRTYYDAA GQQLVDDTKT NNRTSHAWH KLFVDQKTQY LVGEPVTFTS DNKTLLEYVN ELADDDFDDI LNETVKNMSN KGIEYWHPFV DEEGEFDYVI F PAEEMIVV ...文字列: MADIYPLGKT HTEELNEIIV ESAKEIAEPD TTMIQKLIDE HNPEPLLKGV RYYMCENDIE KKRRTYYDAA GQQLVDDTKT NNRTSHAWH KLFVDQKTQY LVGEPVTFTS DNKTLLEYVN ELADDDFDDI LNETVKNMSN KGIEYWHPFV DEEGEFDYVI F PAEEMIVV YKDNTRRDIL FALRYYSYKG IMGEETQKAE LYTDTHVYYY EKIDGVYQMD YSYGENNPRP HMTKGGQAIG WG RVPIIPF KNNEEMVSDL KFYKDLIDNY DSITSSTMDS FSDFQQIVYV LKNYDGENPK EFTANLRYHS VIKVSGDGGV DTL RAEIPV DSAAKELERI QDELYKSAQA VDNSPETIGG GATGPALENL YALLDLKANM AERKIRAGLR LFFWFFAEYL RNTG KGDFN PDKELTMTFT RTRIQNDSEI VQSLVQGVTG GIMSKETAVA RNPFVQDPEE ELARIEEEMN QYAEMQGNLL DDEGG DDDL EEDDPNAGAA ESGGAGQVS |
-分子 #2: Head completion protein gp15
| 分子 | 名称: Head completion protein gp15 / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
| 分子量 | 理論値: 11.629402 KDa |
| 配列 | 文字列: MDIQRVKRLL SITNDKHDEY LTEMVPLLVE FAKDECHNPF IDKDGNESIP SGVLIFVAKA AQFYMTNAGL TGRSMDTVSY NFATEIPST ILKKLNPYRK MAR |
-分子 #3: Head completion protein gp16
| 分子 | 名称: Head completion protein gp16 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
| 分子量 | 理論値: 12.55499 KDa |
| 配列 | 文字列: MYEEFPDVIT FQSYVEQSNG EGGKTYKWVD EFTAAAHVQP ISQEEYYKAQ QLQTPIGYNI YTPYDDRIDK KMRVIYRGKI VTFIGDPVD LSGLQEITRI KGKEDGAYVG |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 406 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.64 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 96 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 120000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 3876 / 平均露光時間: 1.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 520000 |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: 詳細: The map has been low pass filtered to 20 Angstrom. |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 2.0) |
| 最終 3次元分類 | クラス数: 5 / ソフトウェア - 名称: cryoSPARC (ver. 2) 詳細: After three rounds of 2D classification in CryoSPARC 401,807 particle images were selected for the next two rounds of refinement. |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 2.0) |
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2) ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2)詳細: Local resolution variations in the reconstruction was estimated using ResMap 使用した粒子像数: 401807 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Residue range: 254-344 |
|---|---|
| 詳細 | Manual inspection of the residues in the complete gp6, gp15 and gp16 polypeptide chains tracing was done using COOT and refined in Phenix |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 90 |
| 得られたモデル |  PDB-7z4w: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)